技术特征:
1.一种基于染色质随机折叠过程预测染色质域结构tad的方法,其特征在于,包括如下步骤:(1)为待研究的染色质区间构建携带染色质可及性信息的染色质高分子链模型集合;(2)模拟携带染色质可及性信息的染色质高分子链模型在微小球形空间中的随机折叠过程,计算得到染色质3d结构的集合;(3)基于层级优化法得到高精度的待研究的染色质区间的3d结构集合;(4)基于步骤(3)得到的染色质3d结构集合,构建平均接触矩阵以显示tad结构。2.如权利要求1所述的方法,其特征在于,所述步骤(1)包括:步骤(11):选取待研究的染色质区间,序列长度为l,并获取该区间对应的多细胞平均染色质可及性数据实验数据,其中,对于哺乳动物细胞,l的取值范围为:10mb<l<8000mb;步骤(12):将待研究染色质区间等分为序列长度为l的m个染色质片段,m=l/l,并根据步骤(11)得到的染色质可及性实验数据,为每个所述染色质片段确定一个归一化的可及性信号值λ,以对每个所述染色质片段的染色质可及性性质进行定量,其中,l的取值范围为:5kb<l<5000kb;步骤(13):将每个所述染色质片段对应的粗粒化链珠的数目视为独立的随机泊松变量,其取值服从泊松分布,概率为:其中,k
i
表示第i个染色质片段对应的链珠数目,λ
i
是第i个染色质片段的染色质可及性信号值,每个链珠具有相同的直径;步骤(14):由m个染色质片段构成的待研究的染色质区间的高分子链模型所包含的链珠的总数目为步骤(15):由于第i个染色质片段的链珠数目k
i
是一个随机泊松变量,可以为待研究的染色质区间随机产生多个高分子链串珠模型,形成一个集合;该集合中每个模型携带确定的染色质可及性信息,它们的平均值与所述步骤(11)中的多细胞平均染色质可及性实验数据相一致。3.如权利要求1所述的方法,其特征在于,所述步骤(1)中染色质可及性信息采用来自多细胞atac-seq或dnase-seq实验数据。4.如权利要求1所述的方法,其特征在于,所述步骤(1)中构建的染色质高分子链模型的精度为步骤(12)等分后染色质片段序列长度l kb。5.如权利要求1所述的方法,其特征在于,所述步骤(2)包括:步骤(21):从不同的随机结构出发,对每条高分子链的随机折叠过程进行多次分子动力学模拟;步骤(22):通过对不同高分子链和不同随机结构出发的模拟结果进行收集,得到一个待研究的染色质区间的3d结构集合。6.如权利要求1所述的方法,其特征在于,所述步骤(3)包括:步骤(31):构建低精度的高分子链模型,从随机结构出发进行优化;步骤(32):在低精度优化结构的基础上,采用插值法产生高精度的3d结构;步骤(33):
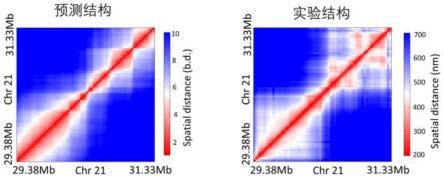
以所述步骤(32)产生的结构作为初始结构进行高精度的优化,得到指定的高精度结构。7.如权利要求1所述的方法,其特征在于,所述步骤(4)包括:步骤(41):每个3d结构的高分子模型都由m个等序列长度染色质片段组成;在任意一个3d结构中,测量3d结构中每一对染色质片段质心之间的距离,可以为3d结构构建单个距离矩阵;步骤(42):根据模型中珠链的直径设定一个距离阈值,两个片段之间的距离低于阈值时,认为该两个片段之间存在接触;据此,将步骤(41)中构建的单个距离矩阵转化为单个接触矩阵;步骤(43):将单个距离矩阵和单个接触矩阵在整个3d结构集合上进行平均,可得到平均距离矩阵和平均接触矩阵;步骤(44):定义分离分数,用以定量分析平均接触矩阵中每一个基因位点上下游染色质区域的分离程度;通过分离分数显示tads结构。
技术总结
本发明公开一种基于染色质随机折叠过程预测染色质域结构TAD的方法,包括如下步骤:(1)为待研究的染色质区间构建携带染色质可及性信息的染色质高分子链模型集合;(2)模拟携带染色质可及性信息的染色质高分子链模型在微小球形空间中的随机折叠过程,计算得到染色质3D结构的集合;(3)基于层级优化法得到高精度的待研究的染色质区间的3D结构集合;(4)基于步骤(3)得到的染色质3D结构集合,构建平均接触矩阵以显示TAD结构。本发明采用层级优化法对携带染色质可及性信息的高分子链模型的随机折叠过程进行分子动力学模拟,在10kb精度下预测了哺乳动物全基因组3D结构中40%以上的TAD结构。TAD结构。TAD结构。
技术研发人员:孟露明 罗琼 刘柏平
受保护的技术使用者:华南农业大学
技术研发日:2022.06.27
技术公布日:2022/9/13
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。