用于确定细胞培养过程中的过程变量的方法
1.本发明属于哺乳动物细胞培养领域。更具体地说,本发明的目的是一种用于基于一组过程变量的历史在线值和离线值在线确定过程目标参数的方法。
背景技术:
2.对于制药行业中治疗剂的生产,对质量和再现性提出了最高要求。为此,制定了经验标准(gmp指南、良好生产规范)来定义目标值、过程限制和偏差,以满足这些要求。美国食品和药物管理局(fda)最近通过pat(过程分析技术)计划要求制药行业更好地了解所执行的过程,以提高其产品质量[1]。近年来,新技术(诸如基于计算机的模型)已被用于促进对例如用于生产治疗性蛋白质的cho细胞的细胞培养过程的理解。
[0003]
生物反应器最常用于培养细胞。在培养期间,这里记录了各种过程变量。这些使得能够实现过程监测和控制,并用于维持受控的环境条件。在线值与离线值之间进行了区分。这两个值都提供了有关该过程的重要信息。在线值由用于直接在线控制的适当传感器收集。然而,离线值将通过手动采样和随后的外部分析方法来确定。这种离线参数是,例如,活细胞密度、葡萄糖和乳酸浓度。这些可用于评估当前培养条件,并在必要时干预过程的调节。
[0004]
样品分析需要更多的人工,尤其是在高通量培养系统的情况下。在一些情况下,这些外部方法也可能导致错误和设备故障。为了使过程更加高效和稳健,可以使用在培养期间已经记录的在线值来获得在线信息。通过这种方式,可以分析现有的测得参数及其关系,以便使用合适的机器学习数学模型进行描述。
[0005]
用于在分批补料过程中监测生物量的人工神经网络(ann)已有描述[8]。kroll等人描述了一种基于模型的软传感器,用于确定cho细胞生物量的亚群[9]。
[0006]
hutter,s.等人公开了中国仓鼠卵巢灌注细胞培养中免疫球蛋白g的糖基化通量分析(process 6(2018)176)。他们描述了一种基于代谢通量分析的方法,以深入了解糖基化途径。hutter等人专注于灌注细胞培养实验中的代谢通量分析。仅使用离线确定的参数来拟合机械(线性)模型,使用随机森林模型对输入参数对糖基化结果的影响进行排序。因此,hutter等人公开基于离线数据并在培养后进行的统计分析,即使历史数据具有(生物学)意义的建模工具。没有公开预测或在线算法。
[0007]
在white paper“biopharma pat
–
quality attributes,critical process parameters&key performance indicators at the bioreactor”(可查阅https://www.researchgate.net/publication/326804832_biopharma_pat_-_quality_attributes_criticcal_process_parameters_key_performance_indicators_at_the_bioreactor)中对过程分析技术进行了高级概述。该论文描述了培养原则(例如分批、补料分批和灌注、监测方法)。其中如溶解氧等测量的影响用于获得过程理解。没有公开任何输出参数的预测或任何机器学习方法。
[0008]
rubin,j.等人报告说,ph偏移会影响cho细胞培养性能和抗体n-连接糖基化(bioprocess.biosys.eng.,41(2018)1731-1741)。作者公开了一项关于细胞培养ph值对抗
体糖基化影响的研究,该研究使用在任何培养中进行过程参数的典型离线测量,以及ph值变化对其的影响。
[0009]
downey,b.j.等人报告了一种在早期工艺开发中使用介电光谱预测活细胞体积(vcv)的新方法(biotechnol.prog.30(2014)479-487)。
[0010]
xiao,p.等人报道了补料分批培养中cho细胞大小增加阶段的代谢特征(appl.microbiol.biotechnol.101(2017)8101-8113)。
[0011]
kroll,p.等人报道了一种用于监测哺乳动物细胞培养过程中生物量亚群的软传感器(biotechnol.lett.39(2017)1667-1673)。作者使用浊度物理传感器基于线性模型测量活细胞计数(vcc,相当于vcd)。
技术实现要素:
[0012]
本发明至少部分地基于以下发现:通过从历史数据集中选择某些特定过程变量,可以获得有用的数据驱动模型,其含有用于培养cho细胞的重要参数,诸如实时vcd(活细胞密度)、vcv(活细胞体积)、葡萄糖和乳酸。使用根据本发明的方法,可以在整个培养过程中为目标变量提供精确的类似在线值而无需采样。
[0013]
本发明的目的是一种在表达抗体的cho细胞的培养中和培养期间,通过该cho细胞的培养模型仅使用来自所述培养的在线测得值确定活细胞密度和/或活细胞体积和/或培养基中的葡萄糖浓度和/或培养基中的乳酸浓度的方法,其特征在于,该模型基于包含以下项的特征矩阵来产生:
‘
时间’、
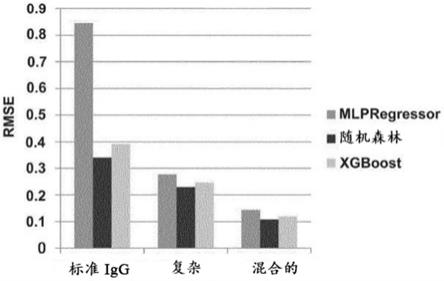
‘
cht.pv’、
‘
acot.pv’、
‘
fed2t.pv’、
‘
gew.pv’、
‘
co2t.pv’、
‘
aco.pv’、
‘
ao.pv’、
‘
n2.pv’、
‘
lge.pv’、
‘
co2.pv’、
‘
fed3t.pv’、
‘
our’及
‘
ph.pv’。
[0014]
特征表示参数如下:
[0015][0016]
在一个实施例中,该模型使用随机森林方法而产生。
[0017]
在一个实施例中,训练数据集包含至少10次培养运行,优选至少60次培养运行。
[0018]
在一个实施例中,该模型是用训练数据集获得的,该训练数据集包括表达复杂igg,即包含与野生型y形全长抗体不同形式的抗体的哺乳动物细胞的培养运行,例如通过包含额外的结构域,诸如一个或多个fab。在一个实施例中,训练数据集还含有表达标准igg,即没有额外或缺失结构域的y形野生型样抗体的哺乳动物细胞的培养运行。
[0019]
在一个实施例中,大约80%的可用于模型形成的数据集被用作训练数据集,而其余数据集被用作测试数据集。
[0020]
在一个实施例中
[0021]
a)以80:20的比率将可用于建模的数据集随机划分为训练数据集和测试数据集,
[0022]
b)形成模型,
[0023]
c)从训练数据集确定用于确定数据集的目标参数的均值和标准偏差,且从测试数据集确定用于确定记录的目标参数的均值和标准偏差,
[0024]
d)重复步骤a)至c),直到实现关于测试数据集与训练数据集之间的划分的可比较的平均值和标准偏差,即在彼此最多10%以内,优选最多5%以内。
[0025]
在一个实施例中,数据集中的缺失数据点由插值补充。
[0026]
在一个实施例中,数据集含有至少60分钟的数据点,优选大约每5至10分钟的数据点。
附图说明
[0027]
图1:以aco.pv为例的线性内插测得值。插值范围从第0.5天到第13.5天。
[0028]
图2:示例性培养的活细胞密度的内插测量曲线。插值和确定系数:peleg拟合(r2=0.957)、单变量样条(r2=0.998)和三阶多项式拟合(r2=0.864)。
[0029]
图3:来自项目2的ambr250运行的数据集的示例性相关性分析。不同插值策略的相关系数比较。该图显示vcd的单个在线参数的散点图。
[0030]
图4:根据目标变量vcd在整个数据集上的互信息计算的信息内容。
[0031]
图5:两次独立运行的随机森林vcd估计。在图的上部可以实现r2为0.20317的估计。在图的下部分,可以实现r2为0.54896的估计。
[0032]
图6:目标变量
‘
vcd’的模型mlpregressor(a)、随机森林(b)和xgboost(c)的新创建测试数据集的预测直方图。预测值的拟合vcd值的误差显示在x轴上。y轴表示误差的相对频率。
[0033]
图7:对于测试数据集的两个示例性运行,随机森林的vcd的估计。
[0034]
在图的上部分,实现了r2为0.98944的估计。在图的下部分可以实现r2为0.99837的估计。
[0035]
图8:根据目标变量葡萄糖的互信息计算整个数据集的信息内容。
[0036]
图9:根据测试数据集的两个示例性运行的随机森林来估计葡萄糖。
[0037]
在图的上部分,可以实现r2为0.99的估计值。在图的下部分,可以实现r2为0.97的估计值。
[0038]
图10:根据目标变量乳酸的互信息计算整个数据集的信息内容。
[0039]
图11:目标变量乳酸的mlpregressor(a)、随机森林(b)和xgboost(c)的测试数据集的预测直方图。添加到预测值的乳酸值的误差显示在x轴上。y轴表示误差的相对频率。
[0040]
图12:对于测试数据集的两个示例性运行,通过xgboost对乳酸进行估计。在图的上部分,可以实现r2为0.99的估计值。在图的下部分,可以实现r2为0.98的估计值。
[0041]
图13:利用不同数量的训练数据集计算mlpregressor、随机森林和xgboost的rmse。
[0042]
图14:对于单次培养,随机森林vcd的估计。vcd的peleg拟合以蓝色显示,vcd的估计值以橙色显示。
[0043]
图15:表示整个培养周期过程中每个采样的平均直径。项目1和3具有复杂分子格式(此处以蓝色显示,左侧)作为产品。项目2和4具有y形ig-g格式(此处以绿色显示,右侧)作为目标产品。方框图含有均值;单位显示为标准化。
[0044]
图16:图的左部分:随机森林对vcd的估计。红色部分是用于针对真实值测试数据集的估计值。蓝色部分是用于针对真实值训练数据集的估计值。测试数据集和训练数据集的理想估计值以黑色显示。图的右部分:随机森林对vcv的估计。红色部分是用于针对真实值测试数据集的估计值。蓝色部分是用于针对真实值训练数据集的估计值。测试数据集和
训练数据集的理想估计值以黑色显示。
[0045]
图17:表示每个项目在整个培养周期过程中每个样品的平均直径。项目1=紫色,项目2=红色,项目3=绿色,且项目4=蓝色。方框图含有均值。
[0046]
图18:使用随机森林模型(最佳模型)比较vcd/vcv。
[0047]
图19:考虑所有模型(mlpregressor、随机森林、xgboost)和训练数据集,rmse的行为取决于目标参数vcv。
[0048]
图20:测试数据集和训练数据集的rmse的差异条形图,目标变量vcv的最佳模型。
[0049]
本发明的具体实施例
[0050]
1.一种在哺乳动物细胞培养期间确定一个或多个过程变量的方法,其特征在于,
[0051]
过程变量是单独确定的
[0052]
i)通过哺乳动物细胞培养的数据驱动模型,该模型已使用包含过程变量
‘
时间’、
‘
cht.pv’、
‘
acot.pv’、
‘
fed2t.pv’、
‘
gew.pv’、
‘
co2t.pv’、
‘
aco.pv’、
‘
ao.pv’、
‘
n2.pv’、
‘
lge.pv’、
‘
co2.pv’、
‘
fed3t.pv’、
‘
our’及
‘
ph.pv’的特征矩阵来产生,
[0053]
以及
[0054]
ii)单独/仅使用来自培养的在线测得值。
[0055]
2.根据实施例1所述的方法,其特征在于,在线测得值至少用于培养的过程变量
‘
时间’、
‘
cht.pv’、
‘
acot.pv’、
‘
fed2t.pv’、
‘
gew.pv’、
‘
co2t.pv’、
‘
aco.pv’、
‘
ao.pv’、
‘
n2.pv’、
‘
lge.pv’、
‘
co2.pv’、
‘
fed3t.pv’、
‘
our’和
‘
ph.pv’。
[0056]
3.一种用于在哺乳动物细胞的培养期间将葡萄糖浓度调整至目标值的方法,其包括以下步骤
[0057]
a)至少确定培养的过程变量
‘
时间’、
‘
cht.pv’、
‘
acot.pv’、
‘
fed2t.pv’、
‘
gew.pv’、
‘
co2t.pv’、
‘
aco.pv’、
‘
ao.pv’、
‘
n2.pv’、
‘
lge.pv’、
‘
co2.pv’、
‘
fed3t.pv’、
‘
our’和
‘
ph.pv’的当前值,
[0058]
b)使用a)中确定的值,通过所述哺乳动物细胞培养的数据驱动模型确定培养基中的当前葡萄糖浓度,该模型是使用包含过程变量
‘
时间’、
‘
cht.pv’、
‘
acot.pv’、
‘
fed2t.pv’、
‘
gew.pv’、
‘
co2t.pv’、
‘
aco.pv’、
‘
ao.pv’、
‘
n2.pv’、
‘
lge.pv’、
‘
co2.pv’、
‘
fed3t.pv’、
‘
our’和
‘
ph.pv’的特征矩阵来产生,
[0059]
以及
[0060]
c)如果b)中确定的当前葡萄糖浓度低于目标值,则添加葡萄糖直到达到目标值,从而将葡萄糖浓度调整到目标值。
[0061]
4.根据实施例1至3中任一项所述的方法,其特征在于,过程变量选自包含以下过程变量的群组:活细胞密度、活细胞体积、培养基中的葡萄糖浓度和培养基中的乳酸浓度。
[0062]
5.根据实施例1至4中任一项所述的方法,其特征在于,该方法在没有采样的情况下进行并且仅使用来自所述培养的在线确定/测得值。
[0063]
6.根据实施例1至5中任一项所述的方法,其特征在于,数据驱动模型通过机器学习来产生。
[0064]
7.根据实施例1至6中任一项所述的方法,其特征在于,数据驱动模型是使用从包括人工神经网络和集成学习的组中选择的方法来产生。
[0065]
8.根据实施例1至7中任一项所述的方法,其特征在于,数据驱动模型是使用随机
森林方法来产生。
[0066]
9.根据实施例1至7中任一项所述的方法,其特征在于,数据驱动模型是使用mlpregressor方法来产生。
[0067]
10.根据实施例1至7中任一项所述的方法,其特征在于,数据驱动模型是使用xgboost方法来产生。
[0068]
11.根据实施例1至10中任一项所述的方法,其特征在于,数据驱动模型是通过监督学习来产生。
[0069]
12.根据实施例1至11中任一项所述的方法,其特征在于,数据驱动模型是通过交叉验证进行验证。
[0070]
13.根据实施例12所述的方法,其特征在于,交叉验证为10倍交叉验证。
[0071]
14.根据实施例1至13中任一项所述的方法,其特征在于,数据驱动模型是使用训练数据集来产生,该训练数据集包含至少10次培养运行。
[0072]
15.根据实施例14所述的方法,其特征在于,训练数据集包含至少60次培养运行。
[0073]
16.根据实施例1至15中任一项所述的方法,其特征在于,可用于模型产生的数据集的大约80%用作训练数据集,且其余数据集用作测试数据集。
[0074]
17.根据实施例1至16中任一项所述的方法,其特征在于,
[0075]
a)将可用于建模的数据集以70:30与80:20之间的比率随机划分为训练数据集和测试数据集,
[0076]
b)形成模型,
[0077]
c)从训练数据集确定用于确定数据集的过程变量的平均值和标准偏差,并且从测试数据集确定用于确定数据集的过程变量的平均值和标准偏差,
[0078]
d)重复步骤a)到c)直到达到可比较的平均值和标准偏差,即相对于测试数据集和训练数据集在彼此的10%以内,优选地在彼此的5%以内,其中在a)下获得的划分对于/随着每次新运行都不同。
[0079]
18.根据实施例1至17中任一项所述的方法,其特征在于,用于产生数据驱动模型的数据集各自含有相同数量的数据点。
[0080]
19.根据实施例1至18中任一项所述的方法,其特征在于,数据集中用于产生数据驱动模型的数据点各自针对相同的培养时间点。
[0081]
20.根据实施例1至19中任一项所述的方法,其特征在于,通过插值获得数据集中的缺失数据点。
[0082]
21.根据实施例20所述的方法,其特征在于,通过三阶多项式拟合获得葡萄糖浓度和/或活细胞体积的缺失数据点。
[0083]
22.根据实施例20至21中任一项所述的方法,其特征在于,通过单变量样条拟合获得乳酸浓度的缺失数据点。
[0084]
23.根据实施例20至22中任一项所述的方法,其特征在于,通过peleg拟合获得活细胞密度的缺失数据点。
[0085]
24.根据实施例1至23中任一项所述的方法,其特征在于,每个数据集至少每144分钟含有一个数据点。
[0086]
25.根据实施例1至24中任一项所述的方法,其特征在于,每个数据集至少每60分
钟含有一个数据点。
[0087]
26.根据实施例1至25中任一项所述的方法,其特征在于,每个数据集大约每5至10分钟含有一个数据点。
[0088]
27.根据实施例1至26中任一项所述的方法,其特征在于,哺乳动物细胞是cho细胞。
[0089]
28.根据实施例1至27中任一项所述的方法,其特征在于,哺乳动物细胞是cho-k1细胞。
[0090]
29.根据实施例1至28中任一项的方法,其特征在于,哺乳动物细胞表达并分泌治疗性蛋白质。
[0091]
30.根据实施例1至29中任一项所述的方法,其特征在于,哺乳动物细胞表达并分泌抗体。
[0092]
31.根据实施例30所述的方法,其特征在于,该抗体是单克隆和/或治疗性抗体。
[0093]
32.根据实施例30至31中任一项所述的方法,其特征在于,该抗体不是标准igg抗体,即是野生型四链全长抗体或是复合抗体,即包含附加抗体和/或与标准抗体相比非抗体结构域的抗体。
[0094]
33.根据实施例1至32中任一项所述的方法,其特征在于,数据驱动模型利用训练数据集而产生,该训练数据集仅包含复杂igg培养运行。
[0095]
34.根据实施例1至33中任一项所述的方法,其特征在于,数据驱动模型利用训练数据集而产生,该训练数据集还包含标准igg培养运行。
[0096]
35.根据实施例1至34中任一项所述的方法,其特征在于,哺乳动物细胞表达并分泌复杂或标准igg。
[0097]
36.根据实施例1至35中任一项所述的方法,其特征在于,培养体积为300ml或更少。
[0098]
37.根据实施例1至36中任一项所述的方法,其特征在于,培养体积为250ml或更少、200ml或更少、100ml或更少、75ml或更少、在200与250ml之间、或在50与100ml之间。
[0099]
38.根据实施例1至37中任一项所述的方法,其特征在于,培养为补料分批培养。
[0100]
39.根据实施例1至38中任一项所述的方法,其特征在于,培养在搅拌釜反应器中进行。
[0101]
40.根据实施例1至39中任一项所述的方法,其特征在于,培养中存在浸没式充气。
[0102]
41.根据实施例1至40中任一项所述的方法,其特征在于,培养是在一次性生物反应器(sub)中进行。
[0103]
42.根据实施例1至41中任一项所述的方法,其特征在于,哺乳动物细胞是悬浮培养的或者哺乳动物细胞是悬浮生长的哺乳动物细胞。
[0104]
43.根据实施例1至42中任一项所述的方法,其特征在于,数据驱动模型是通过回归分析来产生。
[0105]
44.一种活细胞体积在产生数据驱动模型中作为目标参数的用途,其用于确定以300ml或更少体积培养哺乳动物细胞的过程变量。
[0106]
45.根据实施例44所述的用途,其特征在于,过程变量选自包含以下过程变量的群组:活细胞密度、活细胞体积、培养基中的葡萄糖浓度和培养基中的乳酸浓度。
[0107]
46.根据实施例44至45中任一项所述的用途,其特征在于,在不采样的情况下进行培养。
[0108]
47.根据实施例44至46中任一项所述的用途,其特征在于,哺乳动物细胞是cho细胞。
[0109]
48.根据实施例44至47中任一项所述的用途,其特征在于,哺乳动物细胞是cho-k1细胞。
[0110]
49.根据实施例44至48中任一项的用途,其特征在于,哺乳动物细胞表达并分泌治疗性蛋白质。
[0111]
50.根据实施例44至49中任一项所述的用途,其特征在于,哺乳动物细胞表达并分泌抗体。
[0112]
51.根据实施例50所述的用途,其特征在于,该抗体是单克隆和/或治疗性抗体。
[0113]
52.根据实施例50至51中任一项的用途,其特征在于,抗体不是标准igg抗体或者是复合抗体。
[0114]
53.根据实施例44至52中任一项所述的用途,其特征在于,数据驱动模型利用训练数据集而产生,该训练数据集仅包含复杂igg培养运行。
[0115]
54.根据实施例44至53中任一项所述的用途,其特征在于,数据驱动模型利用训练数据集而产生,该训练数据集还包含标准igg培养运行。
[0116]
55.根据实施例44至54中任一项所述的用途,其特征在于,哺乳动物细胞表达并分泌复杂或标准igg。
具体实施方式
[0117]
为了能够实现高通量的测试培养,特别是对于复杂分子和分子形式,培养容器的尺寸必须减小并且培养必须自动化。培养的成功取决于受控的过程变量,并且只有在提供最佳培养条件时才能以高产率生产所需分子。因此,需要对相关过程变量进行快速高效的控制,以便能够设置相应的过程变量并保持最佳培养条件。这种控制对于小规模平行培养尤其必要,因为每个培养都必须单独监测。尤其是所谓的离线过程变量在这里是一个问题,因为一方面所需的采样和单独分析导致时间偏移,即培养继续并且一旦离线确定的过程变量与实际过程变量不同,过程变量,且另一方面,与在线可用过程变量相比,采样点的数量要小得多,这导致对该过程变量的时间控制较差。
[0118]
因此,本发明的一个目的是使不能在线确定而只能离线确定的过程变量(特别是由于所采用的培养容器的尺寸)如同在线可用的过程变量一样,在所使用的培养规模上,基于数据驱动模型并且实时可得。
[0119]
为了生产重组蛋白,生物反应器大多使用补料分批工艺进行[4]。除了补料分批工艺外,还有其他操作模式,诸如分批工艺和连续培养模式。
[0120]
分批补料或补料工艺是部分开放系统之一。此工艺的优点是在此工艺期间可以向培养物中加入营养物质,诸如葡萄糖、谷氨酰胺和其他氨基酸。可以避免由此产生的底物限制,并且可以确保更长的处理时间。底物可以连续添加或以(一种或多种)浓缩丸剂的形式添加。合适的补料策略可用于更好地控制抑制作用和有毒副产物的积累。然而,这需要足够的知识以及对过程的控制。
[0121]
为了在哺乳动物细胞(诸如cho细胞)的培养期间提供和保持最佳条件,几乎只使用生物反应器[2]。所用的生物反应器大部分为搅拌釜式反应器。培养在悬液中进行,即具有悬浮生长的细胞。
[0122]
需氧哺乳动物细胞,诸如cho细胞,需要氧气来维持其细胞代谢。通常通过对培养液进行浸没式充气来为细胞提供氧气。反应器中溶解氧的浓度是需氧细胞培养最重要的参数之一。溶解在培养基中的氧浓度由许多传输阻力决定。扩散导致氧气从气泡中传输到细胞中,从而使得其最终被细胞代谢。描述了可以使用氧气传输速率(输氧速率,缩写为otr)来进行转运机制,而细胞本身对氧气的消耗可以使用氧气消耗速率(吸氧速率,缩写为our)来确定[2]。适当的废气分析可以提供计算our和otr所需的数据。过程变量(诸如温度、ph值和溶解氧浓度)由合适的传感器监测,并包含在培养内要控制的参数中。这些过程变量对哺乳动物细胞系的有效生产力具有显著影响[3]。
[0123]
为了缩短生物反应器的开发和设置时间,研究和开发越来越关注一次性技术(一次性生物反应器;缩写:sub)。这些系统的巨大优势在于不需要复杂的清洗过程和必要的复杂且昂贵的清洗方法,诸如cip(原位清洗)和sip(原位消毒)。
[0124]
自动化高通量培养系统诸如ambr250系统(自动化微型生物反应器)有助于加速药物开发。该系统内有十二个一次性生物反应器,每个生物反应器的体积均为250ml。自动液体处理器用于移液和采样。操作由中央过程软件控制。整个ambr250系统位于层流箱下方,以确保操作期间的无菌环境。
[0125]
在过去的二十年中,软传感器在工业上越来越多地用于过程变量的监测[6]。所述过程变量通常只能通过高分析工作或在外部(即离线)来确定。特别是,在小规模采用一次性系统时,通常无法安装所需的额外传感器(空间和可用性或与一次性生物反应器的连接性,可能无法进行伽马辐照等)。因此,缺乏重要过程变量的连续数据,尤其是在小规模培养时,器可用于过程监测并允许调节所述过程变量,即过程目标参数。“软传感器”这个名称结合了“软件”和“传感器”这两个术语。术语“软件”表示模型的计算机辅助编程。这些模型的输出提供有关培养的信息,特别是过程变量的实时值,原本由于缺少相应的物理传感器而无法获得这些信息[5]。
[0126]
基本上,软传感器可以划分为两类,模型驱动的软传感器和数据驱动的软传感器。
[0127]
模型驱动的软传感器受制于理论过程模型。这些需要详细了解正在进行的过程并使用状态微分方程描述所述过程。这意味着必须使用机械模型来描述过程的动态行为。此类模型主要是为过程工厂的规划和设计而开发的,并侧重于描述理想平衡状态。
[0128]
在数据驱动的软传感器(所谓的黑盒模型)中,使用基于机器学习的模型。其中包括经验模型,它使用历史数据来描述过程变量的相关性。生物过程是复杂的,且关于培养的哺乳动物细胞代谢的各个和任一方面还没有完全了解。
[0129]
数据驱动的软传感器在制药行业的应用领域很大。通常,对培养进行监测和记录。
[0130]
本发明人现已发现,此类历史数据可用于产生数据驱动模型,用于离线过程变量的在线估计。
[0131]
过程变量主要是实时确定的,即可用的;它们通常只能通过增加分析工作和相关的时间偏移来确定。此外,对于某些过程变量(诸如生物量或某些底物和产物浓度)的在线监测,并非总是可以使用稳健且长期稳定的在线传感器系统[7]。虽然这些参数包含有关培
养过程的重要信息,但它们仅在培养期间的有限时间点可用,即在采样和离线分析的那些时间点可用。
[0132]
利用小型系统,诸如ambr250系统,由于缺少探头端口,无法测量某些过程变量,诸如浊度和/或电导率。此外,由于它们的设计,一些常见的探头需要相对较大的空间,这在这些小容量系统中是不可用的。
[0133]
机器学习是应用算法来描述数据集的基本结构。机器学习可以划分为两部分:监督学习和非监督学习。
[0134]
当模型准备基于训练数据对未来或未知数据进行预测时,使用监督学习。它是受监督的,因为训练数据集已经含有有关所需输出值的信息。一个实例是垃圾邮件的过滤[10]:因此,该算法接收由垃圾邮件和非垃圾邮件组成的数据集,该数据集已经含有有关它通过学习阶段的垃圾邮件/非垃圾邮件的信息。利用未标记的新电子邮件,该算法现在尝试预测它是什么类型的消息。由于这是分类目标变量(垃圾邮件/非垃圾邮件),因此使用术语“分类”。
[0135]
在非监督学习的情况下,尝试获取数据集内的关系,但不向算法提供目标变量。重点是探索数据的底层结构,以便从中提取有意义的信息。该组最简单的实例是聚类。在此探索性数据分析中,尝试将数据集划分为有意义的子组,而无需事先了解实际的组成员身份。
[0136]
如果目标变量是连续变量,则称为回归或回归分析。用于描述回归模型的变量称为自变量或解释变量。基于此,尝试找到输入变量与目标参数之间的数学关系,以便能够预测结果。
[0137]
根据本发明的方法使用监督学习,其中通过回归描述目标变量。
[0138]
建模可以在以下步骤中示意性地对齐:目标变量的预处理、学习、评估和估计。
[0139]
需要对数据进行预处理,以确保模型能够正确解释其所基于的信息。数据集以特征矩阵x的形式准备,且含有m个特征(列)和n行,因此其表示解释变量。每行n含有特定数据点的特征说明。
[0140][0141]
目标变量排列在向量y中。因此,特征矩阵x
(n)
的每一行都含有目标变量y
(n)
的相关值的信息。
[0142]
统计分析用于识别合适的特征。一旦确定了合适的特征位置并创建了相应的特征矩阵,模型就可以使用子集(整个数据集的70%-80%)进行学习。该子集称为训练数据集。
[0143]
典型的数据预处理可能包括为模型提供标准化形式的数据集。因此,每个特征的数据都具有标准正态分布的特性,均值为0,且标准偏差为1。这增加了特征彼此之间的可比性,并使学习算法能够实现其最佳性能[10]。
[0144]
学习是模型构建的核心部分。在学习期间,模型试图理解和识别数据之间的关系。每个模型都遵循具有特定参数的数学公式。这些在训练过程内进行调整,以尽可能好地描述数据之间的关系。
[0145]
一些模型,诸如神经网络,还有在学习期间不会改变的其他参数。这些被称为超参数。它们影响模型的复杂性或学习过程的速度,并在训练过程之前确定。选择正确的超参数没有固定的公式。因此,不同的模型使用不同的超参数进行训练,然后进行测试。只有这样才能判断哪种模型最合适。
[0146]
随机化和基于栅格的算法用于搜索超参数的最佳组合。每个超参数由具有不同值的列表表示。模型在网格搜索(gridsearch)中使用来自相应列表的每种可能的组合进行训练。通过随机搜索可以减少所需的计算工作量。使用不同的随机参数组合,其中计算工作量可以在此预先确定。在一个实施例中,首先利用随机搜索来执行模型以粗略估计超参数,然后执行网格搜索以对超参数进行微调。学习的目的是训练模型,以便将偏差和方差保持在尽可能低的水平。
[0147]
模型通常比使用未知数据集进行后续预测更好地学习训练数据之间的关系。这种行为称为过拟合。因此,该模型已经记住了训练数据集并用新数据描述了不够准确度的关系。类似的行为也可以归因于过度的方差。此处,模型使用过多的输入参数来训练数据集,导致复杂的模型只适合具有高数据方差的数据集。因此,该模型在无法映射实际关系的情况下学习了数据的噪声。
[0148]
另一方面,如果模型不够复杂,无法对测试数据集的变化做出反应,则此被称为欠拟合。那么偏差太大,模型只能不精确地映射训练数据与测试数据的关系。
[0149]
在学习期间,训练数据集的k倍交叉验证提供了避免模型过拟合的可能性[11]。将训练数据集划分为k个子集。然后,使用k-1个子集来训练模型,且其余子集用作测试数据集。将这个过程重复k次。以这种方式,对k个模型进行训练,且获得k个目标变量的估计。
[0150]
每次运行都会产生模型的性能估计ei。例如,作为回归的性能估计,使用均方偏差作为误差的度量。在实践中,在大多数情况下,10倍交叉验证已被证明是偏差和方差的良好折衷方案[12]:
[0151][0152]
人工神经网络(ann)于1943年由warren mcculloch和walter pitts用神经元的数学模型描述。通过这种方式,可以使生物系统中的信息传递变得易于理解[13]。frank rosenblatt然后能够将人工神经元的mcculloch-pitts模型与学习规则联系起来,从而描述感知器[14]。感知器仍然是ann的基础。
[0153]
简单的感知器有n个输入x1,....,xn∈ir,每个输入都有一个加权w1,....,wn∈ir。输出由o∈ir表示。具有适当加权的输入信号的处理是传播函数(输入函数)σ,
[0154][0155]
它描述了神经元的网络输入。通过激活函数φ,
[0156]
o=φ(σ),
[0157]
然后确定感知器的输出o。可以对φ使用各种函数,这可以导致感知器的激活。
[0158]
因此,激活函数根据阈值和网络输入计算神经元被激活的强度[15]。如果这些神经元中的几个以合适的结构互连,则可以映射输入层与输出层之间的复杂关系。简单神经
元的这种结构互连的最简单形式是前馈网络。它们按层排列,且由输入层、输出层和几个隐藏层组成,取决于结构。
[0159]
在前馈网络(所谓的多层感知器)中,一个层中的每个神经元都连接到下一层中的所有其他神经元。因此,这些网络向前传播通过网络创建的信息内容。每个神经元使用初始随机选择的权重对输入信号进行加权,并添加偏差项。该神经元的输出对应于所有加权输入数据的总和。根据一层神经元的数量和隐藏层的数量,可以确定神经网络的复杂性。
[0160]
含有误差反馈(反向传播)的多层前馈网络主要用于ann的监督学习[16]。
[0161]
这种神经网络的训练可以划分为三个步骤:
[0162]
·
步骤1:前馈;
[0163]
·
步骤2:误差计算;
[0164]
·
步骤3:反向传播。
[0165]
在第一步中,对网络的输入层进行输入,该输入通过网络逐层传播,直到有来自网络的输出。将网络的输出与第二步中的期望值进行比较,且使用误差函数计算网络的误差。根据当前权重,隐藏层内的每个神经元对计算误差的贡献程度不同。在第三步中,误差通过网络向后传播,其中权重根据单个神经元的权重对误差的贡献进行调整。反向传播算法的目标是最小化误差,且通常使用梯度下降法[17]。利用这种方法,网络的输出与预期输出之间的二次距离被计算为误差函数:
[0166][0167]
为了计算每个神经元的权重对误差的贡献,误差函数err必须从考虑的权重w
ij
导出。因此,这里只能使用连续和可微的激活函数[17]。这决定了将在下一个迭代步骤中使用的权重调整增量。这种关系可以用数学方式描述如下:
[0168][0169]
学习速率η与迭代次数一起,是在训练模型之前建立的超参数。重复这两个步骤,直到达到最大迭代次数或定义的误差值,并且对于未知输入可以获得良好的结果。
[0170]
此外,随机森林(rf)算法可用于回归问题的机器学习[18]。rf通过大量决策树进行学习,因此属于集成学习器的类别。决策树可以从根(顶部节点,没有前身)展开。每个节点根据特征将数据集划分为两组。根的后继可以是叶子(无后继)或节点(至少有一个后继)。节点和叶子由一条边连接。在回归问题的情况下,[19]
[0171]
·
为每个内部节点(包括根)分配一个特征;
[0172]
·
将要预测的目标变量的特定值分配给决策树的每个叶子;
[0173]
·
对于每条边,将一个关系分配给一个阈值。
[0174]
在优选实施例中,rf使用根据breiman[18]的打包原理(自举聚集原理)来创建合适的训练集,其中训练集是通过从整个训练数据集进行替换采样来创建的。一些数据可能会被多次选择,而其他数据则不会被选择作为训练数据。训练集的数量总是与整个训练数据集的数量相对应。每个所选择的训练集用于使用决策树(分类器)产生决策。然后对所有训练集的决策求平均,其中多数决策确定最终分类。因此,自举样品的产生在单个分类器之
间产生了低相关性。此外,可以减少单个分类器的方差且提高整体分类性能[18]。
[0175]
在优选实施例中,特征用于在树的创建期间决定分割(节点的划分),该特征做出关于数据集的特征的随机选择的最清楚的决定。所选择的分割不再被选为所有特征的最佳分割,而是随机选择的特征内的最佳分割。由于这种随机化,树的偏差(失真、系统误差)在创建过程中会增加。由于rf中含有的所有树的平均值的形成,方差会减小。减少的方差比偏差的增加具有更大的附加值,从而提高模型的准确性[20]。
[0176]
此外,由于始终考虑所有单个决策的平均值,因此在rf预测中几乎可以防止模型过拟合[18]。
[0177]
xgboost(extreme gradient boosting)使用回归树的集合作为模型形成基础。已经描述的打包原理和特殊的增强技术都用于训练集合,以实现最准确的预测。简单来说,增强技术可以看作是由许多弱学习器组成的梯度下降方法的组合[21]。这些弱学习器通常并不比随机猜测更精确,并且在创建集合的过程中被分组为强学习器。这种弱学习器的典型实例是只有一个节点的简单回归树。增强算法的原理是选择难以分类的训练数据,以便通过这些弱学习器从这些分类不佳的对象中学习,从而提高集合的性能。由于xgboost的复杂性,该算法被认为是黑盒。然而,由于其可扩展性和解决问题的速度,该算法非常成功地用于不同机器学习模型的直接比较[22]。
[0178]
xgboost实现的方法结合了梯度下降法和增强技术,下面使用tianqi chen的原始文献“xgboost:a scalable tree boosting system”来描述[22]。
[0179]
利用由k个决策树组成的集合,该模型可以根据以下条件进行描述:
[0180][0181]
其中fk是单个决策树的预测。在所有决策树中看到,可以做出预测
[0182][0183]
其中xi是第i个数据点的特征向量。为了训练模型,优化损失函数l。在回归问题的情况下,使用rmse(均方根误差):
[0184][0185]
正则化是防止模型过拟合的重要部分:
[0186][0187]
其中t是叶子的数量,且w
2j
是第j个叶子的实现得分。如果将正则化和损失函数放在一起,模型的基本目标函数可以公式化为
[0188]
obj=l ω,
[0189]
其中损失函数确定预测能力,且正则化控制模型的复杂性。使用梯度下降法优化
目标函数。给定要优化的目标函数在每次迭代中计算梯度下降
[0190][0191]
并且沿着下降梯度变化,从而使目标函数obj最小化。
[0192]
为了创建回归树,内部节点基于数据集的特征进行划分。所得边定义了允许划分数据集的值范围。回归树内的叶子被加权,其中权重对应于预测值。迭代次数表示打包和增强过程重复的频率。xgboost算法提供非常广泛的超参数列表,这些超参数对形成良好模型有很大帮助。
[0193]
无论使用何种模型,相关性都可用于评估和表示两个变量之间的线性关系。pearson相关系数r(或r2)提供用于评价这种关系的常用度量。它是无量纲的,且根据以下公式计算:
[0194][0195]
并且在-1≤r≤ 1以内变化。计数器将两个变量x和y的偏差乘积之和描述为均值,这对应于经验协方差s
xy
。分母是单个经验标准偏差s
x
和sy的乘积的根。要相关的数量的平均值被描述为和根据fahrmeir[23]的线性关系可以解释为:
[0196]
·
r《0.5:弱线性关系
[0197]
·
0.5≤r《0.8:中等线性关系
[0198]
·
0.8≤r:强线性关系
[0199]
在相关分析中,需要注意的是,这里只能示出线性关系。bravais-pearson相关系数因此不适用于描述非线性关系。这可能意味着尽管相关系数为0.0≤r≤0.2,但变量之间存在很强的非线性相关性。
[0200]
通过互信息,可以确定两个随机变量的非线性相关性。它用于信息论[24]。在概率的帮助下,描述随机变量与第二个随机变量相比的信息内容。基本的形式关系是:
[0201][0202]
kraskov等人和ross等人相应地扩展了这种方法,以便它可以用于选择合适的连续变量[25][26]。
[0203]
必须使用适当的指标来比较不同的模型。在这些的帮助下,可以就模型描述目标变量的准确性做出陈述。
[0204]
确定系数r2表示模型可以描述目标变量y的方差的哪个比例。确定系数可根据以下来计算:
[0205][0206]
其中是第i个实例的估计目标变量,且yi是相关联的真实值。是均值。确定系数可以取0与1之间的值。确定系数越接近1,模型就越能拟合目标变量。
[0207]
均方根误差(rmse)是另一种可用于确定模型质量的统计量度。这里计算实际值与
估计值的均方根距离:
[0208][0209]
通过对误差求平方然后形成根,rmse可以解释为待估计变量的标准偏差。其中n是观察次数,且是目标变量y的估计值。rmse表示的误差是绝对误差值,它根据所检查的目标参数提供不同大小的值。因此,将rmse与均值联系起来是有意义的:
[0210][0211]
因此,可以相对于平均真实值计算rmse。这允许更好地评估不同大小的目标变量的误差。
[0212]
方法
[0213]
利用根据本发明的方法,可以在培养期间,特别是在小培养规模上,从在线过程变量中实时确定细胞生长,即细胞密度的时间线,和某些代谢物,特别是葡萄糖和乳酸的时间线。因此,利用根据本发明的方法,可以为以前不能实时获得而只能离线获得的过程变量提供实时值。这代表对细胞生长和某些代谢物(特别是葡萄糖和乳酸)的时间线的常规确定方法的改进,因为根据本发明的方法不需要从培养基中采样。
[0214]
在优选实施例中,根据本发明的方法用于从在线过程变量中确定培养体积为300ml或更少的哺乳动物细胞的补料分批培养中的细胞密度、葡萄糖浓度和乳酸浓度,其中该方法在没有采样即反馈控制采样的情况下进行。
[0215]
根据本发明的方法允许在小规模上,即培养体积为300ml或更少的情况下,完全自动地进行培养,即无需采样,其中相关过程变量,诸如细胞密度,不能在线确定,而只能离线确定。
[0216]
本发明的方法特别适合于小规模监测和控制哺乳动物细胞的培养。
[0217]
在根据本发明的方法中,提供一种在cho细胞培养中确定活细胞密度、葡萄糖和乳酸浓度作为目标参数的方法,其中该方法采用基于数据的软传感器。机器学习模型用于描述不同的目标变量。
[0218]
本发明至少部分基于以下发现:用于模型产生的过程变量的选择对所确定的目标过程变量的质量具有显著影响。
[0219]
此外,本发明至少部分地基于将现有数据集划分(即分配)成训练数据集和测试数据集的类型影响模型质量的发现。
[0220]
此外,本发明至少部分地基于所产生的抗体类型影响最佳靶参数的选择的发现。
[0221]
下面使用155个示例性数据集描述根据本发明的方法,这些数据集是从ambr250系统中的培养获得的。这不应被理解为限制根据本发明的教导或根据本发明的方法,而是作为根据本发明的教导的示例性应用。已经用相同或不同培养系统产生的其他数据集同样可以很好地用于根据本发明的方法并且在根据本发明的方法中使用。
[0222]
分析并检查了155个数据集的合适特征。相应的插值策略用于映射目标参数,使得所选择的模型可以在离散时间点为所有目标参数提供值。评估模型的误差和模型质量。基于它的方法允许提供相应目标变量/过程变量的稳健且精确的模型。
[0223]
数据集中培养产生的抗体的分子形式不同。下表1中显示了各种项目和分子形式以及相应培养数量的概述。
[0224]
表1:数据概览。
[0225][0226]
与整个培养过程相关的数据,即在线参数集,以及相关的日期和时间戳用于每次培养。不同过程值的数据密度随时间线而变化。数据密度的这些偏差可归因于以下事实:由于系统的原因,如果测得值被为每个测得值专门定义的增量改变,则仅记录在线参数的新数据点。为了提供连续的过程数据并确保运行可以相互比较,相应的在线参数被插入所有丢失的时间戳。
[0227]
应该注意的是,对于在线过程变量,过多的数据平滑将会导致测得值波动的损失。然而,这种噪声也代表正在发生的任何过程相关变化,并且包含在过程值中作为信息。因此,重要的是不要过度平滑过程值,且即使在插值之后也可以访问工艺过程中的更改。
[0228]
离线数据含有不同数量的分析值,这取决于培养期间的样品数量(在8与13个之间)。每个数据集含有每个数据点的日期和时间戳以及离线参数的相关分析值。
[0229]
通过对在线数据和离线数据进行插值的预处理产生数据集,该数据集同时含有所有过程变量的相同数量的数据点,无论它们是在线过程变量还是离线过程变量。分析是基于内插数据集。如果对于所有在线过程变量和离线过程变量,数据点在相同频率和相同时间可用,则不需要这种插值。
[0230]
由于可用的在线数据和离线数据的预处理,由于不同测量频率而导致的单个过程变量的不同时间曲线被标准化为统一的时间曲线,即单个时间线。由技术和过程管理引起的不良值被确定出和取消选择或校正,且现有的时间间隙被关闭,使得一个培养的一个数据集和所有培养的所有数据集中的所有过程变量关于过程变量的时间和数量是一致的。
[0231]
为了使在培养开始时打开控制或在培养结束时关闭控制所引起的测量信号的波动不会伪造模型的形成,在培养的第一和最后12小时收集的数据没有被使用。在特定实例中,这意味着使用了从第0.5天到第13.5天的时间范围。这保证了过程变量的变化只能归因于细胞培养中的过程。对整个数据集进行在线数据的插值。图1显示了过程值
‘
ao.pv’的线性插值的实例。
[0232]
从图1中可以看出,很好地描述了具有线性插值的在线信号的过程。在开始时(《0.5天),可以看到当控制开始时测得值是如何波动的。峰值(短时间内较大的过程值变化)也可以通过这种类型的插值很好地映射。
[0233]
对于离线数据,获得的分析值(vcd、vcv、葡萄糖、乳酸)用三种不同的插值拟合。图
2显示了使用不同拟合方法对vcd进行插值的实例。
[0234]
计算相应的确定系数r2以评估vcd的单个插值。单变量样条在此处实现了最高r2值,但倾向于显著过拟合。因此,单变量样条几乎准确地描述了每个测得值,但并未描绘生物系统的典型生长曲线。另一方面,peleg拟合与多项式拟合之间存在较小差异。然而,peleg拟合可以更好地描述生物系统的不同生长阶段,因此用于vcd目标变量的插值[27]。
[0235]
乳酸和葡萄糖曲线的插值表明,单变量样条以更好的r2映射离线数据,并更好地描述乳酸情况下的曲线。由于多项式拟合从第10天开始为乳酸内插负值,因此单变量样条的插值被定义为乳酸的目标向量y。然而,对于葡萄糖,多项式拟合(三阶)用于描述目标变量(葡萄糖:单变量样条(r2=0.999)和多项式拟合(r2=0.958);乳酸:单变量样条(r2=0.999)和多项式拟合(r2=0.959))。
[0236]
此外,用于预处理的离线数据点太少(三个或更少)的数据集不再用于分析。这是两个数据集的情况。因此,整个内插和调整的数据集含有153个培养。
[0237]
由于最大分辨率为五分钟的内插数据集含有大量数据点,因此仅以1/10天的分辨率进行分析以减少计算工作量。程序可用于此目的。
[0238]
图3显示了来自项目2(12个培养)的数据集。可以看出,不同的插值方法(peleg拟合、单变量样条和多项式拟合)对相关性强度的影响很小。
[0239]
在图3的散点图中,在线参数被显示为特征(线)。列代表vcd的不同插值。散点图的椭圆总是含有95%的数据。椭圆越靠近,变量之间的线性关系越强。计算的bravais-pearson相关系数如下表2所示。
[0240]
表2:来自项目b的样品数据集的pearson相关系数的数值对应于图3的数值。
[0241][0242]
以“o2.pv”的值为例,计算出的插值系数彼此非常接近(0.9547;0.9490;0.9490)。
[0243]
相应地对整个数据集进行了相关性分析。下面的表3显示了以此方式确定的bravais-pearson相关系数。
[0244]
表3:计算整个数据集(153个培养)的pearson相关系数,目标变量vcd适合peleg拟合。
[0245][0246]
与单次ambr250运行的相关性分析(参见前面的表3和图3)相比,相关性分析显示出整个数据集的线性关系明显更弱。除了相关性的强度之外,对整个数据集的分析还产生了其他作为最佳候选的在线参数。还发现自变量彼此相关。下表4部分显示出参数“o2.pv”和“n2.pv”与其他自变量的相关性。
[0247]
表4:自变量相互之间的相关性,以o2.pv和n2.pv为例,它们在之前对项目b进行的相关性分析中具有最高的相关性值。
[0248][0249]
当自变量彼此相关时,一个是指多重共线性。如此处使用
‘
o2.pv’的实例所示,图3中
‘
n2.pv’和
‘
o2.pv’的两个最佳相关系数与其余独立参数之间存在明显的线性关系。
[0250]
图4显示出整个数据集的目标变量vcd的所有特征的计算信息内容(互信息)。图4显示出一些可用特征具有关于vcd目标变量的高级信息。对于vcd,互信息因此可以具有最高索引为
‘
时间’、
‘
cht.pv’、
‘
acot.pv’、
‘
fed2t.pv’、
‘
gew.pv’、
‘
co2t.pv’、
‘
aco.pv’、
‘
ao.pv’、
‘
o2.pv’、
‘
n2.pv’及
‘
lge.pv’。
[0251]
基于信息内容的计算和相关性分析的结果,选择最佳的十个过程变量(cht.pv、acot.pv、fed2t.pv、gew.pv、co2t.pv、aco.pv、ao.pv、lge.pv、o2.pv和n2.pv),并创建相应的特征矩阵x。该矩阵含有可用数据集的内插数据。选择特征的五分钟分辨率(f1...f
10
)和培养持续时间(以小时为单位)作为矩阵中的附加列:
[0252][0253]
训练数据集和测试数据集的划分方式使得这些数据集完全来自项目2的培养。根据特征矩阵的分布划分目标变量
‘
vcd’。
[0254]
为了检查所获得模型的质量,计算了整个测试数据集上误差的相对频率密度。使用目标变量vcd的mlpregressor(a)、随机森林(b)和xgboost(c)确定的模型的整个测试数据集的预测直方图在x轴上显示了估计vcd值与预测值相比的误差,且在y轴上显示误差的相对频率。所有三个分布都显示出向左偏斜的趋势,这表明vcd被低估了。此外,对所有直方图的检查表明,所有三个模型的估计都产生了可比较的结果。xgboost显示出计算误差的最均匀分布,然而这里也可以看到对目标变量的高估。
[0255]
对于每个模型,基于整个测试数据集计算rmse和r2。这两个值都与目标变量vcd的
peleg拟合有关。三个模型的结果总结于下表5中。
[0256]
表5:mlpregressor、随机森林和xgboost的vcd估计结果。
[0257]
模型rmser2mlpregressor0.380.58随机森林0.330.67xgboost0.340.66
[0258]
所有模型在rmse和确定系数方面都取得了可比较的结果。
[0259]
如果检查一些使用随机森林(最佳模型)确定的特定数据集,可以看出无法准确映射整个培养期间vcd的peleg拟合(见图5)。从第5天开始,图中上部的模型无法正确描绘数据与vcd的关系。该图的下部显示了相反的行为。该模型从一开始就估计出过高的vcd,因此无法对vcd进行足够精确的描述。
[0260]
现在令人惊讶地发现,将具有高信息内容的特征矩阵中的特征与具有显着较低信息内容但仍具有可确定信息内容的那些特征交换可以显着提高预测的质量。
[0261]
已经发现,通过特征
‘
co2.pv’、
‘
fed3t.pv’、
‘
our’和
‘
ph.pv’以及删除冗余特征
‘
o2.pv’(用n
2 and o2充气)来扩展矩阵导致预测质量的提高。
[0262]
改进后的特征矩阵含有以下14个特征:
‘
时间’、
‘
aco.pv’、
‘
acot.pv’、
‘
ao.pv’、
‘
cht.pv’、
‘
co2.pv’、
‘
co2t.pv’、
‘
fed2t.pv’、
‘
fed3t.pv’、
‘
gew.pv’、
‘
ph.pv’、
‘
n2.pv’、
‘
lge.pv’和
‘
our.pv’。
[0263]
此外,已经发现训练数据集和测试数据集的选择或划分对预测的质量有影响。
[0264]
当比较已经针对目标变量选择的训练数据集和测试数据集时,发现由项目2的培养组成的训练数据集的vcd分布具有平均值μ
训练
=84.60,标准偏差为σ
训练
=48.62,而测试数据集的平均值为μ
测试
=64.22,标准偏差为σ
测试
=38.02。
[0265]
已经发现,如果要对表达结构不同的蛋白质的细胞进行预测,则仅从一个项目中获取训练数据集是不利的。已经发现将训练数据集随机分布在整个现有数据集上是有利的。
[0266]
在本实例中,为了更均匀地分布数据集,产生了0与152之间的30个随机数(因为有153个数据集)。每个数字代表一次培养运行。重复产生随机数,直到可以使用训练模型实现关于测试数据集与训练数据集之间划分的可比平均值和标准偏差。最后的划分导致μ
训练
=80.72,σ
训练
=47.11和μ
测试
=80.11,σ
测试
=48.70,并且是用作进一步过程中两个数据集的分割比率。
[0267]
因此,在根据本发明的方法的一个实施例中,现有的优选预处理的数据集被划分为训练数据集和测试数据集,其中训练数据集是总数据集的70%-80%(在该实例中为80%,因此,123次培养运行),并且测试数据集含有整个数据集的20%-30%的数据(在本实例中,如上所述验证的整个数据集的30个随机选择的培养可用于验证模型)。
[0268]
然后使用扩展的特征矩阵以及数据集的新分布对模型进行训练和测试。为此保留了如上所述的优化超参数的策略。从利用新划分的训练数据集和测试数据集的vcd估计值的对应直方图可以看出,所有三个模型的误差分布都显著变窄了,这可以归因于对目标参数的更精确估计(图6)。
[0269]
所有三个模型都可以实现更清晰地围绕目标变量的真实值(0处的x轴)波动的误
mlpregressor0.180.95随机森林0.350.81xgboost0.270.88
[0284]
图12显示了来自测试数据集的示例性培养的乳酸的xgboost预测值。在上部分图像中可以看到对拟合乳酸过程的近乎理想的描述;在下部分,该过程可以用0.98的r2来描述。
[0285]
对于验证,首先进行了一项研究,以确定哪个模型能够最有高地描述测试数据集上特征的相互关系。为此,模型最初只提供了十个数据集用于学习。随着过程的进行,每个数据集的数量增加了十个。这导致了十二个训练过程,其中模型接收了10至120个数据集。每次训练结束后,目标变量都会基于测试数据集进行估计。计算各自的rmse。如上所述,测试数据集还由30个随机验证的所选择的数据集组成。vcd被选为目标变量。这导致了图13中描述的学习行为。
[0286]
如图13所示,与神经网络相比,随机森林和xgboost在使用少量数据集预测测试数据集时都能够实现更小的误差。然而,这种影响似乎随着训练数据集数量的增加而减弱,因此与其他两个模型相比,可以从大约80个数据集开始实现可比的误差。在最大数量为120个数据集的情况下,随机森林达到最低的rmse。但是,所有模型的误差都在非常窄的范围内。
[0287]
对测试数据集的30个培养的vcd预测模型的估计进行了详细评价。尽管在整个数据集(直方图、确定系数、rmse)上取得了良好的结果,但发现一些预测仍然显示出明显更大的偏差。图14显示了一个培养运行,其中vcd的估计过程明显高于实际分布。
[0288]
在项目1和项目3的培养中越来越多地观察到估计的准确性不足。在这两个项目的培养中,培养的细胞产生了复杂的分子形式。
[0289]
发现基于igg的形式的vcd(其具有天然igg抗体的特征性y形或大部分保留它)(项目2和4)平均高于具有复杂分子形式作为目标产物的细胞中的vcd(项目1和3),并且计算的细胞直径对于具有复杂分子形式的项目具有更高的值。
[0290]
图15以方框图的形式显示了每个样品中按y形igg(igg,项目2和4)和复杂igg(复杂,项目1和3)分组的项目的平均细胞直径,以及标准偏差。该图显示绿色方框图(复杂蛋白质格式;在每个时间点左侧)位于蓝色方框图(y形igg抗体;在每个时间点右侧)上方。在培养开始时,两种分子形式仍比较接近。随着培养时间的推移,具有复杂分子形式作为目标产物的细胞只会变得更大。相比之下,可以看到具有标准抗体的细胞在第7天之前一直变大,但随后没有发现细胞直径进一步增加。
[0291]
结果发现,对于igg形式,较高的vcd与较小的细胞直径之间的关系,以及复杂蛋白质形式中较小的vcd和较大的细胞之间的关系,导致vcd的预测不准确。
[0292]
还发现,活细胞体积(vcv)代表比vcd更合适的目标变量,不仅适用于产生复杂抗体形式的培养,而且适用于产生y形igg抗体的培养。
[0293]
vcv使用以下公式计算:
[0294][0295]
因此,与vcd相比,vcv是描述培养中活生物量的更好近似值。
[0296]
由于vcv的计算值与所有其他离线参数一样,仅含有采样次数,因此新目标参数利
用三阶多项式拟合。然后针对新的目标大小对模型进行训练和评价,如上文针对其他目标参数所述。
[0297]
rmse和确定系数用于评价单个模型。综上所述,具有14个特征的最佳模型取得了以下结果:
[0298]
表9:针对目标变量vcd的最佳模型的rmse和确定系数的比较
[0299]
模型rmser2mlpregressor0.230.86随机森林0.200.89xgboost0.220.85
[0300]
对于目标变量vcv,单个模型的计算误差和确定系数总结在下表10中:
[0301]
表10:针对目标变量vcv的最佳模型的rmse和确定系数的比较
[0302]
模型rmser2mlpregressor0.140.94随机森林0.110.97xgboost0.120.96
[0303]
通过使用目标变量vcv而不是vcd,所有模型都能够实现高于0.9的确定系数。模型的改进可以在较低的rmse和较高的r2值中看到。
[0304]
为了证明活细胞密度与细胞体积之间比较的改进结果,获得了代表整个训练集和测试数据集的估计的散点图。随机森林估计vcd和vcv的最佳结果。两个散点图如图16所示。
[0305]
如果将两个散点图相互比较,可以看出,vcv的预测更接近理想估计,并且与vcd的预测相比,测试数据集和训练数据集的分布明显更小。如果只考虑训练数据(蓝点),模型更好地学习特征与细胞体积的关系,而不是与活细胞密度的关系。因此,这些特征允许对所有训练模型的整个测试数据集的细胞体积进行更精确的估计。
[0306]
将抗体分成不同组和仅使用有限的数据集在方法训练方面影响质量的程度研究如下。
[0307]
如果针对目标参数vcv的过程分别考虑所有四个项目,则获得图17所示的方框图。可以看出,项目4中的vcv位于项目1和项目3的一侧与项目2的另一侧之间。这意味着来自项目1、3和4的数据集也可以归类为复杂的igg抗体格式(分类2)。因此,用这种分类重复计算。还测试了训练数据集和测试数据集的各种组合。结果在表11以及图18和图19中示出。
[0308]
表11:训练数据集和测试数据集的不同组合的rmse。
[0309][0310]
不同的组合表明,使用随机森林方法的预测取得了最佳结果,即最低的rmse。
[0311]
与vcd相比,当使用vcv作为目标参数时,rmse在训练数据集或测试数据集的所有组合中都显示出显著改善(减少)。
[0312]
训练数据集和测试数据集的不同组合显示出,根据分子格式选择数据集会影响目标参数的rmse。在使用标准格式的数据集进行模型训练和估计复杂格式的vcd或vcv的情况下,这种组合实现最高的rmse。使用复杂分子格式的数据集进行训练并预测vcd或vcv导致更小的rmse。如果混合数据集用于标准y-igg和复杂分子格式,则可以实现最小的rmse。
[0313]
此外,还根据训练数据集和测试数据集的估计对模型进行了评价,以检查已经训练的模型是否过拟合。目标变量vcv的训练模型是在测试数据集和训练数据集上估计的。根据rmse评价估计值,然后以条形图的形式显示测试数据集与训练数据集之间的差异(图20)。
[0314]
图20显示测试数据集的mlpregressor的误差率低于训练数据集的误差率。因此,计算出的差值为负。随机森林和xgboost在测试数据集上有较大的误差,这使得这里显示的差异为正。因此,基于决策树的两种模型都倾向于过拟合。
[0315]
现有技术
[0316]
现有技术使用诸如葡萄糖、乳酸、氨、vcd等参数(所有这些都是离线参数)作为随
机森林回归分析的输入变量来解释细胞内活动的动态行为,但不用于预测或建模离线参数。
[0317]
与现有技术相比,在本发明中,用于机器学习模型的参数是排他的在线参数(其用于控制发酵条件)。
[0318]
因此,本发明利用在整个培养过程中产生的典型在线测量参数和统计模型来估计诸如vcv、葡萄糖等参数,而不需要额外的传感器或采样。
[0319]
总结与展望
[0320]
通过内插现有的在线培养数据集和离线培养数据集,可以获得标准化和统一的数据集,其用于模型产生以预测仅离线可用的目标参数。
[0321]
对于离线数据(其被认为是进一步过程中的目标变量),必须找到一个插值,其可以代表性地描述相应目标参数的过程。由于活细胞密度与生物系统的生长过程有关,因此诸如多项式拟合或单变量样条拟合等常规插值通常仅以不充分的准确性来描述该目标参数。不正确的外推将导致对目标变量的错误描述。尽管所选择的差值产生了与r2相当的结果,但根据m.peleg[27]选择的差值最能描述细胞培养过程的生长过程。插值策略的背景在于结合了用于描述细胞生长的连续逻辑方程和用于描述死亡行为的镜像逻辑方程(费米方程)。
[0322]
相关性分析的结果仅在一定程度上不受插值策略选择的影响。
[0323]
通过将数据集调整为训练数据集和测试数据集的调适分割比,可以提高vcd目标变量估计的准确性。为此,针对目标变量的分布选择了验证数据集,以便彼此的平均值和标准偏差尽可能小。目标不是人为地产生更好的数据集进行预测。相反,假设之前产生的测试数据集不能用于足够准确地描述整个数据集。在此参考交叉验证作为相应的方法。
[0324]
细胞体积的计算以及与细胞大小的相关关系可以描述比vcd更好的生物量近似值,从而导致vcv作为新的目标参数。
[0325]
计算的细胞体积作为生物量描述的近似值,提供了比以前通过分析样品确定的培养物活细胞密度更高的关于过程特征的信息含量。细胞培养物的平均体积可以从测量的细胞平均直径得出。可以显示出细胞的大小,尤其是那些具有复杂靶分子作为产物的细胞,随着培养时间的增加而不断增加。然而,活细胞密度不能映射这种关系。最后,活细胞体积比活细胞密度可以更好地描述培养细胞的代谢活动。
[0326]
为了实时确定目标参数,应该在定义的时间间隔内(例如十分钟)进行估计。对于cho细胞,此间隔是可接受的分辨率,因为它们的倍增时间约为24小时。
[0327]
以下实施方案和附图仅用于说明本发明。保护范围由未决权利要求定义。然而,在不脱离根据本发明的原理的情况下可以对所公开的实施例进行修改。
[0328]
参考文献
[0329]
[1]j.glassey,et al.,biotechnol.j.6(2011)369-377.
[0330]
[2]f.garcia-ochoa,et al.,biochem.eng.j.49(2010)289-307.
[0331]
[3]e.trummer,et al.,biotechnol.bioeng.94(2006)1033
–
1044.
[0332]
[4]y.-m.huang,et al.,biotechnol.prog.26(2010)1400-1410。
[0333]
[5]bc mulukutla,et al.,met.eng.14(2012)138-149.
[0334]
[6]r.luttmann,et al.,biotechnol.j.7(2012)1040-1048.
[0335]
[7]t.becker and d.krause,chem.ing.tech.82(2010)429-440.
[0336]
[8]lz chen,et al.,bioproc.biosys.eng.26(2004)191-195.
[0337]
[9]p.kroll,et al.,biotechnol.lett.39(2017)1667-1673。
[0338]
[10]s.raschka and v.mirjalili,machine learning with python and scikit-learn and tensor-flow:the comprehensive practice manual for data science,deep learning and predictive analytics.frechen:mitp,2.,updated and expanded edition,2018.
[0339]
[11]hsu,chih-wie,et al.,"a practical guide to support vector classification,"taipei,pp.1-16,2003.
[0340]
[12]r.kohavi et al.,ijcai,14(1995)1137-1145.
[0341]
[13]ws mcculloch and w.pitts,bull.math.biophys.5(1943)115-133.
[0342]
[14]f.rosenblatt,psychol.rev.65(1958)386-408.
[0343]
[15]kriesel david,ed.,a brief overview of neural networks.
[0344]
[16]w.lu,“neural network models for distortional buckling behavior of cold-formed steel compression members,”2000.
[0345]
[17]r.rojas,neural networks:a systematic introduction.berlin and heidelberg:springer,1996.
[0346]
[18]l.breiman,“random forests,”machine learn.45(2001)5-32。
[0347]
[19]ro duda,et al.,pattern classification.sl:wiley interscience,2.ed.,2012.
[0348]
[20]l.breiman,machine learn.24(1996)123-140。
[0349]
[21]y.freund and re schapire,j.comp.syst.sci.55:119-139(1997).
[0350]
[22]t.chen and c.guestrin,“xgboost,”in the 22.acm sigkdd international conference(b.krishnapuram,m.shah,a.smola,c.aggarwal,d.shen,and r.rastogi,eds.),pp.785-794.
[0351]
[23]l.fahrmeir,et al.,statistics:the path to data analysis.springer textbook,berlin,heidelberg and s.l.:springer berlin heidelberg,fourth,improved edition ed.,2003.
[0352]
[24]kozachenko,lf,et al.,prob.peredachi informat.23(1987)9-16。
[0353]
[25]a.kraskov,et al.,phys.rev.e 69(2004)pt 2,p.066138.
[0354]
[26]bc ross,plos one 9(2014)e87357.
[0355]
[27]m.peleg,j.sci.food agric.71/2(1996)225-230.
[0356]
缩写列表
[0357]
ambr
ꢀꢀꢀꢀꢀ
自动化微型生物反应器
[0358]
atp
ꢀꢀꢀꢀꢀꢀ
腺苷三磷酸盐
[0359]
打包
ꢀꢀꢀꢀꢀ
自举聚合
[0360]
cho
ꢀꢀꢀꢀꢀꢀ
中国仓鼠卵巢
[0361]
cip
ꢀꢀꢀꢀꢀꢀ
原位清洗
[0362]
fda
ꢀꢀꢀꢀꢀꢀ
食品与药物管理局
[0363]
gmp
ꢀꢀꢀꢀꢀꢀ
药品生产质量管理规范
[0364]
ann
ꢀꢀꢀꢀꢀꢀ
人工神经网络
[0365]
mlp
ꢀꢀꢀꢀꢀꢀ
多级感知器
[0366]
nadph
ꢀꢀꢀꢀ
磷酸盐烟酰胺腺嘌呤二核苷酸磷酸盐
[0367]
our
ꢀꢀꢀꢀꢀꢀ
吸氧速率
[0368]
otr
ꢀꢀꢀꢀꢀꢀ
输氧速率
[0369]
pat
ꢀꢀꢀꢀꢀꢀ
过程分析技术
[0370]
rf
ꢀꢀꢀꢀꢀꢀꢀ
随机森林
[0371]
sip
ꢀꢀꢀꢀꢀꢀ
原位消毒
[0372]
vcd
ꢀꢀꢀꢀꢀꢀ
活细胞密度
[0373]
vcv
ꢀꢀꢀꢀꢀꢀ
活细胞体积
[0374]
xgboost
ꢀꢀ
极端梯度增强
[0375]
符号列表
[0376]
[0377]
[0378][0379]
材料
[0380]
软件:
[0381]
在spyder开发环境中,整个工作使用了编程语言python。实现是在面向对象的编程中进行的。编写了几个类,它们在项目中实现了个体任务。
[0382]
使用的软件
[0383][0384]
方法
[0385]
数据处理
[0386]
整个数据集包括155个培养运行。这些被分解为在线数据和离线数据。数据处理是用python编程语言中的spyder实现的。数据以csv文件形式提供。数据是用“csv”程序库读入的。这允许在开发环境内快速轻松地读取数据并转换为新的数据结构。已实现在线数据的“pifileparser”类和离线数据的“off-linedataparser”类。
[0387]
插值
[0388]
由于数据具有不同的数据密度,因此必须对其进行相应的内插。为此,使用了线性内插和使用移动平均法的内插。这两个函数都已通过“scipy”库实现:“线性插值interp1d”和“移动平均卷积”。这确保了内插值始终位于两个原始测得值之间。因此,插值总是在过程变量的测量信号的自然波动范围内。由于每个过程变量在一个文件中具有不同的时间戳,
因此需要创建另一个csv文件。“timelinemapping”含有各个培养的所有开始和结束时间,且由另一个数据库查询创建。选择了三个不同的间隔来解析数据:
[0389]
·
离线数据相关采样时间的时间戳
[0390]
·
1/10天
[0391]
·
五分钟
[0392]
由于相当低的数据密度和非线性数据过程,没有对离线数据应用线性插值。这里使用了三种不同的插值策略进行拟合:
[0393]
·
peleg拟合
[0394]
·
多项式拟合
[0395]
·
样条
[0396]
根据m.peleg的插值法能够通过附加函数项来绘制生物生长图,从而很好地描述生长过程[27]。因此,活细胞密度的原始数据符合所有三个插值。对于葡萄糖和乳酸,使用多项式和样条方法进行插值,因为这里不假设任何生物行为。在线数据集和离线数据集在不同的时间间隔内合并,并保存为每个培养的csv文件。然后基于这些数据集进行相关性分析。
[0397]
相关性分析
[0398]
相关性分析使用进行。使用可以对数据集进行统计分析。应用与各个目标变量(乳酸、葡萄糖、vcd、vcv)相关的在线数据(特征)的多变量统计。分析数据在描述目标变量时的统计显著性和线性关系。根据bravais-pearson,相关性分析以相关系数的形式显示自变量与因变量之间的线性关系。
[0399]
互信息
[0400]
已经以互信息的形式使用了另一种确定合适特征的方法。在通过互信息确定时,确定信息内容,信息内容包含在自变量x中以描述目标变量y。通过“互信息回归”用“sklearn”来计算和实现依赖关系。根据分辨率为五分钟的数据集的大小,分别计算每个培养的信息内容,然后形成所有培养获得的值的均值。
[0401]
创建特征矩阵/结果向量
[0402]
特征矩阵的创建来自于相关性分析和基于信息内容的统计评价的结果。这可以表示为一个矩阵,且每列含有一个特征和一个具有相应版本特征的时间点。特征矩阵被保存为panda dataframe。因此,可以使用合适的文件格式来训练和测试模型。
[0403]
建模和评价
[0404]
在相关性分析结果的帮助下,为每个目标变量创建了一个单独的数据集。将特征矩阵划分为训练数据集和测试数据集是训练模型所必需的。稍后用于在线预测需要保留完整的验证项目。训练数据集含有整个数据集的80%和因此123个培养运行。
[0405]
由于所有目标变量的预测是一个恒定的目标参数,因此仅使用回归量作为模型。许多因模型而异的超参数可用于模型。因此,模型的训练用于调整超参数,以便尽可能精确地映射目标变量。
[0406]
对于训练本身,整个特征矩阵使用scikit-learn库的标准缩放器进行标准化。
[0407]
超参数优化
[0408]
超参数使用来自scikit-learn库的随机搜索(randomizedsearchcv)和基于网格
的搜索(gridsearchcv)进行了优化。所有模型都使用scikit-learn库的随机搜索(randomizedsearchcv)结合训练数据集的十倍交叉验证进行训练。检查超参数的各个区域以获得最小的rmse。随机搜索进行了30次。因此,在每次迭代中使用不同的、随机选择的超参数集。输出rmse最小的十个模型的超参数。然后,基于来自随机搜索的超参数,对网格搜索的超参数进行更精细的分级。通过对数据集的十倍交叉验证再次进行网格搜索。保存误差最小(rmse最小)的模型,然后用于从测试数据集中估计目标变量。
[0409]
多层感知器
[0410]
scikit-learn库用于实现多层感知器(mlp)。以下列表含有用于训练模型的超参数:
[0411]
·
输入层的神经元数量
[0412]
·
隐藏层的神经元数量
[0413]
·
用于设置权重的求解器算法(adam、lbfgs、sgd)
[0414]
·
激活函数(identity、logistic、tanh、relu)
[0415]
·
学习速率
[0416]
·
最大迭代次数
[0417]
随机森林
[0418]
scikit-learn库也实现了随机森林。以下候选者可用作此优化内的超参数:
[0419]
·
决策树的数量
[0420]
·
每个决策树的特征数
[0421]
·
决策树的最大深度
[0422]
·
创建新节点的最小数据集数
[0423]
·
选择数据集的方法(自举=真/假)
[0424]
xgboost
[0425]
xgboost算法通过xgboost库集成到项目结构中。以下超参数空间对应于:
[0426]
·
集合中回归树的数量
[0427]
·
回归树的最大深度
[0428]
·
学习速率η
[0429]
·
每个决策树的数据集数量
[0430]
·
决策树中子节点的最小权重
[0431]
·
γ误差评价
[0432]
作为使用的超参数。
[0433]
模型评价
[0434]
模型评价主要通过显示误差直方图来实现。这显示了模型在为目标参数的实际值预测测试数据集时所具有的误差(残差)。
[0435]
计算目标参数估计准确度的rmse并与目标参数的平均值进行比较。
[0436]
为了检查模型的过拟合,计算了整个训练数据集和测试数据集的rmse。两个误差之间的差异被用作模型过拟合的指示:
[0437]
过拟合=rmse
测试-rmse
训练
[0438]
为了进一步描述模型质量,使用了整个测试数据集和单独考虑的每个培养的确定
系数。
[0439]
实例1
[0440]
ambr250-培养
[0441]
收集了基于ambr250系统中培养的155个数据集。所用的真核细胞是在细胞外表达靶分子的cho细胞。使用分批补料过程进行培养。使用的ambr系统使得能够同时进行十二次培养。主培养的培养时间为13至14天。一次性生物反应器(250ml)为此提供了反应空间。预培养在摇瓶中进行并持续三周。接种时的细胞体积和数量方面的起始条件在每个反应器中是可比的。所使用的培养基完全是化学成分确定的培养基。每次培养仅使用一批培养基。
[0442]
为了在该系统内提供最佳培养条件,可以使用许多过程变量。要控制的参数是培养基中的ph值、温度和溶解氧浓度。下表含有用于这项工作的所有过程变量的完整列表。
[0443]
表12:在线测得参数
[0444][0445][0446]
通过所谓的pi系统在整个培养期间记录所有测量变量。pi系统仅含有在线测量变量。
[0447]
此处列出的参数可用于监测最佳培养条件。bluesens的废气分析也可用于每个反应器。这可以检测来自生物反应器的废气流中的o2和co2含量,从而为过程控制提供另一个重要组成部分。废气流中的这两个测量变量可用于确定our和otr。
[0448]
在培养期间每天采样。然后使用cedex bio(roche diagnostics gmbh,mannheim,germany)分析代谢物的不同浓度和产物滴度。
[0449]
此外,进行细胞计数测量。测量提供有关活细胞密度、总细胞密度、活力、聚集率和细胞直径的信息。这些参数可用于推断培养物的生长行为。离线尺寸通过cedex(roche diagnostics gmbh,mannheim,germany)细胞计数器测量。这些细胞计数和细胞分析系统的误差在10%的范围内。所有使用的离线测量量如下表所示。
[0450]
表13:离线测量变量。
[0451]
再多了解一些
本文用于企业家、创业者技术爱好者查询,结果仅供参考。