一种基于多元化特征的基因序列分类方法及系统与流程
- 国知局
- 2024-07-12 10:17:21
本发明涉及生物信息处理,尤其涉及一种基于多元化特征的基因序列分类方法及系统。
背景技术:
1、基因序列分类技术,是生物信息学的一个重要分支,主要使用生物学实验或计算机等手段来分类dna序列上的具有生物学特征的片段。这项技术涉及到的关键步骤包括从大量数据中提取出有用的基因信息,然后根据这些信息进行分类和预测。基因序列分类的关键挑战之一是如何分类同一种微生物之间具有的相似性。同一种的微生物通常具有较高的相似性,这主要体现在它们的基因组成上。在生物信息学领域基因序列的分类主要通过比对的方式,相比于自然语言处理技术,缺少了通过了上下文信息提高分类精度和不断学习和优化等优点。
2、同一种微生物的基因也会出现差异性,这些差异性来源于多个因素,包括基因突变、染色体畸变等。基于pcr的16s核糖体rna测序,将相似度97%以上的视为可能是同一个物种,这种基于相似度的分类方式本身就具有一定的不准确性。除了基于比对的方法,常用的自然语言方法是将基因序列做词嵌入,根据词向量的相似性进行分类,词向量的结果更多地受到整个序列上下文的信息的影响,对某一片段特征提取的能力较弱。
技术实现思路
1、本发明提供一种基于多元化特征的基因序列分类方法及系统,解决的技术问题在于:现有的因序列分类方法的准确性不够高。
2、为解决以上技术问题,本发明提供一种基于多元化特征的基因序列分类方法,包括步骤:
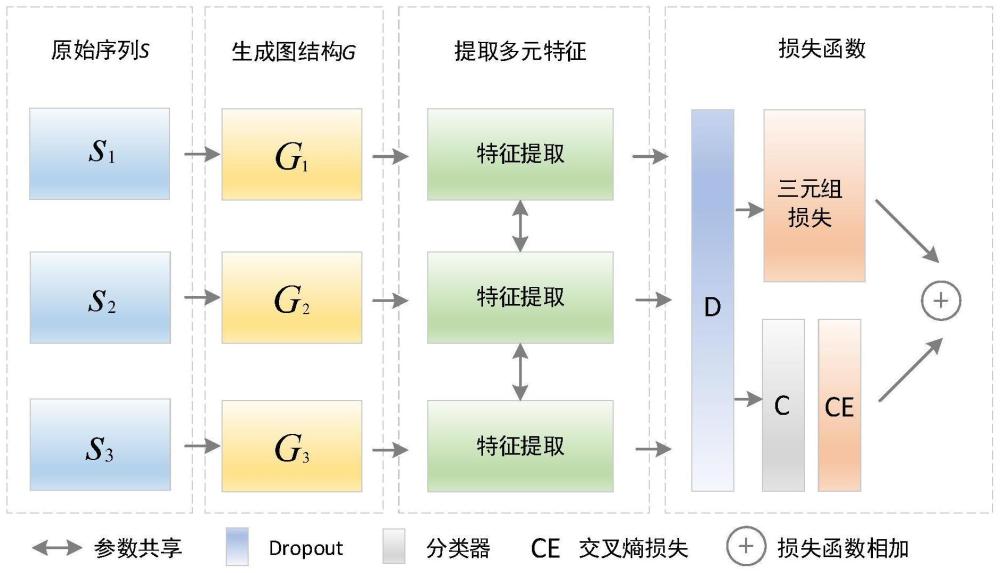
3、s1、获取基因序列s,该基因序列s中包含一个anchor样本s1(锚定样本)、一个positive样本s2(正样本)和一个negative样本s3(负样本);
4、s2、利用k-mer将样本s1、s2、s3各自分成多个大小为k的k-mer片段,k=3、4、5,则样本si的3-mer片段包括3-mersi1、3-mersi2至3-mersiai,样本si的4-mer片段包括4-mersi1、4-mersi2至4-mersibi,样本si的5-mer片段包括5-mersi1、5-mersi2至5-mersidi,ai、bi、di分别表示样本si的3-mer片段、4-mer片段、5-mer片段的数量,i=1,2,3;
5、s3、用节点表示k-mer片段,用边表示k-mer片段之间的重叠关系,构建样本si的3-mer片段、4-mer片段、5-mer片段对应的基因序列图gi3、gi4和gi5,则样本si的基因序列图gi包括gi3、gi4和gi5;
6、s4、将基因序列图g1、g2和g3输入训练好的多元化特征分类模型,得到所述基因序列s的分类结果。
7、进一步地,在所述步骤s4中,所述多元化特征分类模型的分类过程包括步骤:
8、s41、对基因序列图g1、g2和g3各采用一个特征提取模块进行多元特征提取且彼此共享特征提取参数;
9、s42、根据提取的基因序列图g1、g2和g3的多元特征进行分类,输出分类结果。
10、进一步地,在步骤s41中,对基因序列图进行多元特征提取的具体过程包括步骤:
11、s411、对基因序列图g1进行图自注意力提取,得到特征图g1,t;对特征图g1,t进行图自注意力提取,得到特征图g1,tt;
12、s412、对基因序列图g1进行图卷积,得到特征图g1,n;
13、s413、对基因序列图g1、特征图g1,tt和特征图g1,n进行特征融合,得到融合特征图g1,co;
14、s414、对融合特征图g1,co进行特征聚合,得到聚合特征图g1,d;
15、s415、对聚合特征图g1,d、特征图g1,n、特征图g1,t和特征图g1,tt进行特征拼接,得到最后的特征图g1,z;
16、s416、采用与步骤s411~s415相同的步骤对基因序列图g2和g3进行特征提取。
17、进一步地,在所述步骤s4中,训练所述多元化特征分类模型的过程为:
18、将预先完成分类的基因序列作为训练样本,构成训练样本集,其中三个样本为一组,由一个正样本、一个负样本以及一个锚定样本组成;网络构架使用孪生神经网络,该孪生神经网络有三个输入,即一个正样本、一个负样本以及一个锚定样本,每一个样本分别经过与所述步骤s2、s3、s411~s416相同的处理后,通过dropout层,得到各样本的特征向量h,并通过分类器进行分类计算交叉熵损失函数;同时计算三元组损失函数,让相同类别间的距离尽可能地小,不同类别间的距离尽可能大;将计算出的交叉熵损失函数与三元组损失函数进行相加作为损失函数对参数进行优化,且三个特征提取模块之间的参数共享。
19、进一步地,所述损失函数构建为:
20、ltotal=αlc+βlt
21、其中,ltotal表示联合损失,lc代表交叉熵损失,lt代表三元组损失,α、β分别表示交叉熵损失和三元组损失的权重系数。
22、进一步地,交叉熵损失lt定义为:
23、
24、其中,b表示一次性训练所选取的样本组数量,i表示第i个样本组,j表示每组样本中第j个数据,一组样本由三组数据组成故j的取值范围为1、2、3,δj为这三组数据的权重分配,δ1对应anchor样本、δ2对应positive样本、δ3对应negative样本,且δ1+δ2+δ3=1,hij为第i组第j个数据的特征输出值,n为输出节点的个数即分类的类别个数,bk分别表示一个组的第k个输出节点的权重和偏置,分别表示一个组中每个样本的权重和偏置,上标t表示矩阵转置;
25、三元组损失lt定义为:
26、lt=max(d(a,p)-d(a,n)+margin,0)
27、其中,d(·)计算两个样本之间的相似性,margin是一个大于0的常数,a为anchor样本,p为positive样本是与a同一类的样本,n为negative样本是与a不同类的样本,max(,)表示取较大值。
28、进一步地,在步骤s414中,特征聚合是指:
29、每一组输入的数据有三个样本,而每一个样本会被映射为三个图结构,其节点分别为按照3-mer、4-mer、5-mer分割后得到的片段,一个长度为4的片段对应着两个长度为3的片段,一个长度为5的片段对应两个长度为4的片段,将3-mer的特征向量按照该对应关系进行特征聚合给4-mer,同理将4-mer的特征向量聚合给5-mer;
30、将4-mer节点数据聚合到5-mer节点的公式为:
31、h′abcde=σ(αabcde′whabcd+αa′bcdewhbcde)
32、其中,hbcde表示在4-mer图结构数据中的节点,h′abcde表示在5-mer图结构数据中完成特征聚合的节点,w为共享参数对节点特征进行增维,αabcde′表示4-mer节点abcd与5-mer节点abcde的注意力系数,αa′bcde表示4-mer节点bcde与5-mer节点abcde的注意力系数,a、b、c、d、e每个字母代表一个碱基,a、b、c、d、e表示任意碱基a、t、g、c,σ(·)为激活函数;
33、将3-mer节点数据聚合到4-mer节点的原理与将4-mer节点数据聚合到5-mer节点相同。
34、进一步地,在所述步骤s3中,对于k-mer片段,若节点之间出现长度为k-1的重叠关系,即认为节点之间存在联系,用一条边将该两个节点进行连接。
35、本发明还提供一种基于多元化特征的基因序列分类系统,其关键在于:包括基因序列获取单元、密码子生成单元、序列图生成单元和分类单元;所述基因序列获取单元、所述密码子生成单元、所述序列图生成单元和所述分类单元分别用于执行上述方法中所述的步骤s1、s2、s3、s4。
36、本发明还提供一种计算机可读存储介质,其关键在于:其上存储有计算机程序,该程序被处理器执行时实现上述基于多元化特征的基因序列分类方法。
37、本发明提供的一种基于多元化特征的基因序列分类方法及系统,该方法先借助k-mer和图生成的方式将基因序列转换为图数据(基因序列图),并通过图的结构对基因序列进行多元化的特征提取,其中包括局部特征、全局特征、混合特征及聚合特征。在局部特征提取和混合特征提取中采用了图自注意力机制,将需要重点关注的局部信息分配了较高的权重,在全局特征提取中采用了图卷积特征提取,在该过程中可以捕捉到图像中的局部模式和结构,并将这些信息整合为全局特征。在聚合特征提取中采用了图间的消息传递,捕获不同层级的特征。本。本方法及系统在对序列的多元化特征提取时,使用了注意力机制,而在特征融合时使用了残差连接,保证了提取到基因序列中更加显著的特征同时避免了过平滑,同时使用孪生神经网络衡量输入样本的差异性,从而提高基因序列分类的准确度。
本文地址:https://www.jishuxx.com/zhuanli/20240615/85955.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表