基于多模型预测的抗乳腺癌候选药物的筛选方法及装置
- 国知局
- 2024-07-11 17:41:57
本发明属于医学数据挖掘的,具体涉及一种基于多模型预测的抗乳腺癌候选药物的筛选方法及装置。
背景技术:
1、乳腺癌是目前世界上最常见,致死率较高的癌症之一。乳腺癌的发展与雌激素受体密切相关,有研究发现,雌激素受体α亚型(estrogen receptors alpha,erα)在不超过10%的正常乳腺上皮细胞中表达,但大约在50%-80%的乳腺肿瘤细胞中表达;而对erα基因缺失小鼠的实验结果表明,erα确实在乳腺发育过程中扮演了十分重要的角色。目前,抗激素治疗常用于erα表达的乳腺癌患者,其通过调节雌激素受体活性来控制体内雌激素水平。因此,erα被认为是治疗乳腺癌的重要靶标,能够拮抗erα活性的化合物可能是治疗乳腺癌的候选药物。比如,临床治疗乳腺癌的经典药物他莫昔芬和雷诺昔芬就是erα拮抗剂。
2、同时,一个化合物想要成为候选药物,除了需要具备良好的生物活性(此处指抗乳腺癌活性)外,还需要在人体内具备良好的药代动力学性质和安全性,合称为admet(absorption吸收、distribution分布、metabolism代谢、excretion排泄、toxicity毒性)性质。其中,adme主要指化合物的药代动力学性质,描述了化合物在生物体内的浓度随时间变化的规律,t主要指化合物可能在人体内产生的毒副作用。一个化合物的活性再好,如果其admet性质不佳,比如很难被人体吸收,或者体内代谢速度太快,或者具有某种毒性,那么其仍然难以成为药物,因而还需要进行admet性质优化。
3、目前,在药物研发中,分析化合物分子的生物活性和admet性质费时且费力,为了节约时间和成本,通常采用建立化合物活性预测模型的方法来筛选潜在活性化合物。具体做法是:针对与疾病相关的某个靶标(此处为erα),收集一系列作用于该靶标的化合物及其生物活性数据,然后以一系列分子结构描述符作为自变量,化合物的生物活性值作为因变量,构建化合物的定量结构-活性关系(quantitative structure-activityrelationship,qsar)模型,然后使用该模型预测具有更好生物活性的新化合物分子,或者指导已有活性化合物的结构优化。当前国内外有诸多研究基于数据分析和深度学习来解决这个问题,包括zang q.等通过采用随机森林特征选择方法,提取了与erα拮抗剂活性最相关的结构特征,并运用svm结合rf算法,从大量描述符中筛选识别,构建qsar模型。shaoqichen等提出了用于协作药物发现的基于联邦学习的qsar建模原型平台,即fl-qsar,验证了应用水平联合学习(hfl)的可行性,hfl是最近开发的协作和隐私保护学习框架来执行qsar分析。shanshan hu等提出了一种基于深度学习的新方法,通过连接端到端编码器-解码器模型和卷积神经网络(cnn)架构来实现qsar预测。chang k.等通过特征筛选得到12个分子描述符,分别构建了基于bp神经网络、决策树和随机森林的复合erα生物活性定量预测模型,研究结果表明随机森林模型在预测erα生物活性方面更具优势,可用于预测具有更好生物活性的新化合物分子。
技术实现思路
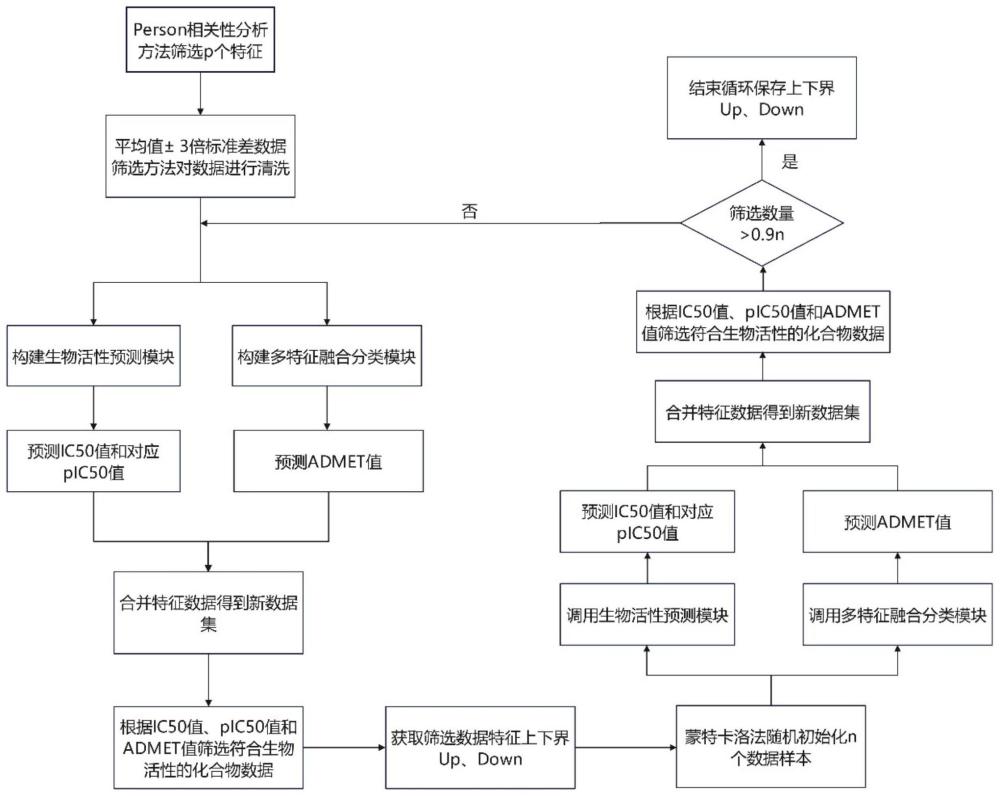
1、本发明的主要目的在于克服现有技术的缺点与不足,提供一种基于多模型预测的抗乳腺癌候选药物的筛选方法及装置,利用数据挖掘等技术从历史数据中深度提取关联性强的操作变量,构建抗乳腺癌候选药物筛选模型,结合蒙特卡洛法通过迭代缩小特征取值上下界的方法确定分子描述符特征的取值范围,从而为同时优化erα拮抗剂的生物活性和admet性质提供预测服务,能够在保证在使抑制erα具有更好的生物活性的同时,具有更好的admet性质,为抗乳腺癌药物研究和成分筛选提供导向作用,提高了化合物的应用价值。
2、为了达到上述目的,本发明采用以下技术方案:
3、第一方面,本发明提供了一种基于多模型预测的抗乳腺癌候选药物的筛选方法,包括下述步骤:
4、s1、对生物活性数据集进行数据分析和预处理,获取特征数据集;所述特征数据集包括分子描述符和化合物的smiles信息;
5、s2、构建抗乳腺癌候选药物筛选模型,抗乳腺癌候选药物筛选模型包括生物活性预测模块和多输入特征融合分类模块,用于获取生物活性值与化合物admet值;所述基因活性预测模块用于利用k-fold交叉验证方法拆分特征数据集,并基于拆分后的特征数据集利用lightgbm模型对抗乳腺癌候选药物筛选模型进行训练,完成生物活性值的预测;所述多输入特征融合分类模块用于利用bp神经网络和bi-lstm算法对特征数据集分别提取分子描述符和化合物的smiles信息中多维信息,将多维信息拼接融合后预测获得化合物admet值;
6、s3、将生物活性值和化合物admet值合并,获取新数据集;根据生物活性值和化合物admet值的筛选条件,选择符合生物活性的化合物数据集,得到有效数据;根据有效数据计算获取特征上下界;
7、s4、根据筛选特征上下界采用蒙特卡洛法随机初始化有效数据,继续利用抗乳腺癌候选药物筛选模型获取新的生物活性值与化合物admet值,重复步骤s2-s3,当筛选得到的样本数量达到指定条件后,迭代终止,保存最终的特征上下界。
8、作为优选的技术方案,所述数据分析和预处理,具体为:
9、利用pearson相关性分析方法对数据集中的分子描述符进行筛选,筛选出特征最显著的前p个分子{x1,x2,x3……xp},形成初始特征集;
10、计算初始特征集的平均值±3倍标准差,划定样本数据上下界;根据样本数据上下界,对初始特征集进行边界值处理,即将超出样本数据上下界的值替换成样本数据上下界的边界值,如下式:
11、
12、
13、
14、其中,表示初始特征集的样本整体平均值,xi表示初始特征集的样本,n表示样本数,sn表示初始特征集的样本标准差,boundary表示样本数据上下界的边界值。
15、作为优选的技术方案,所述利用k-fold交叉验证方法拆分特征数据集,具体为:利用k-fold交叉验证方法对特征数据集进行随机排序并将特征数据集划分成k组,将k-1组划分成训练集,剩余的划分成验证集。
16、作为优选的技术方案,还包括以下步骤:
17、构建两个对比模型,输入基于拆分后的特征数据集后对生物活性值进行预测,获取对比模型的预测值;对比模型包括基于模拟退火算法优化的bp神经网络预测模型和优化的深度学习bp神经网络预测模型;
18、根据预测值和实际值计算相关度r2,将计算结果保存;
19、将基因活性预测模块的预测值进行数据填充。
20、作为优选的技术方案,所述利用bp神经网络和bi-lstm算法对特征数据集分别提取多维信息,将多维信息拼接融合后预测获得化合物admet值,具体为:
21、利用bp神经网络提取分子描述符,预测获取分子描述符的n维信息;利用bi-lstm算法提取化合物的smiles信息,预测获取化合物的smiles信息的n维信息;
22、将分子描述符的n维信息和化合物的smiles信息的n维信息进行拼接融合,形成2n维数据;
23、将2n维数据输入预设的全连接层,获取化合物admet值;所述化合物admet值为5维向量,向量维度包括caco-2、cyp3a4、herg、hob和mn。
24、作为优选的技术方案,所述生物活性值和化合物admet值的筛选条件,具体为:生物活性值大于6.5,且化合物admet值大于2。
25、作为优选的技术方案,所述步骤s4,具体为:
26、利用蒙特卡洛法分别随机初始化k个p种特征数据作为样本;
27、重复步骤s2-s3,获取多个迭代的生物活性值和化合物admet值,根据生物活性值和化合物admet值的筛选条件筛选样本,当样本数量超过一定比例的总样本数量后,停止迭代并将此时的上下界作为p个特征的范围。
28、第二方面,本发明还提供了一种基于多模型预测的抗乳腺癌候选药物的筛选系统,应用于所述的基于多模型预测的抗乳腺癌候选药物的筛选方法,包括特征工程模块、第一处理模块、第二处理模块以及药物筛选模块;
29、特征工程模块,用于对生物活性数据集进行数据分析和预处理,获取特征数据集;所述特征数据集包括分子描述符和化合物的smiles信息;
30、第一处理模块,用于构建抗乳腺癌候选药物筛选模型,抗乳腺癌候选药物筛选模型包括生物活性预测模块和多输入特征融合分类模块,用于获取生物活性值与化合物admet值;所述基因活性预测模块用于利用k-fold交叉验证方法拆分特征数据集,并基于拆分后的特征数据集利用lightgbm模型对抗乳腺癌候选药物筛选模型进行训练,完成生物活性值的预测;所述多输入特征融合分类模块用于利用bp神经网络和bi-lstm算法对特征数据集分别提取分子描述符和化合物的smiles信息中多维信息,将多维信息拼接融合后预测获得化合物admet值;
31、第二处理模块,用于将生物活性值和化合物admet值合并,获取新数据集;根据生物活性值和化合物admet值的筛选条件,选择符合生物活性的化合物数据集,得到有效数据;根据有效数据计算获取特征上下界;
32、药物筛选模块,用于根据筛选特征上下界采用蒙特卡洛法随机初始化有效数据,继续利用抗乳腺癌候选药物筛选模型获取新的生物活性值与化合物admet值,重复运行第一处理模块和第二处理模块,当筛选得到的样本数量达到指定条件后,迭代终止,保存最终的特征上下界。
33、第三方面,本发明提供了一种电子设备,所述电子设备包括:
34、至少一个处理器;以及,
35、与所述至少一个处理器通信连接的存储器;其中,
36、所述存储器存储有可被所述至少一个处理器执行的计算机程序指令,所述计算机程序指令被所述至少一个处理器执行,以使所述至少一个处理器能够执行所述的基于多模型预测的抗乳腺癌候选药物的筛选方法。
37、第四方面,本发明提供了一种计算机可读存储介质,存储有程序,所述程序被处理器执行时,实现所述的基于多模型预测的抗乳腺癌候选药物的筛选方法。
38、本发明与现有技术相比,具有如下优点和有益效果:
39、(1)本发明通过基于pearson相关性分析方法和平均值±3倍标准差数据筛选方法完成对于样本数据有效特征的筛选方法,能够降低了数据的复杂度,提高特征筛选的效率,同时也保证了筛选特征的有效性,为后面模型预测的构建打下良好的基础。
40、(2)本发明通过提出基因活性预测模块,利用k-fold交叉验证方法拆分特征数据集,并基于拆分后的特征数据集利用lightgbm模型对抗乳腺癌候选药物筛选模型进行训练,完成生物活性值的预测,极大提升数据预测的准确率,具有很强的鲁棒性和抗干扰能力。
41、(3)本技术基于蒙特卡洛方法循环迭代的筛选方法,能够筛选出有效的化合物分子描述符,并预测其最优取值范围,在能够具备良好抗乳腺癌活性的同时还可以在人体内具备良好的药代动力学性质和安全性。
42、(4)本发明构建了化合物生物活性的定量预测模型和admet性质的分类预测模型,结合蒙特卡洛法通过迭代缩小特征取值上下界的方法确定分子描述符特征的取值范围,利用数据挖掘等技术从历史数据中深度提取关联性强的操作变量,同时兼顾数据的深度和广度。
本文地址:https://www.jishuxx.com/zhuanli/20240615/85682.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。