一种基于深度学习技术识别耐药致病微生物的方法与流程
- 国知局
- 2024-07-11 17:41:57
:本发明涉及微生物风险评估与预测,具体涉及一种基于深度学习技术模型判定与预测环境中耐药致病微生物的方法。
背景技术
0、背景技术:
1、长期以来,日益恶化的环境污染事件始终是国内外大多数城市安全的重要威胁。与此同时,耐药致病微生物已成为公共卫生的另一个重大关切。抗生素抗性使得人们在接受抗生素治疗过程中不得不加大剂量,但与此同时高剂量的抗生素会增加环境中的病原微生物耐药性,从而形成恶性循环,因此耐药致病微生物对人类社会产生了巨大的威胁。
2、受限于培养方法,通过传统的培养法获得的病原微生物类型有限,目前可培养微生物占全部微生物总量的不到1%,而qpcr技术不能实现高通量的耐药致病菌的检测,并且两种传统方式的检测结果均不具有时效性,进而导致难以全面正确的评估环境中耐药致病微生物的风险。近年来快速发展的宏基因组技术给全面识别耐药致病微生物提供了基础。
3、但是,现有技术中以往宏基因组技术识别耐药致病微生物的过程中难以判断水平基因转移机制产生的耐药致病菌。水平基因转移,是指生物将遗传物质传递给其他细胞而非其子代的过程,是自然界中抗生素抗性基因传播的重要机制,也是产生耐药致病菌的重要途径。质粒和噬菌体介导的水平基因转移是最主要的两种途径:质粒是细菌细胞内的小环状dna分子,它们可以携带一系列基因,包括耐药性基因。当细菌在水体中存在时,它们可以通过水平基因转移的方式将质粒传递给其他细菌,这种质粒介导的基因传递是耐药性基因在细菌群落中传播的一种重要途径。当环境中存在抗生素或其他选择性压力时,耐药性质粒的传播可能导致形成具有抗生素耐药性的新细菌群体。噬菌体是一种寄生于细菌的病毒,它们也可以介导基因传递。在水环境中,噬菌体可以感染细菌并将dna插入宿主细胞的基因组中。这导致了水平基因转移,其中包括耐药性基因的传递。这种现象尤其重要,因为它可以导致原本无害的细菌在感染后获得了新的耐药性,变成了耐药致病菌。
4、宏基因组技术,尤其是二代测序技术,虽然技术成本较低,应用度广,但产生的短序列难以判断质粒与噬菌体产生的耐药致病菌。为了应对这一挑战,本技术提出了一种基于深度学习技术的方法,用于判定耐药致病微生物风险,这一方法旨在通过宏基因技术,对耐药致病微生物进行识别。
5、与传统的数学模型不同,深度学习技术具有更高的灵活性,并且具有反馈与自学习能力,能够提供准确的评价与预测结果,使决策者能够快速制定科学的应急措施以减小突发事故对生态环境和生产生活的影响。这一技术将为水污染事件的应急决策提供新的可能性,有望大幅提高水源地和水体的安全性,保护公共卫生和生态环境免受微生物污染的威胁。
技术实现思路
0、技术实现要素:
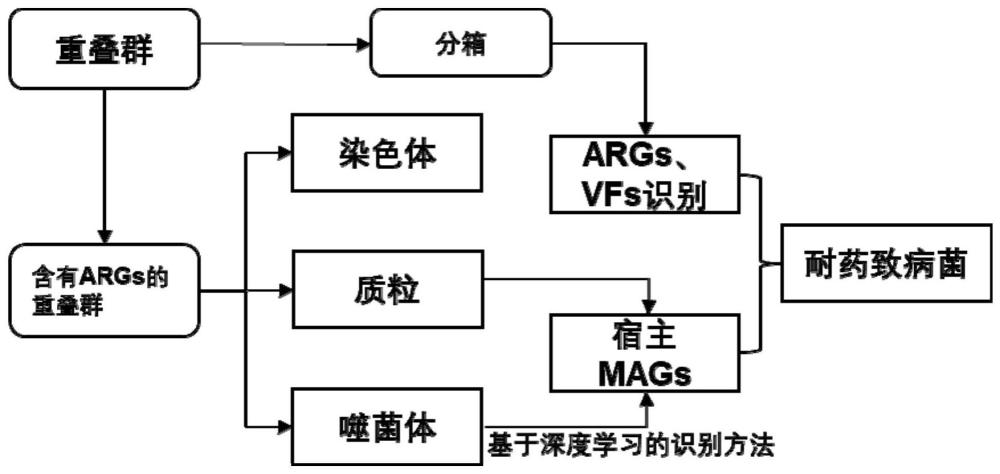
1、为了解决上述问题,本发明首先借助深度学习技术通过宏基因组识别由噬菌体、质粒通过水平基因转移产生的耐药致病菌,进而合并通过序列比对识别的带有抗生素抗性基因(args)和毒力因子(vfs)的mags,从而确定环境中所有的潜在耐药致病菌,为预防、防治生态环境的微生物污染问题提供基础。
2、本发明提供的技术方案之一,是一种基于深度学习技术模型,确定环境中耐药致病微生物的方法,包括以下步骤:
3、(1)环境样品采集:
4、采集待测环境水样,冷藏或冷冻保存,以防止微生物活动和dna降解;
5、(2)实验样本制备:
6、a.样品过滤:将采集的水样经0.22μm滤膜过滤,以去除悬浮颗粒物,保留微生物细胞;
7、b.dna提取:从滤膜截留的微生物样品中提取总dna;
8、(3)实验数据获取:
9、a.测序:对提取的总dna样品进行高通量测序;优选illumina测序平台;
10、b.序列质量控制:对原始测序数据raw reads进行质量过滤,得到高质量读段clean reads;
11、进一步地,所述质量过滤包括去除低质量序列、去除适当的引物和接头序列,以确保数据的准确性;优选利用ngs qc toolkit软件进行质量控制,获得宏基因组数据;
12、(4)对经过质量控制的宏基因组数据进行组装,获得重叠群(contigs);
13、进一步地,组装过程采用无参的组装方式(de novo),组装软件与策略选择包括根据图论或者机器学习的方法;优选利用megahit软件对所述宏基因组进行组装,获取重叠群(contigs);
14、进一步地,组装后的重叠群要求最小长度不得低于3000bp;
15、(5)筛选携带抗生素抗性基因的重叠群
16、使用prodigal对重叠群(contigs)进行基因预测,通过blastp将重叠区序列与最新版本的comprehensive antibiotic resistance database(card,v 3.2.6)中的抗生素抗性蛋白(args)进行比对,设置e值阈值为1e-10、70%的序列相似性阈值和50的比特分数阈值,即e值≤1e-10,序列相似性≥70%,比特分数≥50,从中筛选出携带抗生素抗性基因的重叠群(arg contigs);
17、(6)对噬菌体和质粒序列的识别:
18、分别采用基于深度学习的噬菌体鉴定模型phamer(https://github.com/kennthshang)和质粒鉴定模型plasflow对步骤(5)筛选出的携带抗生素抗性基因的重叠群(arg contigs)中的序列进行比对分析,识别其中的噬菌体和质粒序列;
19、进一步地,通过phamer鉴定的得分高于0.9的重叠群(contigs)被认为是噬菌体;
20、进一步地,plasflow是一种用于识别和分类质粒的计算工具,使用机器学习算法来检测和鉴定质粒序列;采用软件plasflow的默认参数对携带抗生素抗性基因的重叠群(arg contigs)中的质粒序列进行识别;
21、(7)对步骤(4)获得的重叠群(contigs)进行分箱(binning)操作,获得宏基因组组装基因组(mags);
22、具体地,利用metawrap等软件对步骤(4)获得的重叠群(contigs)序列进行分箱(binning)操作,获得宏基因组组装基因组(mags);
23、(8)通过序列比对识别带有抗生素抗性基因(args)和毒力因子(vfs)的mags,一方面本部分识别的耐药致病菌为全部耐药致病菌的一部分,另一方面为识别噬菌体和质粒提供宿主信息。具体步骤如下:
24、首先,使用prodigal对步骤(7)分箱获得的mags进行蛋白序列的识别,然后使用与步骤(5)相同的方法和标准将mags与最新版本的comprehensive antibiotic resistancedatabase(card,v 3.2.6)card数据库进行比对,以鉴定携带抗生素抗性基因(args)的微生物,即为耐药微生物(简写为arg-mags);
25、然后,同样blastp将步骤(7)分箱获得的mags序列与vfdb数据库中的毒力因子(vfs)进行比对,设置e值阈值为1e-10、70%的序列相似性阈值和50的比特分数阈值,即e值≤1e-10,序列相似性≥70%,比特分数≥50,从中筛选出携带毒力因子的微生物,即为致病微生物(简称为vf-mags);
26、最后,arg-mags与vf-mags的交集即为耐药致病菌,以下简称arg-vf-mags。
27、(9)将步骤(6)获得的质粒、噬菌体与步骤(8)识别出的vf-mags建立联系,以识别携带args的质粒和噬菌体的宿主微生物(mags),本部分识别的耐药致病菌即为水平基因转移机制产生的耐药致病菌;
28、a.模型构建与训练
29、首先,从ncbi下载已经完成测序的原核生物的质粒和噬菌体序列,并同时下载他们标注的宿主微生物序列,即获取经过验证过的、对应关系确定的质粒-原核生物和噬菌体-原核生物的基因组序列;利用prodigal对基因组中的功能蛋白序列进行识别,得到质粒、噬菌体和原核生物的蛋白序列文件;
30、然后,通过如下步骤构建图形卷积网络(graphical convolution network,gcn)模型:
31、(i)将噬菌体、质粒和原核生物作为gcn中的节点,并通过blastp工具获取来自质粒、噬菌体和原核生物的蛋白质序列、比对相似性,从而构建出质粒、噬菌体和原核生物的知识图谱,用于确定质粒与原核生物、噬菌体与原核生物之间的关联性,即宿主关系;
32、(ii)针对步骤(i)中构建的知识图谱,使用常规gcn结构模式对节点进行特征嵌入,gcn的特征向量来自质粒、病毒和原核生物的节点对应关系,激活函数为relu;
33、(iii)在完成gcn建模后,应用两层常规神经网络分类器来提取gcn的图特征,该特征即为质粒、噬菌体对宿主微生物的感染概率;
34、(iv)由于训练数据来源于ncbi数据库中下载的具有真实宿主关系的质粒、噬菌体和宿主原核生物的基因组数据,因此通过他们的真实对应关系对(iii)步骤获得的感染概率进行校正,计算差距,通过误差反向传递的方式反馈到模型网络中;
35、(v)通过adam梯度下降的方法对神经网络中的参数进行迭代,以使得步骤(iv)中的差距最小,以获得良好的训练结果。
36、b.模型应用于结果获取
37、将训练好的模型进行封装后,输入步骤(6)中识别出来的全部质粒、噬菌体和步骤(8)中获得的vf-mags序列文件,即可获得携带有args的质粒的宿主mags和携带有args的噬菌体的宿主mags,此即为通过水平基因转移机制产生的耐药菌;
38、(10)取步骤(8)识别出来的带有抗生素抗性基因(args)和毒力因子(vfs)的arg-vf-mags与步骤(9)识别出来的通过水平基因转移机制产生的耐药致病mags的合集,即为全部耐药致病微生物。
39、有益效果:
40、本发明全面的获取了环境宏基因样本中的耐药致病微生物分布特征。质粒、噬菌体等可移动遗传原件相关的抗生素抗性基因(args)在抗生素抗性研究中经常被忽视,这使得它们成为对公共卫生构成威胁的未监管的抗性因素。本技术方案能够(i)高准确度和召回率地识别携带args的噬菌体和质粒;(ii)识别噬菌体、质粒与宿主微生物组装基因组(mags)的关联,以预测潜在的抗生素耐药病原体;同时(iii)能描绘携带args的染色体、质粒和噬菌体的分布情况。
本文地址:https://www.jishuxx.com/zhuanli/20240615/85683.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表