基于多脑区多层次基因调控网络的疾病关联基因识别方法
- 国知局
- 2024-07-12 10:20:20
本公开实施例涉及基因分析,尤其涉及一种基于多脑区多层次基因调控网络的疾病关联基因识别方法。
背景技术:
1、脑疾病是一类由遗传、环境和生物学因素共同影响的复杂神经系统障碍疾病,其发病机理大部分仍不清晰。全基因组关联分析(genome-wide association study,gwas)虽能迅速定位与疾病相关的基因组区域,但由于大部分疾病相关位点位于非编码区,因此难以直接找出驱动疾病发展的具体基因。近年来,高通量测序技术揭示了非编码区变异在基因转录调控中的重要作用。在后gwas时代,许多生物信息学方法通过整合功能基因组学知识,旨在从gwas结果中识别遗传调控基因。分子数量性状位点(quantitative trait loci,qtl)数据是一种研究基因型变异与多组学分子表型(如基因表达水平、dna甲基化程度、蛋白质表达水平等)关联的数据类型。传统的基于统计模型的方法,如共定位、孟德尔随机化和全转录组关联研究,可以用于从qtl数据中鉴定潜在的遗传调控关系。然而,这些传统统计方法具有统计分布假设的局限性,且聚焦在基因组变异的局部区域,难以发现远端驱动基因。基于生物分子网络的疾病基因分析方法考虑了基因间的全局关系,因此成为了研究的热点。然而,如何在生物网络分析框架下整合多组学qtl数据以挖掘致病基因,仍是一个尚未被充分研究的问题。
2、疾病与基因的关联研究是最近几年生物信息学的热点之一,研究人员已经提出了许多疾病基因预测的方法。目前,计算疾病基因关联的方法可分为四类:基于统计模型的疾病基因预测方法、基于机器学习的疾病基因预测方法、基于网络扩散模型的疾病基因预测方法和基于图表示学习方法的疾病基因预测方法。
3、尽管已经取得了一系列成就,但在探索复杂疾病的新致病基因过程中,研究者需要考虑以下两个关键问题:(1)疾病通常是由多种因素共同作用的结果,单纯依赖对疾病基因的分析可能难以全面揭示其复杂性。生物网络的应用,通过考虑更广泛的生物学背景,例如基因间的相互作用、信号传导途径、蛋白质相互作用等能够帮助研究者更深入地理解疾病的整体机制。(2)传统的统计方法多采用线性模型,这在一定程度上限制了其对非线性生物关系建模的能力。生物系统中许多关系是非线性的,而图神经网络方法更适合处理这类复杂关系。此外,传统统计方法在特征提取方面相对受限,可能未能充分挖掘多层次、多尺度、多模态数据间的深层关联。
技术实现思路
1、为了避免现有技术的不足之处,本技术提供一种基于多脑区多层次基因调控网络的疾病关联基因识别方法,用以解决现有技术中存在难以全面揭示其复杂性,未能充分挖掘多层次、多尺度、多模态数据间的深层关联的问题。
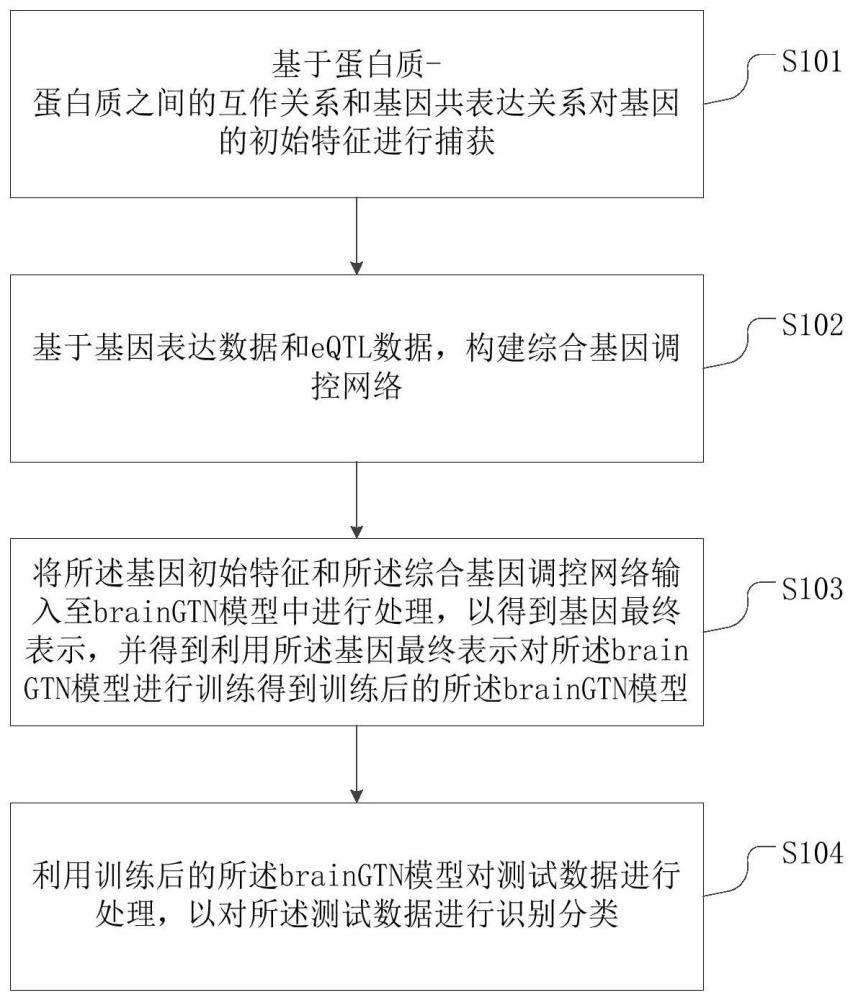
2、根据本公开实施例,提供一种基于多脑区多层次基因调控网络的疾病关联基因识别方法,该方法包括:
3、基于蛋白质-蛋白质之间的互作关系和基因共表达关系对基因的初始特征进行捕获;
4、基于基因表达数据和eqtl数据,构建综合基因调控网络;
5、将所述基因初始特征和所述综合基因调控网络输入至braingtn模型中进行处理,以得到基因最终表示,并得到利用所述基因最终表示对所述braingtn模型进行训练得到训练后的所述braingtn模型;
6、利用训练后的所述braingtn模型对测试数据进行处理,以对所述测试数据进行识别分类。
7、进一步的,所述对蛋白质-蛋白质之间的互作关系和基因共表达关系的基因初始特征进行捕获的步骤中,包括:
8、分别对所述蛋白质-蛋白质之间的互作关系和所述基因共表达关系进行降维处理,以得到基因相关联的ppi向量和基因的低维属性表示向量;
9、利用加权融合法将所述ppi向量和所述表达值向量进行融合,以得到基因向量;
10、将所述融合向量输入至若干个隐藏层的多层感知器中,以得到基因节点表示,并将来自最后一个隐藏层的基因的所述基因节点表示转换为概率向量,所述概率向量包含所有其他基因对的条件概率;
11、基于所述概率向量,利用softmax函数定义基因的邻域表示;
12、将所述邻域表示和所述基因节点表示相加,得到基因初始特征最终表示。
13、进一步的,所述基因相关联的ppi向量的表达式为:
14、(1)
15、其中,为属性变换层的权重矩阵,为基因所代表的蛋白质与其他蛋白质之间的交互关系的置信分数;
16、所述基因的表达值向量的表达式为:
17、(2)
18、其中,为基因的低维属性表示向量,为基因的表达值向量,为属性变换层的权重矩阵,为属性变换层的激活函数;
19、所述基因向量的表达式为:
20、(3)
21、其中,为基因表达信息相对于ppi信息的重要性系数;
22、所述基因节点表示的表达式为:
23、(4)
24、(5)
25、其中,为初始基因节点表示,为层0的激活函数,为层0的权重矩阵,用于变换输入特征,为层0的偏置项,为第k层的基因节点表示,表示经过 k层变换后的基因表示,为层 k的激活函数,为第 k层的权重矩阵,为第k层的偏置项;
26、所述概率向量的表达式为:
27、(6)
28、其中,为基因向量与基因向量相关的概率,为具有基因与所有其他基因相关的条件概率的输出概率向量;
29、基因向量与基因向量相关的概率的表达式为:
30、(7)
31、其中,是基因向量作为邻居的邻域表示,为基因向量作为邻居的邻域表示,为基因向量之外的其他基因的向量表示;
32、所述基因初始特征最终表示的表达式为:
33、(8)
34、其中,为基因节点表示,是基因向量作为邻居的邻域表示。
35、进一步的,所述基于基因表达数据和eqtl数据,构建综合基因调控网络的步骤中,包括:
36、基于aracne方法,利用基因表达数据推断基因之间的调控网络,构建第一多层基因调控网络;
37、根据eqtl数据,构建基于eqtl数据的第二多层基因调控网络;
38、将第一多层基因调控网络和第二多层基因调控网络进行整合,得到综合基因调控网络。
39、进一步的,所述将所述基因初始特征和所述综合基因调控网络输入至braingtn模型中进行处理,以得到基因最终表示的步骤中,包括:
40、所述braingtn模型中的图卷积神经网络将所述基因初始特征和所述综合基因调控网络进行融合以得到第一基因表示,且对所述第一基因表示进行线性投影和层归一化;
41、向序列末尾的所述第一基因表示附加一个可学习嵌入,并将嵌入后的结果输入至所述transformer层堆栈中进行分析,得到第二基因表示;
42、基于所述第二基因表示,利用线性投影并进行softmax操作以生成节点最终表示;
43、利用融合层组合不同维度的所述节点最终表示,以得到基因最终表示。
44、进一步的,所述第一基因表示的表达式为:
45、(9)
46、其中,为表示激活函数,为一个对角矩阵,其对角线元素为每个节点的度,归一化后表示为,为邻接矩阵a的归一化版本,为表示第l层的节点特征矩阵,为表示第1层到第1+1层的权重矩阵
47、线性投影和层归一化后所述第一基因表示的表达式为:
48、(10)
49、其中,为可学习的权矩阵,为transformer维数,为gcn最终嵌入的维数,为基因经过gcn层之后的表示;
50、所述第二基因表示的表达式为:
51、(11)
52、其中,,,分别为层中单个注意头学习到的查询矩阵、键矩阵和值矩阵,为经过transformer层后的第二基因表示,为第二基因表示附加一个可学习嵌入后的基因表示;
53、所述节点最终表示的表达式为:
54、(12)
55、其中,为可学习的权重
56、所述基因最终表示的表达式为:
57、(13)
58、其中,为共享注意力向量,为第维的权重,为节点进行softmax操作后的表示,为权重矩阵,为偏置量
59、进一步的,所述利用训练后的所述braingtn模型对测试数据进行处理,以对所述测试数据进行识别分类的步骤中,包括:
60、将所述测试数据输入至训练后的所述braingtn模型中,利用二元交叉熵作为损失函数对所述测试数据进行处理,以对所述测试数据进行分类。
61、进一步的,所述braingtn模型的损失函数的表达式为:
62、(14)
63、其中,为损失函数,为真实标签,为预测出的标签,为模型对应于第 i个样本的预测输出,n为类别的数量,数值为2。
64、本公开的实施例提供的技术方案可以包括以下有益效果:
65、本公开的实施例中,通过上述基于多脑区多层次基因调控网络的疾病关联基因识别方法,一方面,该方法融合了源自不同脑组织的多组学数据,其中包括基因表达量以及基因组学数据。另一方面,通过结合蛋白质相互作用关系和基因表达数据来学习网络中基因的初始特征表示。这种嵌入表示不仅保留了基因网络的结构特征,还综合了基因表达的统计关联。再一方面,利用图卷积神经网络和transformer技术,学习基因在局部和全局层面的相互关系。通过自注意力机制,该框架能够融合多层节点特征,从而得出每层的权重分布,进而提高致病基因的预测准确性。
本文地址:https://www.jishuxx.com/zhuanli/20240614/86300.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表