基于可变剪接百分比特征的致病基因预测方法
- 国知局
- 2024-07-12 10:16:43
本公开实施例涉及生物信息,尤其涉及一种基于可变剪接百分比特征的致病基因预测方法。
背景技术:
1、目前,致病基因预测模型用于预测疾病与基因是否存在关联,可以减少生物验证的成本。现有的致病基因预测模型大多利用多种生物数据,如蛋白质域、分子功能、生物过程、细胞成分等,并结合机器学习模型对基因进行分类。如gsi利用基因注释集构造信号矩阵并结合机器学习模型预测疾病基因;pudi收集不同生物数据并利用半监督框架进行致病基因预测;rwr利用种子基因在多生物源异构图上随机游走获取无标签样本的得分;huamnet利用不同的生物信息,构造了一个带权的基因网络。虽然这些方法取得了良好的性能,但都未考虑转录组可变剪接特征。
2、可见,亟需一种预测精准度高的基于可变剪接百分比特征的致病基因预测方法。
技术实现思路
1、有鉴于此,本公开实施例提供一种基于可变剪接百分比特征的致病基因预测方法,至少部分解决现有技术中存在预测精准度较差的问题。
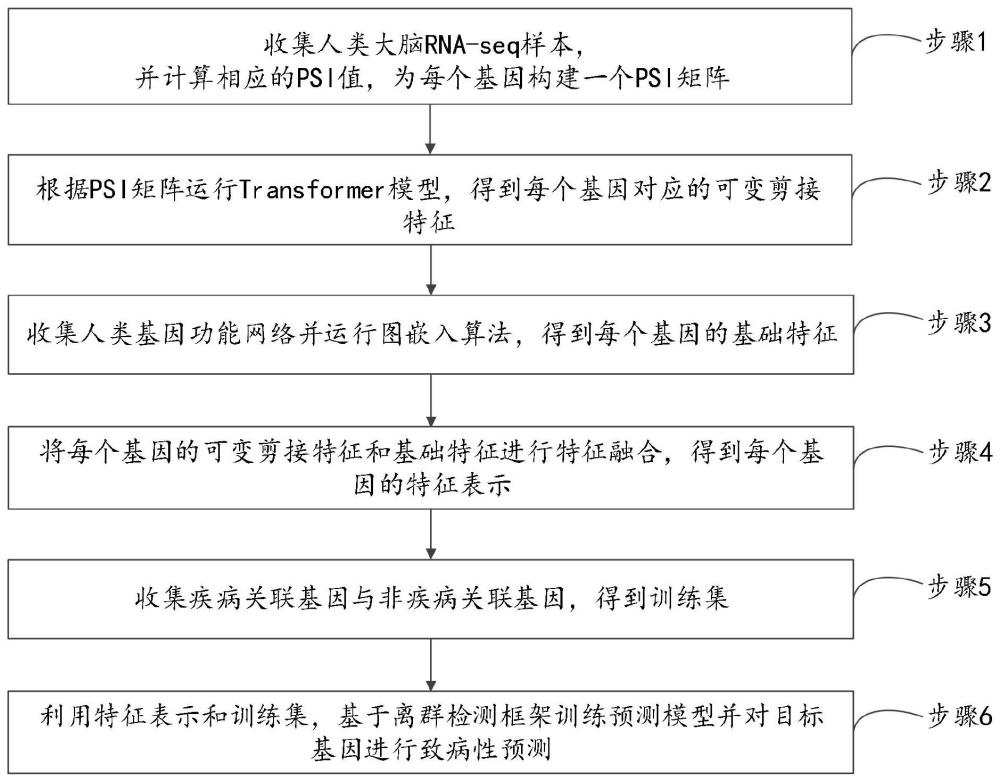
2、本公开实施例提供了一种基于可变剪接百分比特征的致病基因预测方法,包括:
3、步骤1,收集人类大脑rna-seq样本,并计算相应的psi值,为每个基因构建一个psi矩阵;
4、步骤2,根据psi矩阵运行transformer模型,得到每个基因对应的可变剪接特征;
5、步骤3,收集人类基因功能网络并运行图嵌入算法,得到每个基因的基础特征;
6、步骤4,将每个基因的可变剪接特征和基础特征进行特征融合,得到每个基因的特征表示;
7、步骤5,收集疾病关联基因与非疾病关联基因,得到训练集;
8、步骤6,利用特征表示和训练集,基于离群检测框架训练预测模型并对目标基因进行致病性预测。
9、根据本公开实施例的一种具体实现方式,所述步骤1具体包括:
10、步骤1.1,从样本数据库收集预设数量的人类大脑rna-seq样本;
11、步骤1.2,在注释数据库下载人类的基因注释文件作为计算psi矩阵的输入;
12、步骤1.3,利用psi计算方法以人类大脑rna-seq样本和基因注释文件作为输入,为每个基因得到一个psi矩阵m×ni,其中,m表示rna-seq样本数量,ni表示第i个基因对应的剪接事件个数。
13、根据本公开实施例的一种具体实现方式,所述步骤2具体包括:
14、步骤2.1,将每个基因的psi矩阵通过填充0,转为一个m×nmax矩阵,其中nmax为基因中的最大剪接特征数量,以及,将psi矩阵中的na值也填充为0;
15、步骤2.2,如果收集到其他组织rna-seq样本,则选择组织间差异最显著的剪接事件作为输入,以及,如nmax大于阈值,则选择fdr最小的前n个特征,为每个基因保留m×n的psi矩阵;
16、步骤2.3,利用transformer模型为每个基因得到一个剪接特征
17、根据本公开实施例的一种具体实现方式,所述步骤3具体包括:
18、步骤3.1,从humannet数据库中收集人类基因功能网络;
19、步骤3.2,基于人类基因功能网络运行node2vec算法,得到基因i的网络特征表示作为基础特征。
20、根据本公开实施例的一种具体实现方式,所述特征表示的计算公式为
21、
22、其中,h1(*)与h2(*)为两个不同的映射头,||为拼接函数,用于连接两个向量。
23、根据本公开实施例的一种具体实现方式,所述步骤6具体包括:
24、步骤6.1,根据预设的采样数量,对训练集中阳性基因集合p与无标签基因集合u进行可放回的采样,形成平衡训练集;
25、步骤6.2,基于mlp架构构建基因疾病关联评分模型;
26、步骤6.3,在基因疾病关联评分模型加入dropout层,利用截断技术和平衡训练集对基因疾病关联评分模型进行训练并计算其对应的损失函数;
27、步骤6.4,当损失函数符合预设条件时,结束训练得到预测模型并对目标基因进行致病性预测。
28、根据本公开实施例的一种具体实现方式,所述基因疾病关联评分模型的表达式为
29、s(xi)=w2σ(w1xi)
30、其中,w1为一个可训练矩阵,将xi映射为一个d维向量σ(*)为relu激活函数,w2为一个可训练d维向量,最终为每个基因得到一个疾病关联评分。
31、根据本公开实施例的一种具体实现方式,所述损失函数的表达式为
32、l=(1-y)|dev(x)|+ymax(0,a-dev(x))
33、其中,其中μ和σ为通过对一个正态分布随机采样计算得到均值与标准差。
34、本公开实施例中的基于可变剪接百分比特征的致病基因预测方案,包括:步骤1,收集人类大脑rna-seq样本,并计算相应的psi值,为每个基因构建一个psi矩阵;步骤2,根据psi矩阵运行transformer模型,得到每个基因对应的可变剪接特征;步骤3,收集人类基因功能网络并运行图嵌入算法,得到每个基因的基础特征;步骤4,将每个基因的可变剪接特征和基础特征进行特征融合,得到每个基因的特征表示;步骤5,收集疾病关联基因与非疾病关联基因,得到训练集;步骤6,利用特征表示和训练集,基于离群检测框架训练预测模型并对目标基因进行致病性预测。
35、本公开实施例的有益效果为:通过本公开的方案,利用transformer模型,得到每个基因的剪接特征,与其他常用的特征进行特征融合,得到了基因新的特征表示;并且,此发明在下游致病基因预测任务中,使用离群检测方法,为致病基因预测提供了新的算法框架。此发明所提出的psi特征提取方法,可以对任何rna-seq数据集使用,并适用于任何psi计算方法,并且可以与任何其他常用特征进行特征融合。通过对多个疾病数据集进行评估,所提出的方法可以提高现有的方法的准确性。总之,本发明所提供的方法,相对完善了转录组学在致病基因预测方向的不足,可以为其他方法提供新的组学特征,以提高致病基因预测的准确性。
技术特征:1.一种基于可变剪接百分比特征的致病基因预测方法,其特征在于,包括:
2.根据权利要求1所述的方法,其特征在于,所述步骤1具体包括:
3.根据权利要求2所述的方法,其特征在于,所述步骤2具体包括:
4.根据权利要求3所述的方法,其特征在于,所述步骤3具体包括:
5.根据权利要求4所述的方法,其特征在于,所述特征表示的计算公式为
6.根据权利要求5所述的方法,其特征在于,所述步骤6具体包括:
7.根据权利要求6所述的方法,其特征在于,所述基因疾病关联评分模型的表达式为
8.根据权利要求6所述的方法,其特征在于,所述损失函数的表达式为
技术总结本公开实施例中提供了一种基于可变剪接百分比特征的致病基因预测方法,属于生物信息技术领域,具体包括:收集人类大脑RNA‑seq样本,并计算相应的PS I值,为每个基因构建一个PS I矩阵;根据PS I矩阵运行Transformer模型,得到每个基因对应的可变剪接特征;收集人类基因功能网络并运行图嵌入算法,得到每个基因的基础特征;将每个基因的可变剪接特征和基础特征进行特征融合,得到每个基因的特征表示;收集疾病关联基因与非疾病关联基因,得到训练集;利用特征表示和训练集,基于离群检测框架训练预测模型并对目标基因进行致病性预测。通过本公开的方案,提高了预测精准度。技术研发人员:李洪东,张孝琪,刘锦受保护的技术使用者:中南大学技术研发日:技术公布日:2024/6/11本文地址:https://www.jishuxx.com/zhuanli/20240615/85877.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表