背景特异性腺嘌呤碱基编辑器及其用途的制作方法
- 国知局
- 2024-06-20 11:11:39
背景技术:
1、碱基编辑器能够实现靶向点突变在基因组dna中的精确安装,而不产生双链dna断裂(dsb)。腺嘌呤碱基编辑器(abe)将靶标a·t碱基对转换为g·c碱基对。由于g·c碱基对到a·t碱基对的突变是最常见的新生突变形式,因此abe有潜力纠正几乎一半的已知人类致病性点突变。原始腺嘌呤碱基编辑器abe7.10可以在dna中执行非常干净且高效的a·t至g·c转换,并且在培养的细胞、成年小鼠、植物和其他生物体中产生非常低水平的非期望副产物,例如小插入或缺失(indels)。参见2018年2月8日公开的国际公开号wo 2018/027078,2019年4月25日公开为wo 2019/079347的国际专利申请号pct/us2018/056146;koblan etal.,nat biotechnol 36,843-846(2018);以及gaudelli et al.,nature 551,464-471(2017)。
2、虽然腺嘌呤碱基编辑器(abe)原则上可以纠正最大类致病性点突变,但是可以观察到脱靶效应。特别是,经常观察到对附近非靶标腺苷的编辑——这种现象称为旁观者编辑。先前为尽量减少脱靶效应所做的努力涉及靶标腺苷附近的原间隔区相邻基序(pam)的特异性。本领域需要新的具有腺苷脱氨酶结构域的腺嘌呤碱基编辑器,该腺苷脱氨酶结构域对靶标腺苷的背景有偏好和/或特异性,例如关于紧邻该靶标腺苷的5’和/或3’的核苷酸身份的背景。
技术实现思路
0、发明概述
1、本公开提供了腺苷脱氨酶和包含这些腺苷脱氨酶的碱基编辑器,其对靶标腺苷具有背景偏好和/或背景特异性。因此,提供了背景特异性和背景偏好的腺苷脱氨酶变体和碱基编辑器。这些碱基编辑器可用于通过较少的旁观者编辑创建精确的碱基编辑,这对于治疗应用至关重要,因为任何旁观者编辑都可能导致靶向区域出现不期望的突变。本公开还提供了这些碱基编辑器和向导rna的复合物。本公开还提供了编码所公开的背景特异性和背景偏好的腺苷脱氨酶变体和碱基编辑器的多核苷酸和载体;含有这些脱氨酶变体、载体和/或碱基编辑器的药物组合物和细胞;以及含有这些脱氨酶变体、载体和/或碱基编辑器的试剂盒和组合物。本公开还提供了用任何这些碱基编辑器编辑靶核酸序列的方法,包括编辑具有对该靶标背景特异性的靶标的方法,例如编辑具有5’嘧啶背景(即,紧邻待编辑的腺嘌呤碱基5’的嘧啶)特异性的靶标。
2、本文提供了腺嘌呤碱基编辑器,其含有任何所述腺苷脱氨酶(例如,seq id no:1-6的脱氨酶)与核酸可编程dna结合蛋白结构域或napdnabp结构域的融合体。本文提供的腺嘌呤碱基编辑器(abe)可以能够维持dna编辑效率,并且在一些实施方案中表现出相对于现有腺嘌呤碱基编辑器例如abe7.10改进的dna编辑效率。在一些实施方案中,本文描述的abe表现出减少的旁观者编辑,同时保持高的中靶编辑效率。在一些实施方案中,本文描述的abe表现出接近于零的旁观者编辑频率。在一些实施方案中,本文提供的腺嘌呤碱基编辑器导致在dna底物中形成更少的插入缺失。
3、最近通过将腺苷脱氨酶融合到napdnabp结构域(例如,cas9结构域)而开发的腺嘌呤碱基编辑器使得能够使用特定靶标窗口内的腺嘌呤碱基编辑器向导rna(grna)靶向单核苷酸脱氨作用用于a:t至g:c的碱基对转换。最近开发了各种具有改进的dna编辑效率的工程碱基编辑器。参见komor,a.c.et al.,improved base excision repair inhibitionand bacteriophage mu gam protein yields c:g-to-t:a base editors with higherefficiency and product purity,sci adv 3(2017);rees,h.a.et al.,improving thedna specificity and applicability of base editing through protein engineeringand protein delivery,nat.commun.8,15790(2017);2018年3月15日公布的美国专利公开号2018/0073012;2017年5月4日公布的美国专利公开号2017/0121693;2017年4月27日公布的国际公开号wo 2017/070633;2015年6月18日公布的美国专利公开号2015/0166980;2017年12月12日发布的美国专利号9,840,699;2018年9月18日发布的美国专利号10,077,453;2020年3月6日提交的国际申请号pct/us2020/21362;2020年10月22日公布的国际公开号wo2020/214842;2019年11月15日提交的国际申请号pct/us2019/61685,于2020年5月22日公布为wo 2020/102659;以及2020年11月25日提交的国际申请号pct/us2020/624628,其中的每一个以其整体并入本文。碱基编辑器(be)通常是cas(“crispr相关”)结构域和核碱基(或“碱基”)修饰结构域(例如,天然或进化的脱氨酶,如腺苷脱氨酶结构域)的融合体。在一些情况下,碱基编辑器还可以包括改变细胞dna修复过程的蛋白质或结构域,以提高所产生的单核苷酸变化的效率、并入和/或稳定性。
4、迄今为止报道的碱基编辑器可能含有催化受损的cas9结构域,例如与核碱基(或“碱基”)修饰结构域融合的cas9切口酶结构域。abe对于致病等位基因的研究和校正特别有用,因为原则上近一半的致病点突变可以通过将a·t碱基对转换为g·c碱基对来校正4,5。迄今为止报道的许多abe包括含有野生型大肠杆菌tada单体(在碱基编辑过程中发挥结构作用)和可催化脱氧腺苷脱氨作用的进化的大肠杆菌tada单体(tada*)的异二聚体,以及cas9(d10a)切口酶结构域的融合蛋白。野生型大肠杆菌tada作为同二聚体,使位于trna反密码子环中的腺苷脱氨,生成肌苷(i)。尽管早期的abe变体需要含有n末端野生型tada单体的异二聚体tada才能获得最大活性2,但joung等人发现,后来的abe变体在有和没有野生型tada单体的情况下具有相当的活性42。
5、最先进的abe是abe7.10,其在2018年8月2日公布的国际公开号wo 2018/027078中公开。最近生成的abe是abe8e,其包含含有称为tada8e的单一脱氨酶变体的腺苷脱氨酶结构域,如2021年8月12日公布的国际公开号wo 2021/158921中所描述的。tada8e相对于tada7.10(abe7.10的腺苷脱氨酶)含有九个突变。tada7.10也是abemax的脱氨酶结构域,abemax是abe7.10的变体,其已进行了密码子优化用于在人类细胞中的表达。
6、本公开至少部分基于现有腺苷脱氨酶tada8e的进化,使用负选择和正选择来选择对位于紧邻靶标腺苷5’的嘧啶(即,胞嘧啶(c)、胸腺嘧啶(t)或尿嘧啶(u))有偏好的脱氨酶。本公开至少部分基于通过现有腺苷脱氨酶tada8e的噬菌体辅助方法进行的进化,使用负选择和正选择来选择对位于紧邻靶标腺苷5’的嘌呤(即,腺嘌呤(a)、或鸟嘌呤(g))有偏好的脱氨酶。这些腺苷脱氨酶在靶序列中诱导较少的旁观者编辑。在一些实施方案中,很少甚至不生成旁观者编辑。除了表现出较低的旁观者编辑以及因此较高的产物纯度之外,所公开的碱基编辑器还可以提供改进的靶向范围和效率。如本文所用,术语“旁观者编辑”是指靠近(接近)靶碱基的核碱基处的同义脱靶点突变,其不改变预期编辑方法的结果(例如,因为它们不改变编码的氨基酸)。旁观者编辑涵盖近似的沉默突变。
7、abe7.10碱基编辑器的腺苷脱氨酶结构域是tada7.10(或tada*),其是先前从大肠杆菌trna腺苷脱氨酶(ectada或tada)进化而来的脱氧腺苷脱氨酶,作用于单链dna2。tada7.10在ectada中包含以下取代:w23r、h36l、p48a、r51l、l84f、a106v、d108n、h123y、s146c、d147y、r152p、e155v、i156f和k157n。本文公开的进化实验的底物是tada-8e,其相对于tada7.10含有以下突变:a109s、t111r、d119n、h122n、y147d、f149y、t166i和d167n。噬菌体辅助进化实验方法的公开参见国际公开号wo 2018/027078;2019年4月25日公布的国际公开号wo 2019/079347;2019年11月28日公布的国际公开号wo 2019/226593;2018年3月15日公布的美国专利公开号2018/0073012,其于2018年10月30日作为美国专利号10,113,163发布;2017年5月4日公布的美国专利公开号2017/0121693,其于2019年1月1日作为美国专利号10,167,457发布;2020年10月22日公布的国际公开号wo 2020/214842,以及2020年5月20日提交的国际专利申请号pct/us2020/033873,2020年11月26日公布的国际公开号wo2020/236982,以及国际公开号wo 2021/158921,其中每一个的内容通过引用整体并入本文。
8、开发了噬菌体辅助连续进化(pace)abe选择系统,结合噬菌体辅助非连续进化(pance)选择系统,并将其应用于tada-8e以选择对具有位于紧邻靶标腺苷5’的嘧啶的靶标腺苷具有增强的特异性的变体。从这些实验演变而来的变体表现出比tada-8e更低的旁观者编辑,例如附近、脱靶腺苷的编辑。例如,在示例性序列gaaga5cca8aggatagactgctgg(seqid no:32)中,嘧啶背景特异性碱基编辑器以比紧邻在鸟嘌呤(其为一种嘌呤)之后的a5腺苷高得多的频率编辑紧邻在胞嘧啶之后的a8腺苷。
9、tad6是从这些pace和pance实验中出现的示例性变体,相对于tada-8e含有四(4)个额外的取代。tada-8e相对于tada7.10序列的突变保留在从这些pance实验中选择的变体中。tad6中相对于seq id no:315的tada7.10序列的这四个新突变为r26g、h52y、r74g和n127d。因此,tad6相对于seq id no:315的tada7.10序列包含r26g、h52y、r74g、a109s、t111r、d119n、h122n、n127d、y147d、f149y、t166i和d167n取代。tad6的氨基酸序列如seq idno:5所示。
10、示例性嘧啶背景特异性碱基编辑器abe-tad6表现出降低的旁观者编辑效果,例如,对于一些哺乳动物靶序列,旁观者编辑频率接近于零。含有tad6脱氨酶变体的abe-tad6,相对于abe7.10和abe8e也表现出更高的产品纯度。该碱基编辑器表现出了更高的产物纯度,同时保持了abe7.10的编辑效率。例如,使用abe-tad6证明产品纯度在60%至80%之间。
11、因此,在一些方面,本公开提供了具有嘧啶(“y”)背景特异性的腺苷脱氨酶,其中“背景”是指紧邻待编辑的腺嘌呤碱基(或靶标腺嘌呤碱基)的5’处的嘧啶或嘌呤的存在。这些脱氨酶可以偏好使靶核酸序列5’-yan-3’中的腺苷脱氨,其中y是c或t;n是a、t、c、g或u;a是靶标腺苷。在一些实施方案中,提供了具有背景特异性的腺苷脱氨酶,用于使5’-yan-3’的靶核酸序列中的腺苷脱氨,其中y是c或t,n是a、t、c、g或u;a是靶标腺苷。如本文所用,“偏好”、“背景偏好”和“背景偏好的”是指相对于靶标腺苷的产物纯度高于40%。如本文所用,“背景特异性”和“背景特异性的”是指相对于靶标腺苷的产物纯度高于55%。在一些实施方案中,表现出超过60%、65%、70%或大于70%的产物纯度。
12、因此,在一些方面,提供了腺苷脱氨酶,其在残基t111、d119、f149、v88、a109、h122、t166和d167处包含突变,并且还包含至少一个、至少两个或至少三个在选自seq idno:315的氨基酸序列中的r26、r74、h52和n127的残基处的突变,或另一种腺苷脱氨酶中的相应突变。在一些实施方案中,相应的突变是seq id no:316-325、433、434、448和449中的任一腺苷脱氨酶中的相应突变,其对应于衍生自大肠杆菌以外的物种的tada脱氨酶。脱氨酶还可包含至少一个选自v82、m94和q154的突变。在一些实施方案中,腺苷脱氨酶在残基r26、h52、r74和n127处包含突变。
13、在具有嘧啶背景偏好或特异性的腺苷脱氨酶中,本文提供了包含t111r、d119n、f149y、r26c、v88a、a109s、h122n、t166i和d167n取代的腺苷脱氨酶,并且还包含至少一个、至少两个或至少三个选自seq id no:315的氨基酸序列中的r26g、h52y、r74g和n127d的取代,或另一种腺苷脱氨酶中的相应取代。在一些实施方案中,相应的突变是seq id no:316-325、433、434、448和449中的任一腺苷脱氨酶中的相应突变。腺苷脱氨酶还可包含至少一个选自v82s、m94i和q154r的取代。腺苷脱氨酶还可包含r26g、h52y、r74g和n127d取代。在一些实施方案中,脱氨酶包含seq id no:5(tad6)的序列。在一些实施方案中,脱氨酶包含seqid no:6(tad6-sr)的序列。在一些实施方案中,脱氨酶包含seq id no:1(tad1)的序列。
14、在一些方面,本公开提供了具有嘌呤(“r”)背景特异性的腺苷脱氨酶。这些脱氨酶可以是偏好使5’-ran-3’的靶核酸序列中的腺苷脱氨的腺苷脱氨酶,其中r是a或g;n是a、t、c、g或u;a是靶标腺苷。提供了对5’-ran-3’的靶核酸序列中的腺苷进行特异性脱氨的腺苷脱氨酶,其中r是a或g,n是a、t、c、g或u;a是靶标腺苷。
15、因此,开发了噬菌体辅助连续进化(pace)abe选择系统,并将其应用于tada-8e以选择对具有位于紧邻靶标腺苷5’的嘌呤的靶标腺苷具有增强的特异性的变体。该pace系统在许多方面与上述嘧啶特异性pace系统相反。也就是说,负选择臂(质粒)和正选择臂(质粒)的组分已被交换,使得5’-嘌呤背景在连续的进化轮中被选择。换句话说,5’-嘌呤位于正选择质粒上,5’-嘧啶位于负选择质粒上。
16、从这些实验进化而来的变体可能表现出比tada-8e更低的旁观者编辑,例如附近、脱靶腺苷的编辑。例如,在示例性序列gaaga5cca8aggatagactgctgg(seq id no:32)中,嘌呤背景特异性碱基编辑器以比紧邻在胞嘧啶(其为一种嘧啶)之后的a8腺苷高得多的频率编辑紧邻在鸟嘌呤之后的a5腺苷。
17、表现出5’-嘧啶背景偏好的示例性腺苷脱氨酶包含相对于seq id no:315的r26g、h52y和n127d取代。腺苷脱氨酶可以包含r74g取代。脱氨酶还可以包含m94i取代。
18、在一些实施方案中,本公开的5’-嘧啶偏好脱氨酶还可以包含至少一个选自v82s和q154r的取代。在一些实施方案中,腺苷脱氨酶包含seq id no:315中的r26g、h52y、r74g、v82s、n127d和q154r取代。在一些实施方案中,腺苷脱氨酶包含seq id no:33、316-325、433、434、448和449中的任一腺苷脱氨酶中的相应突变。在一些实施方案中,脱氨酶包含seqid no:6(tad6-sr)的序列。在一些实施方案中,腺苷脱氨酶包含与seq id no:1-6中任一个具有至少90%、至少92.5%、至少95%、至少98%或至少99%序列同一性的氨基酸序列。在一些实施方案中,腺苷脱氨酶包含seq id no:1、2、3、4、5和6中任一个的氨基酸序列。在一些实施方案中,腺苷脱氨酶包含seq id no:1、5或6的氨基酸序列。
19、在一些方面,本公开提供了包含如本文所述的腺嘌呤碱基编辑器和一种或多种向导rna(例如,单向导rna(“sgrna”))的复合物,以及包含这些复合物的组合物。此外,本公开提供了编码和/或表达本文所述的腺嘌呤碱基编辑器的核酸分子,以及用于表达本文所述的腺嘌呤碱基编辑器和grna的表达载体或构建体,包含所述核酸分子的宿主细胞和用于递送和/或施用本文所述的基于核酸的实施方案的表达载体、和一种或多种grna以及组合物。
20、本公开还提供了复合物,其包含本文所述的腺嘌呤碱基编辑器和与碱基编辑器的napdnabp结构域(例如,cas9结构域)缔合的grna,例如单向导rna。向导rna的长度可以是15-100个核苷酸,并且包含与靶核苷酸序列互补的至少10个、至少15个或至少20个连续核苷酸的序列。
21、本文提供了编码任何所公开的腺苷脱氨酶(或腺嘌呤脱氨酶)和腺嘌呤碱基编辑器的多核苷酸和载体。应当理解,任何融合蛋白,例如本文描述的任何腺嘌呤碱基编辑器,可以以任何合适的方式稳定地或瞬时地引入细胞中。在一些实施方案中,腺嘌呤碱基编辑器可转染至细胞中。在一些实施方案中,可以用编码碱基编辑器的核酸构建体转导或转染细胞。例如,可以用编码碱基编辑器的核酸或翻译的碱基编辑器转导(例如,用编码碱基编辑器的病毒)细胞。作为另外的实例,细胞可以用编码碱基编辑器的核酸或翻译的碱基编辑器(例如,用编码碱基编辑器的质粒)转染。此类转导或转染可以是稳定的或瞬时的。在一些实施方案中,表达碱基编辑器或含有碱基编辑器的细胞可以例如用一种或多种grna分子转导或转染。在一些实施方案中,表达碱基编辑器的质粒可以通过电穿孔(例如,使用atxmaxcyte电穿孔器)、瞬时转染(例如,脂质转染)、稳定基因组整合(例如,转座因子)、病毒转导或其他本领域技术人员已知的方法引入细胞中。
22、还提供了用本文描述的腺嘌呤碱基编辑器编辑靶核酸分子,例如基因组内的单个核碱基的方法。与先前编辑核酸(例如dna)的方法相比,所公开的方法可以表现出减少的旁观者编辑。
23、在某些实施方案中,本文描述的编辑方法导致切割(或切口产生)双链dna的一条链,例如,包含靶标t:a核碱基对的腺嘌呤(a)的链,该靶标t:a核碱基对与包含被切除的靶标胸腺嘧啶(t)的链是相对的。这种切口产生结果用于将错配修复机制直接引导至未编辑的链,确保修饰的核苷酸不会被细胞机制理解为损伤。该切口可以通过在碱基编辑器中使用切口酶napdnabp结构域来创建。
24、在其他方面,本公开提供了用于用编码碱基编辑器和grna的表达构建体表达和/或转导宿主细胞的试剂盒。其进一步提供了用于将表达的腺嘌呤碱基编辑器和表达的grna分子施用至宿主细胞(诸如哺乳动物细胞,例如人类细胞)的试剂盒。本公开还提供了稳定或瞬时表达腺嘌呤碱基编辑器和grna或其复合物的细胞。本公开还提供了包含编码本文所述的任何腺嘌呤碱基编辑器的载体的细胞。
25、在一些实施方案中,提供了使用本文所述的腺嘌呤碱基编辑器(例如,abe-tad6)的治疗方法。本文所述的方法可以包括治疗患有与g:c至a:t点突变相关的疾病、病症或病况或处于与g:c至a:t点突变相关的疾病、病症或病况发展的风险中的受试者,包括向受试者施用腺嘌呤碱基编辑器或含有本文所述的碱基编辑器和向导rna的复合物、本文所述的多核苷酸、本文所述的载体、或本文所述的药物组合物。在一些实施方案中,提供了使用本文所述的腺嘌呤碱基编辑器治疗疾病、病症或病况例如血红蛋白病的方法。
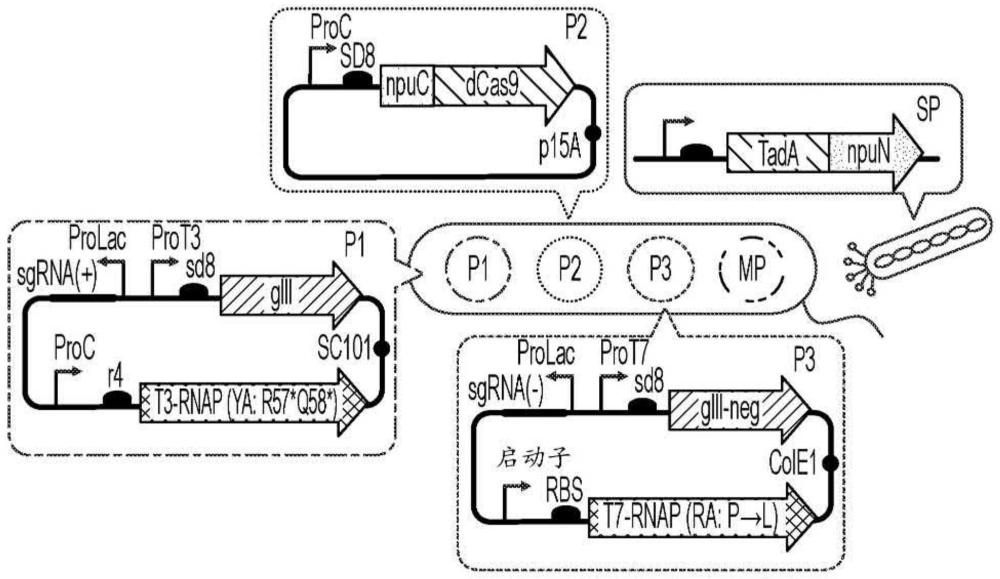
26、本公开提供了新的噬菌体辅助连续进化(pace)abe选择系统。因此,在一些方面,本公开提供了用于进行碱基编辑器的一个或多个结构域(例如,腺苷脱氨酶结构域)的定向进化以工程化任何所公开的腺嘌呤碱基编辑器的载体系统。在一些实施方案中,所公开的pace载体系统包含选择质粒,所述选择质粒包含编码碱基编辑器的表达构建体,所述碱基编辑器包含腺苷脱氨酶蛋白和编码分裂内含肽(例如,npu分裂内含肽)的n端和c端部分的序列,和三个辅助质粒。所公开的pace载体系统可以包含两个应用选择压力的辅助质粒,即设计用于正选择的第一质粒和设计用于负选择的第二质粒。
27、本公开的示例性pace载体系统包含一种或多种辅助质粒,其利用m13噬菌体基因iii来实现噬菌体繁殖的严格性。该基因编码能使噬菌体成功繁殖的必需外壳蛋白。m13噬菌体基因iii阴性也编码外壳蛋白,但基因iii阴性蛋白的并入使噬菌体无法感染后续细菌宿主。
28、在一些实施方案中,除了选择质粒之外,pace载体系统还包含一个或多个辅助质粒。在一些实施方案中,所述一个或多个辅助质粒包含(1)包含表达构建体的第一辅助质粒,所述表达构建体包含(i)编码由t3 rna启动子可操作控制的m13噬菌体基因iii(giii)肽的序列,和(ii)编码t3 rna聚合酶(rnap)的序列,其中编码rna聚合酶的序列含有包含一个或多个失活突变的第一区域;(2)编码分裂内含肽的c末端部分的第二辅助质粒和编码napdnabp(例如cas9蛋白)的序列;(3)包含表达构建体的第三辅助质粒,所述表达构建体包含(i)编码由t7rna启动子可操作控制的m13噬菌体基因iii阴性(giii-neg)肽的序列,和(ii)编码t7 rna聚合酶的包含含有一个或多个失活突变的第二区域的序列,其中失活突变可以在成功的碱基编辑后被校正。在一些实施方案中,cas9蛋白是dcas9蛋白。在一些实施方案中,cas9蛋白是cas9切口酶(ncas9)蛋白。
29、本文阐述了本发明的一个或多个实施方案的细节。本发明的其他特征、目的和优点将从详细描述、实施例、附图和权利要求中变得显而易见。本技术中引用的参考文献通过引用其整体并入本文。
本文地址:https://www.jishuxx.com/zhuanli/20240619/1171.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。