基于深度学习异构网络的药物不良反应预测方法及系统
- 国知局
- 2024-08-08 16:52:52
本发明涉及计算机生物信息网络嵌入和机器学习,具体涉及一种基于深度学习异构网络的药物不良反应预测方法及系统。
背景技术:
1、发生在药品上市后的不良反应已经成为一个重要的全球性的健康问题。广义的药物不良反应包括因药品质量问题或用药不当所引起的有害反应,包括药物的副作用、毒性作用(毒性反应)、后遗反应(后作用)、过敏反应、特异质反应、抗感染药物引起的二重感染、依赖性以及致癌、致畸、致突变作用等。
2、随着信息技术的快速发展,使用机器学习和深度学习技术在药物研发阶段预测潜在的药物不良反应为生物技术提供了巨大的助力。虽然在过去的一段时间里,已经提出了很多使用机器学习或深度学习技术预测药物潜在不良反应的方法,尤其是基于深度学习模型用以预测药物潜在不良反应的方法,但这些方法都普遍存在预测的不良反应相对固定和准确率不高的问题。
3、目前,大部分提出的预测药物不良反应的基于深度学习模型的预测方法直接使用分子指纹作为药物分子的特征或者使用基于分子描述符提取分子特征的方法来提取药物分子特征。
4、如:
5、中国专利文献cn 114429796a公开一种基于多核学习的药物不良反应预测方法,该方法使用药物分子的pubchem分子指纹作为分子特征用于下游任务。pubchem分子指纹使用一个881维的向量来表示分子是否含有881种化学亚结构,这种方法只能标识分子是否含有某种亚结构,而无法准确的描述分子的结构特征和理化性质,阻碍了预测性能的提升。
6、lee等人于2021年提出的文章:《descriptive prediction ofdrug side-effectsusing a hybrid deep learning model》[j].international journal ofintelligentsystems》[j].lee c y,chenyp
7、p.international journal ofintelligent systems.2021.2491-2510.,将药物分子转化为一张2d图片,对该图片做卷积操作来提取药物分子特征,这种方法在一定程度上可以描述分子的结构特征,但是依然无法描述分子的理化性质,模型准确率等性能指标有待进一步提升。
8、此外,还有现有技术提出的预测药物不良反应的基于深度学习模型的预测方法大都采用多标签分类的方法,如:
9、joshi p等人于2022年提出的文章:《aknowledge graph embeddingbasedapproach to predict the adverse drug reactions using a deep neural network》[j].joshi p,masilamani v,mukherjeea.journal ofbiomedicalinformatics.2022.132:104122.;以及中国专利文献cn114743692a公开的一种基于知识图谱的药物不良反应预测系统中,都是采用将药物不良反应预测问题转化为多标签预测问题。然而,传统算法是将多标签问题转化为多个二分类问题,针对不同标签分别建模。这种方法往往忽视了标签之间的相关性、且预测的不良反应种类相对固定。例如不良反应呕吐和腹泻存在一定的相关性,但多标签分类方法针对二者分别单独建模,未捕捉和使用二者之间的关联关系;并且,多标签分类方法只能预测出某种药物是否具有标签中的某种不良反应,导致其最多预测出的不良反应种类数量等于已知标签数量,无法预测尚未知的不良反应,预测范围受限。
10、综上,以上现有技术公开的各药物不良反应的预测方法的预测全面性、准确性都有待提升。
技术实现思路
1、本发明要解决的技术问题是:提供一种基于深度学习异构网络的药物不良反应预测方法与系统。其目的在于借助嵌入权重的图神经网络强大的图数据结构数据处理能力和transformer decoder模块强大的生成能力,构建一个端到端的深度学习模型框架,学习药物与不良反应之间的潜在联系,以此更加全面、完整、准确地预测药物的潜在的不良反应。
2、为了解决上述技术问题,本发明采用以下技术方案:
3、第一方面,本发明提供一种基于深度学习异构网络的药物不良反应预测方法,具体包括如下步骤:
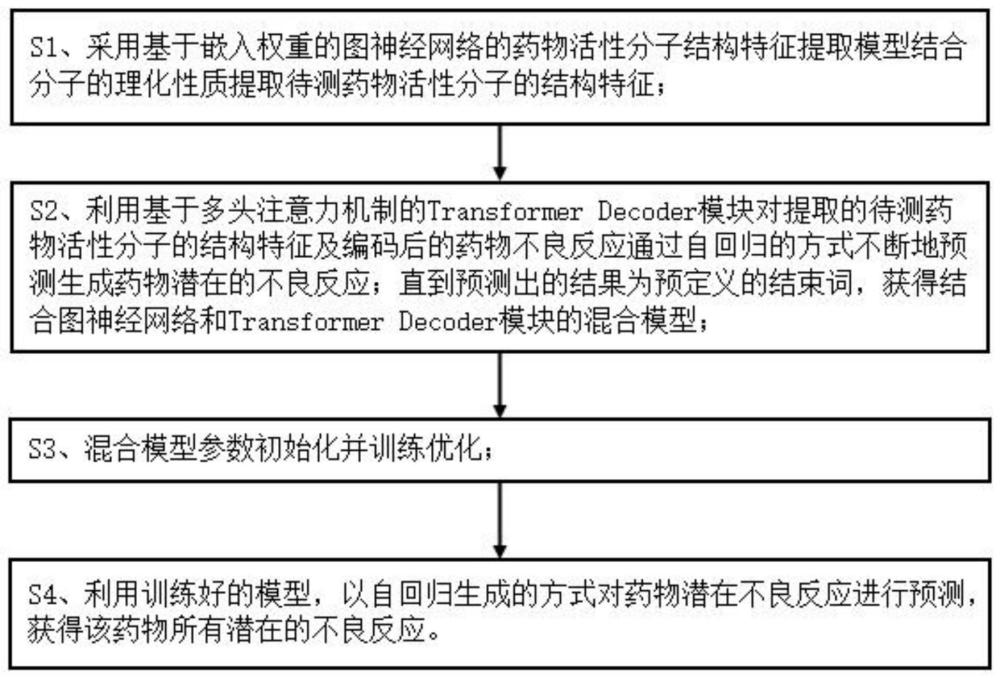
4、s1、采用基于嵌入权重的图神经网络的药物活性分子结构特征提取模型结合分子的理化性质提取待测药物活性分子的结构特征;
5、s2、利用基于多头注意力机制的transformer decoder模块对提取的待测药物活性分子的结构特征及编码后的药物不良反应词向量通过自回归的方式不断地预测生成药物潜在的不良反应;直到预测出的结果为预定义的结束词,获得结合图神经网络和transformer decoder模块的混合模型;
6、s3、混合模型参数初始化与训练优化;
7、s4、利用训练好的模型,以自回归生成的方式对药物潜在不良反应进行预测,获得该药物所有潜在的不良反应。
8、进一步地,
9、步骤s1具体包括如下步骤:
10、s101、获取待测药物活性分子的原子邻接矩阵和原子特征矩阵;
11、s102、将原子作为图数据结构中的节点,原子之间的化学键作为图数据结构中的边,构建基于嵌入权重的图神经网络的药物分子结构特征提取模型;
12、s103、将步骤s101获取的原子特征矩阵和原子邻接矩阵输入到步骤s102构建的药物分子结构特征提取模型,获得聚合后的原子特征矩阵。
13、进一步地,步骤s101具体为:使用rdkit工具包中的方法从以inchi格式表示的待测药物活性分子数据中提取出原子对象及原子之间的原子邻接矩阵,并将原子对象转化为以数值向量表示的原子特征向量,获得该药物活性分子的原子特征矩阵。
14、进一步地,步骤s1获得待测药物活性分子的聚合后的原子特征矩阵的过程便是更新该药物活性分子中每个原子特征的过程,对于待测药物活性分子的每个原子,使用嵌入权重的图神经网络基于邻接矩阵计算该原子的特征向量与其所有邻居原子的特征向量之间的注意力权重,并使用注意力权重和对应的邻居原子特征更新自身的特征,具体过程包括如下步骤:
15、(1)对于每个原子hi,图神经网络首先使用一个共享矩阵w对其特征做线性变换;
16、(2)随后计算原子hi与邻居原子hj之间的注意力分数eij,用于指示不同邻居原子hj对当前原子hi的重要性;计算公式如下:
17、
18、其中,和分别表示当前原子与其邻居原子;w表示对原子特征进行线性变换的共享矩阵;eij为计算出来的注意力分数;α表示注意力分数的计算过程;
19、(3)之后使用softmax函数对注意力分数进行归一化处理,获取注意力权重αij;计算公式如下:
20、
21、其中,exp表示对数函数的计算过程;softmax函数将注意力分数映射为一个介于0-1之间的小数,该小数为当前原子与其邻居原子之间的注意力权重;αij为计算出来的注意力权重;
22、(4)最后使用该权重来计算、聚合当前原子的所有邻居原子的特征,并通过一个非线性变换函数б,将聚合后的特征作为当前原子新的特征,即完成了原子特征的更新;
23、计算公式如下:
24、
25、其中,б为非线性变化函数;为更新后的当前原子的特征矩阵;
26、(5)将药物活性分子中的所有原子按照步骤(1)-(4)进行特征更新,更新后的所有原子特征向量组成更新后的原子特征矩阵,用于下一步操作。
27、进一步地,步骤s2具体包括如下步骤:
28、s201、将以umls(统一医学语言系统,unified medical language system,简称umls)id表示的药物不良反应转化为以数值表示的词向量,并加入位置编码,获得加入位置编码的词向量矩阵;
29、s202、构建基于多头注意力机制的transformer decoder模块:将步骤s103的聚合后的原子特征矩阵和步骤s201的加入位置编码的词向量矩阵输入到transformer decoder模块,输出中间特征矩阵;
30、s203、使用softmax函数将中间特征矩阵转化为药物潜在不良反应的概率表示,取概率最大的潜在不良反应作为本次预测结果;
31、s204、将步骤s203本次(如n次)预测出来的药物潜在不良反应与之前所有次(指前n-1次的所有次结果)预测出来的药物潜在不良反应一起作为输入数据重新输入到步骤s202的transformer decoder模块中,进行下一次预测;
32、也就是指将之前所有的预测结果作为下一次预测的模型输入。比如:模型第一次输入是1,预测结果2;那么第二次输入就是1,2,预测结果是5;那么第三次输入就是1,2,5.以此类推。
33、s205、重复s202-s204,直到预测出的结果为预定义的结束词,得到结合图神经网络和transformer decoder模块的混合模型。
34、进一步地,步骤s201具体为:将同一个待测药物的所有不良反应当做句子,将每个不良反应当做句子中的单词,并在句子的首尾加入开始词和结束词;使用embedding技术将以统一医学语言系统(unified medical language system,umls)id表示的药物不良反应转化为以数值表示的词向量,并在词向量中加入位置编码,以表示相对位置关系,获得加入位置编码的词向量矩阵。
35、进一步地,
36、步骤s201中位置编码是指用于表示单词间相对位置关系的编码向量,位置编码的方式有很多种。优选地,本发明是采用的transformer decoder模块中基于正余弦函数的位置编码方式给词向量添加相对位置信息;其中,偶数位置使用正弦函数,奇数位置使用余弦函数。
37、进一步地,
38、步骤s201中将同一个待测药物的所有不良反应当做句子,将每个不良反应当做句子中的单词具体是指:将数据集中以药物-不良反应对形式存在的药物-不良反应关系,通过预处理步骤转换为药物-[不良反应1,不良反应2,不良反应3,...]的表示形式,[不良反应1,不良反应2,不良反应3,...]整体看做一个句子,其中的不良反应1,不良反应2,不良反应3等看做一个单词,最终预测结果的输出单位是单词。
39、进一步地,
40、步骤s202中的transformer decoder模块由多个decoder layer堆叠而成,每个decoder layer对输入数据都做相同的操作,主要包括词向量自身的多头注意力的计算和词向量与药物原子特征矩阵之间多头注意力的计算。
41、transformer decoder模块一般是指google公司在2017年提出的transformer模型中的decoder模块(解码器)。
42、进一步地,s203中的softmax函数可以将特征映射为介于0-1之间的小数,这个小数便是某种药物具有某种不良反应的概率。
43、进一步地,
44、步骤s3混合模型参数初始化具体包括:
45、药物分子结构特征提取模型中每个原子初始的特征向量、最终输出的原子特征矩阵维度、最后一层网络结构的激活函数、对原子特征向量进行线性变换的共享矩阵w和为缓解过拟合现象设置的神经元随机失活比例;以及transformer decoder模块中的多头注意力的头数、词向量的维度、词典的大小和解码器的层数;对混合模型的学习率和迭代次数进行设定;采用交叉损失函数作为整个模型的损失值,进行逐层反向传播,利用adam迭代更新模型中的参数;其中,交叉损失函数loss的表达式如下:
46、
47、n表示词库中不良反应的种类;yi表示输入的第i个药物不良反应;表示模型预测出的结果为该药物潜在不良反应的概率值。
48、第二方面,本发明还提供一种采用如上所述基于深度学习异构网络的药物不良反应预测方法的预测系统,具体包括:药物活性分子的结构特征提取模块、混合模型构建模块、模型优化模块、预测模块,其中:
49、所述药物活性分子的结构特征提取模块,用于构建基于嵌入权重的图神经网络的药物活性分子结构特征提取模型,并结合分子的理化性质提取待测药物活性分子的结构特征;
50、混合模型构建模块,用于利用基于多头注意力机制的transformer decoder模块对提取的待测药物活性分子的结构特征及编码后的药物不良反应通过自回归的方式不断地预测生成药物潜在的不良反应;直到预测出的结果为预定义的结束词,获得结合图神经网络和transformer decoder模块的混合模型;
51、模型优化模块,用于对构建的混合模型参数初始化并训练优化;
52、预测模块,用于利用训练好的模型,以自回归生成的方式对药物潜在不良反应进行预测,获得该药物所有潜在的不良反应。
53、进一步地,所述药物活性分子的结构特征提取模块包括:
54、原子邻接矩阵及原子特征矩阵获取单元,用于获取待测药物活性分子的原子邻接矩阵和原子特征矩阵;
55、药物分子结构特征提取模型构建单元,用于将原子作为图数据结构中的节点,原子之间的化学键作为图数据结构中的边,构建基于嵌入权重的图神经网络的药物分子结构特征提取模型;
56、聚合原子特征矩阵获取单元,用于将获取的原子特征矩阵和原子邻接矩阵输入到步骤s102构建的药物分子结构特征提取模型,获得聚合后的原子特征矩阵。
57、进一步地,混合模型构建模块包括:
58、词向量转化与位置编码单元,用于将umls id表示的药物不良反应转化为以数值表示的词向量,并加入位置编码,获得加入位置编码的词向量矩阵;
59、解码器单元:用于将聚合后的原子特征矩阵和加入位置编码的词向量矩阵输入到transformer decoder模块,输出中间特征矩阵;
60、归一化处理单元,用于使用softmax函数将中间特征矩阵转化为药物潜在不良反应的概率表示,取概率最大的潜在不良反应作为本次预测结果;
61、自回归生成单元,用于重复预测过程直到预测出的结果为预定义的结束词,得到结合图神经网络和transformer decoder模块的混合模型。
62、第三方面,本发明还提供一种计算机存储介质,其上存储有计算机程序,其中所述计算机程序被执行器执行时实现如上所述的基于深度学习异构网络的药物不良反应预测方法。
63、第四方面,本发明还提供一种电子设备,包括:
64、存储器,其上存储一个或多个程序;一个或多个处理器;
65、当所述一个或多个程序被所述一个或多个处理器执行时,使得所述一个或多个处理器实现如上所述的基于深度学习异构网络的药物不良反应预测方法。
66、本发明中的名词解释:
67、rdkit(the redox toolkit)是一个用于分析和可视化化学数据的开源化学信息学工具包。rdkit主要用于药物设计、生物活性预测、化学反应预测和化学数据处理等领域。
68、inchi,英文全称为international chemical identifier,即国际化合物标识,是由国际纯粹与应用化学联合会和美国国家标准技术研究所(nist)联合制定的,用以唯一标识化合物iupac名称的字符串。
69、transformer decoder:transformer是一种神经网络架构,它的核心部分是由若干个encoder(编码器)和decoder(解码器)组成的。
70、embedding技术是一种词嵌入技术,通过对不同的单词进行编码,进而将其映射为一个表征向量实现将单词映射为数值向量、方便深度学习技术模型处理的目的。
71、本发明具有如下有益效果:
72、本发明提供一种基于深度学习异构网络的药物不良反应预测方法及系统。本发明使用能够处理非欧式空间数据的图神经网络提取药物分子结构特征,充分利用药物活性分子天生具有的图数据结构特征。同时借助transformer decoder模块强大的生成能力,构建了一个端到端的网络表示学习方法,可以较为准确的描述药物分子的结构特征和理化性质,提高预测结果的全面性、完整性和准确性。帮助研究人员更快的挖掘出更多的、更准确的潜在药物不良反应,加快药物研发速度。
73、本发明将药物分子作为图数据结构处理具体是指将原子作为图数据结构中的节点,将原子之间的化学键作为图数据结构中的边。利用处理图数据结构能力较强的图神经网络结合权重嵌入的方式聚合数据并使用使原子的理化性质编码原子的特征,可以较为准确的描述药物分子的结构特征和理化性质,进而提高方法中的模型性能。
74、本发明还利用了transformer decoder模块强大的生成能力,结合药物分子结构特征和理化性质使用自回归预测的方式预测药物潜在的不良反应。在预测第n个不良反应时,会利用前n-1个不良反应的信息,而不是针对每个不良反应单独建模,在一定程度上利用到了不良反应之间的内在联系,且只有预测到事先预设的结束词时才会结束整个预测过程,使方法中的模型可能预测出数据集中的任一种不良反应,预测出的不良反应的种类和数量更为完整和全面。因此相对于现有的技术方案,本发明实例提供的方法能够更为完整,全面且准确的预测出药物潜在的不良反应。
75、最后,本发明提供的是一种端到端的方法,即给定输入后,全程不需要人工干预,模型方法会自动输出所有潜在的不良反应,操作简便。
76、本发明提供的方案与已有的药物不良反应预测方案相比,本发明方法中提供的模型结构简单,操作简便;能够更为完整、全面且准确的预测出药物潜在的不良反应。
本文地址:https://www.jishuxx.com/zhuanli/20240808/270956.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表