基于eDNA的多营养级联合物种分布模型构建方法及应用
- 国知局
- 2024-11-19 09:41:19
本发明涉及一种基于edna的多营养级联合物种分布模型构建方法及应用,属于生态修复。
背景技术:
1、环境dna(edna)技术作为一种新兴的生物监测方法,可采集游离在水体中的生物dna,通过分析来检测和量化各种生物的存在。这种非侵入性和高灵敏性的技术能够在不干扰生物活动和栖息地的情况下推测生态系统状态,极大地提高了生物信息的采集效率,并为环境监测和保护提供了强有力的工具。
2、传统的水生生物监测往往依赖于传统的采样和鉴定方法,这些方法费时费力且对生物群落组成的检测有限。
3、联合物种分布模型(joint species distribution model,jsdm)是一种基于贝叶斯框架和极大似然估计的多元线性混合模型,能够拟合物种发生和环境因子的关联,并且能够量化关联影响程度的大小。相较于传统的单一物种分布模型,jsdm考虑了物种间的相互影响,克服了分析单个物种与环境关系的局限性。在hmsc框架下,分层物种群落模型利用物种的出现数据和环境变量建立模型,同时建立性状数据和环境响应的关系,推测性状差异带对环境响应差异的影响情况。此外,物种间残余关联矩阵可以用来揭示在排除环境因素影响后可能存在的种间相互作用,进一步深化对生态系统内复杂交互作用的理解。
4、水生生物是水生环境的重要组成部分,与水生态状况密切相关,对环境变化具有较高的敏感性和响应性,能够指示水生态环境质量。生态系统功能是通过不同营养级物种及其相互作用实现的,物质和能量通过食物网传递到高营养级从而影响整个生态系统。不同营养级物种之间存在复杂的相互作用,从营养级的角度分析物种在不同环境梯度下的群落组成和响应特征至关重要。
5、目前关于水生态系统中物种分布及环境响应的评估大多基于单一营养级物种或分别评估不同营养级物种的特征,而忽略了不同营养级物种之间的相互作用,未将多营养级物种作为整体纳入评估体系。
技术实现思路
1、为了解决上述存在的问题,本发明公开了一种基于环境dna(即edna)的多营养级联合物种分布模型构建方法及应用,其具体技术方案如下:
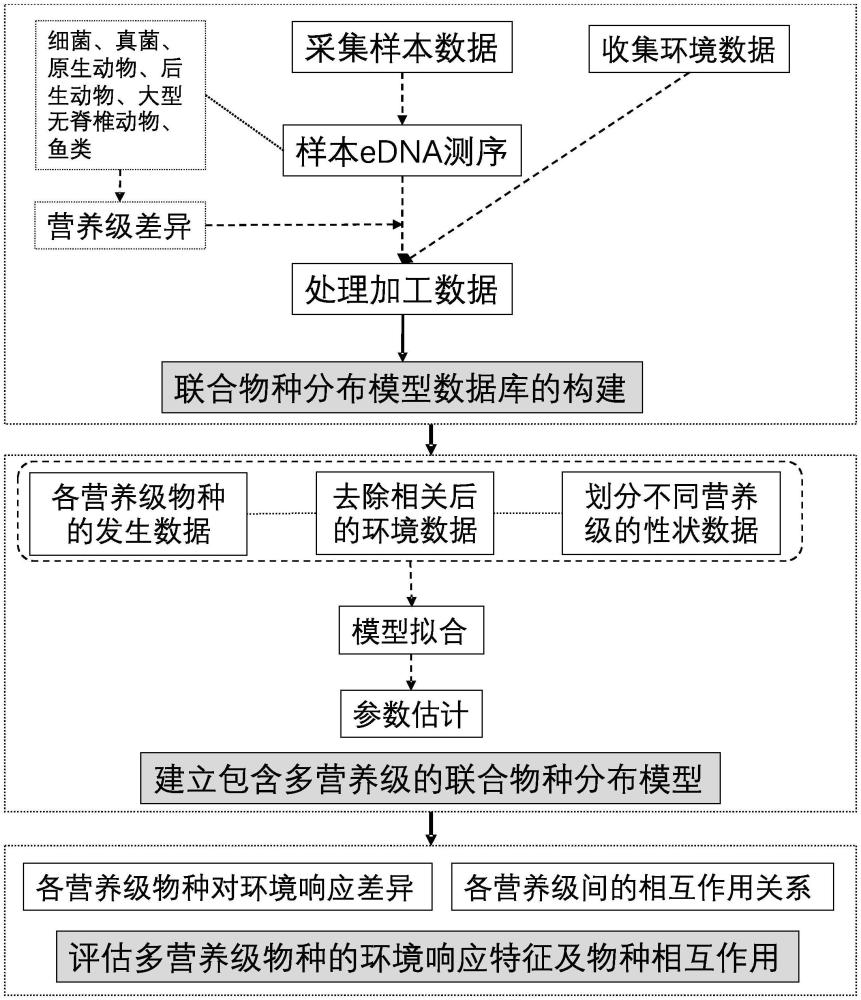
2、一种基于edna的多营养级联合物种分布模型构建方法,包括以下步骤:
3、(1)设定采样点:确定研究区域,根据评估内容和研究区域的现场情况设定采样点;
4、(2)样本采集:采集目标样本,测量所需的环境数据,对目标样本进行环境dna测序以获取生物信息数据;
5、(3)数据处理:整理生物信息数据,将物种划分为不同营养级并整理加工物种发生数据;
6、(4)构建模型:进行模型拟合和参数估计,建立多营养级物种联合的物种分布模型;
7、(5)结果评估:根据模型参数的拟合结果评估不同营养级物种的环境响应特征,评估营养级差异对物种环境响应特征的影响程度,评估不同营养级联合下的群落组成和环境响应情况。
8、进一步的,所述环境数据包括连续型变量和离散型变量,将环境数据进行相关性检验,去除强相关的环境协变量,并且对环境数据进行标准化处理。
9、进一步的,所述采集的目标样本经edna测序获取otu(operationaltaxonomicunits)或asv(ampliconsequencevariants)数据,数据是表格的形式,每一行是一个序列信息,列是各样点的序列数值,数据处理与生物量计算基于otu或asv数据,根据otu或asv数据中的注释信息,与基础数据库进行比对,确定序列对应的物种或分类单元,根据已知的物种信息及其在食物网中的位置,划定各物种的营养级水平,物种的营养水平包括细菌、真菌、原生动物、后生动物、大型无脊椎动物和鱼类。
10、进一步的,所述数据处理过程具体为:
11、筛选和过滤假阳性数据,去除otu或asv数据中存在的假阳性结果,具体为,去除otu或asv绝对丰度小于等于2的序列,otu或asv序列小于2的认定为假阳性数据;
12、经分段函数变换
13、
14、转化为二进制矩阵,物种出现为1,没有出现为0;
15、将绝对丰度数据矩阵转化为0-1矩阵,0-1表示的存在-缺失数据,或通过其他经验公式计算得到edna与物种数量之间的换算关系,进而估算物种的数量;
16、物种的营养级是基于分类学信息的结构域进行划定。
17、进一步的,所述物种发生数据的整理加工是在连续型变量的情况下,对物种发生数据进行归一化和标准化处理;采集的环境数据进行相关性检验或主成分分析以去除强相关性的环境数据对模型结果的影响,并对环境数据进行标准化处理。
18、进一步的,联合物种分布模型是hmsc框架的联合物种分布模型,在r语言的hmsc程序包中实现,所述hmsc框架的联合物种分布模型的建立和拟合的过程特征在于:
19、数据的准备和处理;
20、设置模型结构并拟合模型;
21、检验模型的收敛情况;
22、计算拟合优度以评估模型解释力;
23、参数估计并可视化参数结果。
24、进一步的,所述hmsc框架的联合物种分布模型涉及的矩阵包含物种矩阵、环境协变量矩阵、性状矩阵,物种矩阵和环境矩阵、性状矩阵和环境矩阵采用混合线性回归的方法构建模型结构,并拟合模型完成联合环境变量和多物种的定量分析;
25、性状矩阵的数据是不同的营养级水平。
26、进一步的,所述hmsc框架的联合物种分布模型建立和拟合过程为,模型采用马尔科夫链蒙特卡洛方法(markovchainmontecarlo)进行拟合,统计有效样本尺寸以判断模型是否收敛;
27、模型的拟合度、解释力的评估以参数为依据进行,以最大为依据确定模型结构。
28、一种基于上述的基于edna的多营养级联合物种分布模型构建方法的应用,基于模型结果评价多营养级物种对环境因素的响应,探索多营养级物种沿环境梯度的群落组成变化,并评估营养级差异影响下的环境响应差异。
29、进一步的,根据参数估计结果对物种响应环境进行评估,输出参数beta衡量物种的环境响应,基于beta参数评估不同营养级物种对环境响应的方向和大小,beta参数为正表明物种对环境的响应是正向的,参数为负则表明物种对环境的响应是反向的,其绝对值越大表明物种对环境的响应越强烈、物种对环境则更为敏感,进一步整合所有物种的环境响应情况,识别对群落影响强烈的关键环境因子;
30、基于模型输出参数gamma衡量不同营养级分组的环境响应差异,进行不同营养级环境响应差异的评估,该值为正则表明该营养级在群落水平上对环境的平均响应是正向的,反之则为负向的,进一步对数值大小的比较对比不同营养级对环境响应程度的差异;
31、基于物种残差相关矩阵评估去除了环境变量后的种间关联情况,根据相关性绝对值在0.2-0.8区间内进行物种过滤,保留过滤后的相关物种互作,进一步根据物种所属的营养级水平进行归类,基于过滤后的矩阵中关联值大小建立两两物种的关联网络,关联网络中每个节点与周围节点的连接反映了该节点处物种所代表营养级的关联情况,以此评估不同营养级相互作用方向和大小。
32、本发明的工作原理是:
33、本发明基于edna技术和联合物种分布模型,发明一种用于评估多营养级物种环境响应的方法,通过edna技术可以最大限度地获取区域内所有物种的信息。在hmsc框架下的联合物种分布模型处理多营养级物种,能够高效评估多营养级物种在生物因素和非生物因素共同作用下的物种分布和环境响应。edna技术和联合物种分布模型相结合能够最大限度地挖掘环境中的数据,尤其是一些稀有物种,实现对多营养级物种环境响应和相互作用关系的量化评估。本发明涉及不同营养级之间的相互关联,从更系统的角度完成对多营养级物种分布和环境响应的评估。
34、本发明的有益效果是:
35、本发明基于环境dna技术能够弥补传统采样鉴定信息收集不完全的缺陷,尽可能的收集环境中的物种信息。
36、本发明考虑了多个营养级之间的相互作用,得到多个营养级物种之间的互作关系,能够弥补只考虑单一营养级物种建模的不足,综合判断不同营养级物种的环境响应情况。
37、本发明结合联合物种分布模型,构建同时包含物种发生情况、环境条件数据、采样点位随机效应的物种分布模型,最大限度的解析群落分布的环境驱动因素,并去除环境影响进行物种的关联评估。
38、本发明基于数据驱动模型进行评估,其结构更简单易操作,输出数据快捷便于解读。
39、本发明为对物种水生生物监测和评估提供技术支撑,对生物多样性和水生态环境保护具有重要意义。
本文地址:https://www.jishuxx.com/zhuanli/20241118/329800.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。