一种添加的掩码的scRNA-seq测序数据PCA降维方法
- 国知局
- 2025-01-10 13:31:59
本发明涉及生物信息,更具体地说,它涉及一种添加的掩码的scrna-seq测序数据pca降维方法。
背景技术:
1、scrna-seq测序是一种重要的基因组学技术,可以对单个细胞进行高通量测序,从而揭示细胞间的差异和多样性。而降维则是scrna-seq测序数据分析中的一个重要步骤,旨在将高维数据转化为低维表示,以便更好地理解和解释数据。
2、随着scrna-seq测序技术的发展,每个研究或实验中测定的细胞数量在显著增加,少则产生几百,多则产生几十万的细胞数量,甚至更多。其中,细胞亚型的鉴定是scrna-seq测序技术一个非常重要的基础应用。但由于scrna-seq测序数据通常涉及到很多细胞,但由于scrna-seq测序数据通常涉及到很多细胞,而每个细胞中的基因数量又可能是几万个,所以,scrna-seq测序数据是一个高维的复杂数据,为了有效地对scrna-seq测序数据进行各种处理分析,特别是细胞亚型的鉴定,通常需要首先对scrna-seq测序数据进行降维。pca就是一种常用的降维技术,它通过寻找数据集中的主成分(主方向),将高维数据映射到低维空间。主成分是使得高维数据的方差最大化的低维空间。pca的核心思想是通过将高维数据的协方差矩阵的特征值和特征向量来表示数据的主成分。
3、然而,scrna-seq测序数据具有高纬度,高稀疏等特性,在进行pca降维之前往往需要进行特征选择,筛选出高表达基因。但是,目前的降维方法侧重于挖掘高表达基因之间的线性关系,忽略了低表达基因之间的潜在联系,虽然降维由高表达基因构成的基因表达矩阵可以获得更好的降维效果,但也会丢失低表达基因之间的潜在联系。
4、因此,本发明旨在提供一种添加的掩码的scrna-seq测序数据pca降维方法,用以解决上述问题。
技术实现思路
1、本发明的目的是提供一种添加的掩码的scrna-seq测序数据pca降维方法,本发明通过使用svd方法对数据矩阵进行奇异值分解得到对应的特征值与特征向量,同时使用筛选后的占比较大的特征值对应的特征向量与原矩阵进行点乘得到降维后的数据矩阵,并且为了更好的找到区分数据的投影方向,通过先使用经过处理的掩码矩阵,掩码矩阵掩盖掉了低可变的细胞,然后根据该掩码矩阵求得对应的特征值与特征向量,但是为了不损失原本的信息,本发明使用求得的特征向量与原数据矩阵进行点乘,得到降维矩阵,这样既能更好的降维,同时也不损失原本的信息。
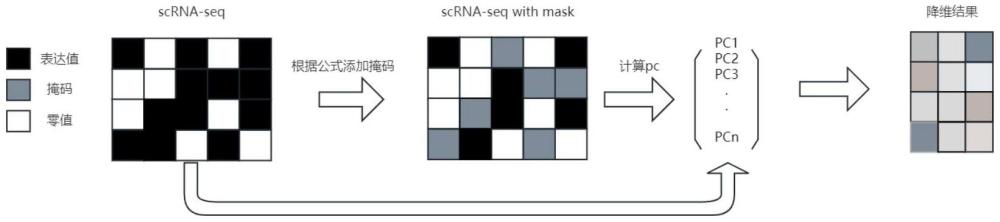
2、本发明的上述技术目的是通过以下技术方案得以实现的:一种添加的掩码的scrna-seq测序数据pca降维方法,包括以下步骤:
3、s1、采集ncbi网站上的公共数据集并进行质量控制与特征选择,得到原始基因表达矩阵;
4、s2、添加掩码,在原始基因表达矩阵中筛选并掩盖低可变基因,从而得出掩码矩阵;
5、s3、奇异值分解,根据掩码矩阵使用svd分解求出降维矩阵;
6、s4、pca降维,根据降维矩阵的特征值与原始基因表达矩阵相乘,得到降维结果;
7、s5、聚类可视化,使用leiden方法进行聚类,并使用t-sne方法进行可视化。
8、本发明进一步设置为:所述步骤s1中数据预处理分别包括归一化处理、缩放处理和特征选择处理。
9、本发明进一步设置为:所述步骤s2中添加掩码的具体流程为:首先通过特征选择,从原始基因表达矩阵中筛选出低可变基因,将其设置为0,从而构造得出掩码矩阵。
10、本发明进一步设置为:所述步骤s3中奇异值分解的具体流程为:使用svd方法对掩码矩阵进行奇异值分解得到对应的特征值与特征向量,然后使用筛选后的占比较大的特征值对应的特征向量与原始基因表达矩阵进行点乘得到降维矩阵。
11、本发明进一步设置为:所述步骤s4中pca降维的具体流程为:使用原始基因表达矩阵与降维矩阵相乘得到降维结果,用于保留掩码所覆盖的信息。
12、本发明进一步设置为:所述步骤s5中聚类可视化的具体流程为:将降维结果作为输入,使用leiden聚类方法对数据进行聚类,并使用细胞类型标签对可视化结果进行着色。
13、综上所述,本发明具有以下有益效果:
14、本发明通过使用svd方法对数据矩阵进行奇异值分解得到对应的特征值与特征向量,同时使用筛选后的占比较大的特征值对应的特征向量与原矩阵进行点乘得到降维后的数据矩阵,并且为了更好的找到区分数据的投影方向,通过先使用经过处理的掩码矩阵,掩码矩阵掩盖掉了低可变的细胞,然后根据该掩码矩阵求得对应的特征值与特征向量,但是为了不损失原本的信息,本发明使用求得的特征向量与原数据矩阵进行点乘,得到降维矩阵,这样既能更好的降维,同时也不损失原本的信息。
技术特征:1.一种添加的掩码的scrna-seq测序数据pca降维方法,其特征是:包括以下步骤:
2.根据权利要求1所述的一种添加的掩码的scrna-seq测序数据pca降维方法,其特征是:所述步骤s1中数据预处理分别包括归一化处理、缩放处理和特征选择处理。
3.根据权利要求1所述的一种添加的掩码的scrna-seq测序数据pca降维方法,其特征是:所述步骤s2中添加掩码的具体流程为:首先通过特征选择,从原始基因表达矩阵中筛选出低可变基因,将其设置为0,从而构造得出掩码矩阵。
4.根据权利要求1所述的一种添加的掩码的scrna-seq测序数据pca降维方法,其特征是:所述步骤s3中奇异值分解的具体流程为:使用svd方法对掩码矩阵进行奇异值分解得到对应的特征值与特征向量,然后使用筛选后的占比较大的特征值对应的特征向量与原始基因表达矩阵进行点乘得到降维矩阵。
5.根据权利要求1所述的一种添加的掩码的scrna-seq测序数据pca降维方法,其特征是:所述步骤s4中pca降维的具体流程为:使用原始基因表达矩阵与降维矩阵相乘得到降维结果,用于保留掩码所覆盖的信息。
6.根据权利要求1所述的一种添加的掩码的scrna-seq测序数据pca降维方法,其特征是:所述步骤s5中聚类可视化的具体流程为:将降维结果作为输入,使用leiden聚类方法对数据进行聚类,并使用细胞类型标签对可视化结果进行着色。
技术总结本发明公开了一种添加的掩码的scRNA‑seq测序数据PCA降维方法,涉及生物信息技术领域,其技术方案要点是:包括采集数据并进行预处理,得到原始基因表达矩阵;添加掩码,筛选并掩盖低可变基因,从而得出掩码矩阵;奇异值分解,使用SVD分解求出降维矩阵;PCA降维,根据降维矩阵的特征值与原始基因表达矩阵相乘,得到降维结果;聚类可视化,使用leiden方法进行聚类,使用T‑SNE方法进行可视化。本发明提出了一种添加的掩码的scRNA‑seq测序数据PCA降维方法,通过添加掩码的方式保留低表达基因之间的潜在联系,同时采用从基因表达综合中心(GEO)下载的数据集,经过传统PCA与掩码PCA方法分别降维,通过聚类精确度比较方法效果,结果表明了在大部分数据集上,聚类效果得到了明显提升。技术研发人员:朱晓姝,刘嘉辉,罗笑南,蒙霜,滕飞受保护的技术使用者:桂林电子科技大学技术研发日:技术公布日:2025/1/6本文地址:https://www.jishuxx.com/zhuanli/20250110/353751.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表