一种含BsmBI酶切位点的引物对及其应用
- 国知局
- 2024-06-20 11:13:43
本发明属于基因工程,尤其涉及一种含bsmbi酶切位点的引物对及其应用。
背景技术:
1、crispr/cas9系统是目前最流行的基因敲除系统之一,利用sgrna和cas9两个主要元件实现基因敲除目的。crispr/cas9系统实现基因敲除的原理如下:sgrna含有特异性靶点序列,可引导cas9核酸酶准确结合到基因组特异位置,并切割基因组dna,形成dna双链断裂,生物体的非同源末端连接修复会导致靶点处发生插入缺失,从而引发移码突变,导致基因功能丧失,实现基因敲除目的。常规的crispr/cas9系统由两个病毒载体构建而成,将sgrna与cas9蛋白在两个载体系统中分开表达,构建过程操作繁琐、时间长。
2、转座子是一种可在基因组dna改变位置的序列,也称为跳跃基因,最早由美国遗传学家mcclintock在研究玉米籽粒的颜色变异中发现。睡美人(sleeping beauty,sb)转座子系统已被广泛应用于基因递送,其工作原理如下:转座酶sb100x识别转座子两端特异反向重复序列(irdr),将转座子序列切出,通过dna攻击的方式插入新的基因组位置。与传统病毒载体感染构建稳定细胞株的方法相比,sb转座子系统缩短了稳定细胞株的构建时间,避免了包装病毒过程中所产生的生物安全问题,同时提高了稳定重组的效率。
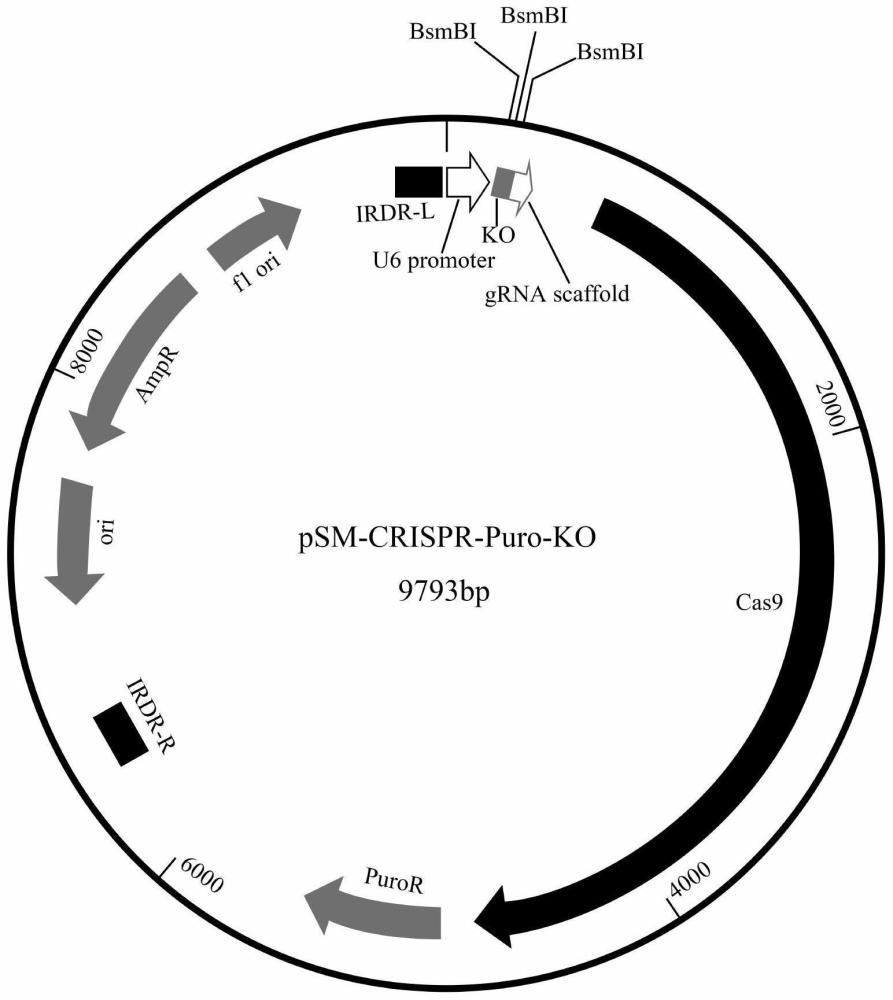
3、中山大学孙逸仙纪念医院郭雅彬研究员及其团队于2018年创造性地将sb转座子系统与crispr/cas9系统融合,开发出转座子协同crispr/cas9系统的稳定敲除单质粒载体psm-crispr-puro(zl201710880749.3),该单质粒载体仅需要一次构建、一次转染即可实现sgrna和cas9蛋白的表达,过程简单、高效、快捷,大大简化质粒构建流程,可缩短实验周期,提高工作效率。然而本发明人在实际使用psm-crispr-puro质粒过程中发现,在构建靶向位点敲除质粒流程中,仍旧需要酶切-琼脂糖凝胶电泳-胶回收酶切产物。为了进一步简化质粒构建流程,本发明人发明了一种含bsmbi酶切位点的引物对,并以psm-crispr-puro载体为基础,构建出psm-crispr-puro-ko载体,可实现酶切-纯化回收酶切产物之后即可用于靶向位点敲除质粒构建,省略了琼脂糖凝胶电泳及紫外线下切胶这两个操作过程。
技术实现思路
1、为了解决上述背景所提出的技术问题,本发明提供了一种含bsmbi酶切位点的引物对,并以psm-crispr-puro载体为基础,利用含bsmbi酶切位点的引物对(39bp)替换psm-crispr-puro载体中grna scaffold序列之前的骨架序列(1889bp),构建出psm-crispr-puro-ko载体,进一步简化了靶向位点敲除质粒的构建流程。
2、该载体以更为方便快捷地用于基因组编辑。该单质粒载体仅需一次转染即可完成sgrna与cas9蛋白的共同表达,同时载体上融入的sleeping beauty转座子元件(irdr)在转座酶sb100x的作用下,将上述cas9系统整合受体基因组中,构建出目的基因敲除稳定株。
3、采取的技术方案如下:
4、本发明提供一种含bsmbi酶切位点的引物对,其核苷酸序列如seq id no:1所示。
5、seq id no:1的核苷酸序列如下:
6、ko-f:5′-caccggagacgatgcgtctcagctgaaccgtctct-3′
7、ko-r:5′-aaacagagacggttcagctgagacgcatcgtctcc-3′。
8、本发明的另一目的是提供一种单质粒载体psm-crispr-puro-ko的构建方法,以psm-crispr-puro载体为基础,利用含bsmbi酶切位点的引物对(39bp)替换psm-crispr-puro载体中grnascaffold序列之前的骨架序列(1889bp),构建出psm-crispr-puro-ko载体。
9、本发明的又一目的是提供一种转座子协同crispr/cas9系统的稳定敲除单质粒载体,是含有irdr-l-irdr-r盒的双链环状质粒,所述irdr-l-irdr-r盒包括irdr-l序列、启动子、ko序列、grnascaffold序列、cas9蛋白序列、抗性筛选基因序列和irdr-r序列。
10、进一步地,所述的irdr-l序列为seq id no:2中自5′端第9567位-9793位碱基的反向互补序列;所述的irdr-r序列为seq id no:2中自5′端第6421位-6648位碱基。所述的启动子是u6启动子,其序列为seq id no:2中自5′端第10位-250位碱基。所述的ko序列为seqid no:2中自5′端第255位-293位碱基。所述的grna scaffold序列为seq id no:2中自5′端第290位-365位碱基。所述的cas9蛋白序列为seq id no:2中自5′端第663位-4766位碱基。所述的抗性筛选基因是指嘌呤霉素(puromycin)抗性基因,其序列为seq id no:2中自5′端第4905位-5501位碱基。
11、本发明的又一目的是提供一种单质粒载体psm-crispr-puro-ko,所述的单质粒载体psm-crispr-puro-ko的核苷酸序列如seq id no:2所示,seq id no:2的核苷酸序列过长,这里不再详述,其序列可见序列表。psm-crispr-puro-ko质粒长度为9793bp。
12、本发明的又一目的是提供psm-crispr-puro-ko载体在构建基因敲除稳定株中的方法及应用,包括以下步骤:
13、步骤1:设计靶向目的基因的guide sequence序列;
14、步骤2:选取合适的sgrna序列,合成两条包含接头的引物;
15、步骤3:bsmbi酶切psm-crispr-puro-ko克隆载体,产物纯化回收,得纯化回收产物;
16、步骤4:引物退火,得退火产物;
17、步骤5:连接纯化回收产物与退火产物;
18、步骤6:连接产物转化感受态细胞,挑取单克隆,测序获得阳性菌;
19、步骤7:阳性菌株扩繁,大提质粒;
20、步骤8:将带有目的基因sgrna序列的psm-crispr-puro-ko载体与转座酶质粒sb100x共转染宿主细胞,使cas9系统整合至宿主基因组中;
21、步骤9:使用嘌呤霉素筛选得到目的基因敲除的稳转细胞株,扩大培养;
22、步骤10:收集培养的细胞,在非变性状态下裂解细胞,离心抽取蛋白,免疫印迹检测目的基因表达水平;获得目的基因敲除稳定株。
23、与现有技术相比,本发明具有如下优点:
24、(1)本发明了设计了一种含bsmbi酶切位点的引物对,以psm-crispr-puro载体为基础,构建出psm-crispr-puro-ko载体,可实现酶切-纯化回收酶切产物之后即可用于靶向位点敲除质粒构建,省略了琼脂糖凝胶电泳及紫外线下切胶这两个操作过程,简化了靶向位点敲除质粒的构建流程。
25、(2)紫外线下切割含目的条带的琼脂糖凝胶是现有技术(psm-crispr-puro)中的必要操作。然而紫外线对人体有致癌作用,本发明涉及的靶向位点敲除质粒构建流程不包含琼脂糖凝胶电泳及紫外线下切胶这两个操作过程,极大的保护了科研人员的实验安全。
26、(3)现有技术(psm-crispr-puro)中的psm-crispr-puro质粒长度为11643bp,本发明中的psm-crispr-puro-ko质粒长度为9793bp。质粒长度越小,回收产物与引物退火产物越容易连接。另外长度越小的重组子对感受态细胞的影响也越小,转化效率更高。与现有技术(psm-crispr-puro)相比,本发明中的质粒长度减小了1850bp,更容易与引物退火产物连接,对感受态细胞的影响也更小。整体来讲,本发明更容易构建靶向位点敲除质粒。
本文地址:https://www.jishuxx.com/zhuanli/20240619/1255.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表