一种基于哥隆尺的基因序列纠错算法与系统的制作方法
- 国知局
- 2024-07-31 20:11:55
本发明涉及数据编码与生物信息领域,具体涉及一种一种基于哥隆尺的基因序列纠错算法与系统进行生物信息中的dna序列纠错计算。
背景技术:
1、在进行dna存储的研究中,将利用基因序列作为存储信息的载体,对基因序列进行编码操作以存储特定信息,对基因序列进行解码操作进行读取对应信息,但基因序列在生物测序和自然生化演变过程中,会有概率发生一定的错误,其错误类型为替换错、插入错和删除错,这样直接对基因序列进行解码操作会无法读取出正确的信息,基因组的规模通常较大,因此希望在这种大规模下以一种低代价的纠错方式指出基因序列的错误信息并予以纠正。
2、目前,现有的dna序列纠错算法或系统具有以下技术问题:
3、前人的研究的传统纠错码通常无法应对生物dna环境中复杂的错误类型,若利用同步码策略冗余度大,成本代价高;所实验的对象多为人工无生物活性和规律性的dna序列,而自然界生物dna序列存在一定规律性。实验证明,无生命特征dna序列复制时,每次复制会引入16%的杂质,相比之下有生命特征的dna每次培养仅引入1.5%的杂质。此外对于大规模的基因组序列进行混合错误类型纠错时,传统纠错编码的结果可靠性也一直有待提高。
技术实现思路
1、有鉴于此,本发明提供了一种基于哥隆尺的基因序列纠错算法与系统,用于解决或至少部分解决传统dna序列纠错算法或系统中的一些弊端。
2、本发明所采用的技术方案是:
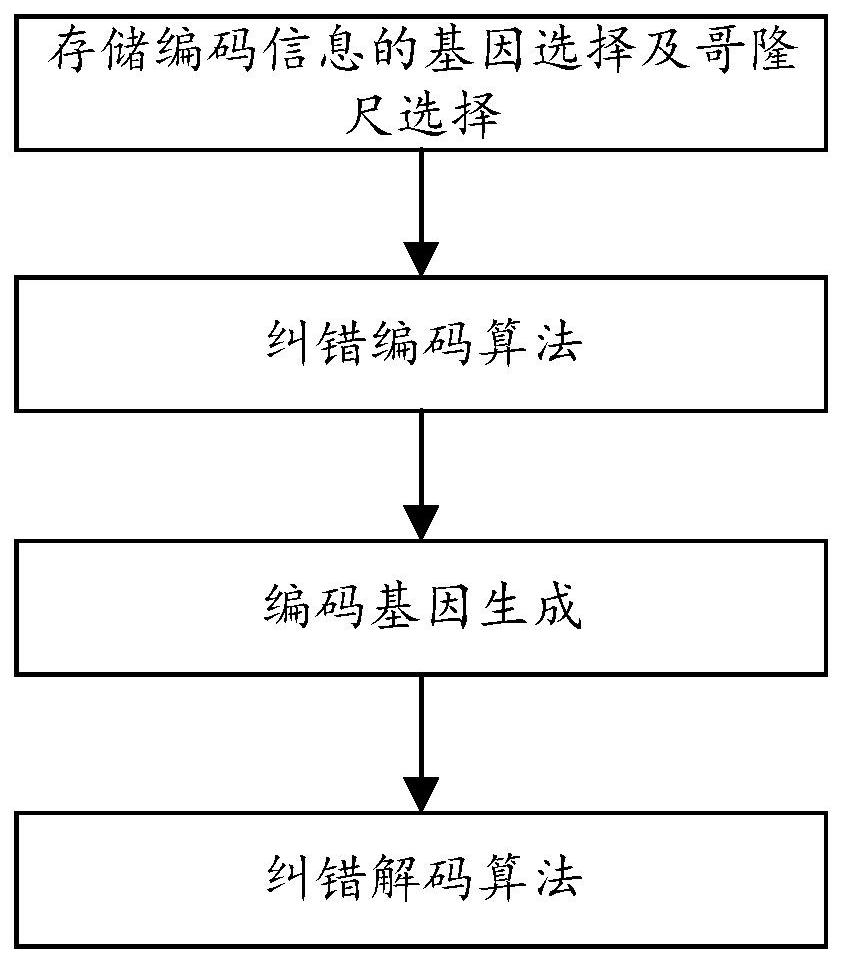
3、第一方面,本发明提供一种基于哥隆尺的基因序列纠错算法,包括编码和解码过程;
4、其中,所述编码过程包括以下步骤:
5、s110:编码前先确定用于存储纠错编码信息的基因,并根据基因的氨基酸长度根据规则选择一个最优哥隆尺,并将基因的起始密码子与终止密码子强制转换成指定的密码子;
6、s120:根据s110选择的哥隆尺构造滑动窗口,对被保护的长dna序列进行滑动扫描;
7、s130:将s120中滑动窗口扫描到的碱基组合成一个短的子序列,对该子序列进行哈希,生成两个哈希值;
8、选择一个存放纠错信息的基因,基因由一段氨基酸序列组成,设置一个与所选基因内氨基酸序列个数相同的数组,初始时数组内全为0;
9、s140:将s130中产生的第一个哈希值映射到存储编码信息的基因中的一个氨基酸中,确定该氨基酸的位置下标,再将s130中的另一个哈希值累加到该位置下标所对应的数组的位置上;
10、s150:重复s120~s140,直到被保护的长dna序列全部扫描完;
11、s160:将最终的存储编码信息的基因的氨基酸序列对应的数组中的数据按照密码子映射关系转换成一个确定的密码子,生成一个新的基因密码子序列;
12、s170:将s160得到的新的密码子序列替换基因的原始序列;
13、所述解码过程包括以下步骤:
14、s210:读入待纠错解码的dna序列,根据编码时标记的基因起始密码子与终止密码子找到存储纠错编码信息的基因;
15、s220:根据基因的氨基酸长度按照规则选择一个与编码时相同的最优哥隆尺;
16、s230:根据s220选择的最优哥隆尺构造滑动窗口;
17、s240:利用滑动窗口对待评估解码的长dna序列进行扫描,将扫描到的碱基组合成一个短的子序列,对该子序列进行哈希,生成两个哈希值;
18、选择一个存放纠错信息的基因,基因由一段氨基酸序列组成,设置一个与所选基因内氨基酸序列个数相同的数组,初始时数组内全为0;
19、s250:将s240中产生的第一个哈希值映射到一个与存储编码信息的基因的氨基酸等长的空白序列中的一个氨基酸位置,再另一个哈希值累加到对应位置下标的数组上;
20、s260:重复s240和s250,直到待评估解码的长dna序列全部扫描完;
21、s270:将最终重新生成的一个氨基酸序列对应的数组中的数据按照密码子映射关系转换成一个确定的密码子序列;
22、s280:将s270生成的新的密码子序列与找到的存储了编码信息的基因密码子序列进行比对,若相同氨基酸对应的密码子不相同,则进行一个标记,记作witness;
23、s290:再次对待纠错的dna序列进行哥隆尺滑动窗口扫描并进行哈希,若某次滑动窗口选择到的碱基组成的子序列的第一个哈希值映射到基因中的一个witness,则将滑动窗口刻度指向的碱基在原基础上增加数字1,表示打分增加1分;
24、s300:重复s290,直到待纠错解码的所有序列都扫描完,统计待纠错序列中的所有碱基的打分情况;
25、s310:对s300中得到的打分数组进行排序,选择得分最高的前若干个碱基,对其进行各种纠错尝试,在某次纠错尝试后重新对待纠错序列进行扫描哈希,计算witness,选择witness最小的那个方案作为本轮的纠错方案并进行纠错;
26、s320:重复s240到s310,直到计算出的witness为0,则表示序列无错,纠错成功,返回纠错后的序列。
27、进一步,所述编码s110包括:
28、最优哥隆尺的选择规则为:按照哥隆尺的长度4倍大于基因的氨基酸长度,隆尺的长度5倍小于基因的氨基酸长度来选择一个最优哥隆尺;
29、选择用于存储编码信息的基因时,选择一些必需基因或者在基因内部利用一些氨基酸来作为基因的纠错编码。
30、进一步,所述编码s130包括:将哥隆尺状的滑动窗口中哥隆尺刻度指向的碱基取出直接组合成一个新的子序列,对该子序列使用哈希算法生成一个长的哈希值,再从该长哈希值中按照固定的比特长度截取出两个新的哈希值。
31、进一步,所述编码s140包括:将s130子序列产生的两个新的哈希值中的第一个对基因的氨基酸长度取模,得到一个数用于指向基因中的一个氨基酸的下标,再在一个与氨基酸序列等长的空白数组中将第二个哈希值累加到空白数组中与第一个哈希值指向的氨基酸相同的下标位置中。
32、进一步,所述编码s160包括:
33、将评估编码s140最后生成的一个与基因氨基酸等长的数组对一个特定的数取模,将取模的结果与氨基酸的密码子关系映射表比对,根据密码子的不同比例以及取模的结果最终确定一个密码子,并利用该密码子替换掉原基因中的对应氨基酸的密码子,最终得到一个新的基因密码子序列。
34、进一步,所述解码s210包括:读入待纠错解码的长dna序列后,根据编码时强制转换的基因起始密码子与终止密码子或者利用内部的纠错编码从长dna序列中找出存储有编码信息的基因序列,并得到基因序列的密码子序列以及氨基酸序列和氨基酸序列的长度。
35、进一步,所述解码s220包括:获得基因的氨基酸序列的长度以后,根据与编码时相同的最优哥隆尺选取规则确定一个最优哥隆尺,并根据最优哥隆尺构造一个相同结构的滑动窗口;
36、根据滑动窗口对待纠错解码的长dna序列进行与编码时完全相同扫描哈希,得到一个新的与基因的氨基酸序列等长的哈希值数组,并利用与编码时相同的密码子映射关系重新映射选择得到一个新的基因密码子序列。
37、进一步,所述解码s280包括:将对待纠错解码的新的哈希值序列重新映射得到的密码子序列与待评估的序列中的基因密码子序列进行比对,若相同氨基酸对应位置的密码子不相同,则将该位置做一个标记,记为一个witness,对所有的密码子序列比对完之后,标记出所有的witness。
38、进一步,所述解码s290包括:对待纠错的dna序列再次使用哥隆尺滑动窗口进行哈希,滑动窗口每滑动一次,对滑动窗口刻度指向的子序列进行哈希,产生的第一个哈希值用于映射到基因的某一个氨基酸,若该氨基酸处被标记为了witness,则对滑动窗口刻度指向的碱基进行打分。初始时每个碱基的分数都为0,打分则是在每个碱基的原基础上分数加1。
39、进一步,所述解码s300包括:
40、滑动窗口对待纠错的所有序列都扫描哈希结束之后,则会得到序列中所有碱基的打分情况,则会得到一个打分情况的数组。
41、进一步,所述解码s310包括:对打分数组进行排序,选则数组中得分最高的前若干个碱基,表示这些碱基都是可疑程度比较高的,对这些碱基进行穷举纠错;在某个碱基处,每进行一次尝试纠错后,就对整个待纠错的原序列重新进行一次滑动窗口的扫描哈希,重新计算得到witness的数量;在所有的错误类型都穷举试探完之后,选择所有类型中witness总数最小的那个方案最为本轮的纠错方案。
42、进一步,所述解码s320包括:
43、重复纠错过程,直到最后计算得到的序列中的witness为0,即表示纠错成功;
44、若序列中碱基的错误数量超过纠错上限时,也可能无法纠正全部错误,返回纠错解码失败。
45、第二方面,本发明提供一种基于哥隆尺的基因序列纠错系统,包括:
46、编码模块和解码模块;
47、其中,编码模块包括:
48、模块b1:确定存储编码信息的基因,以及根据选择的基因的氨基酸的长度选择一个最优哥隆尺;
49、模块b2:将待编码保护的长dna序列与选择的基因输入进编码模块中进行编码;
50、模块b3:将存储了待编码长dna序列信息的基因序列与被保护的长dna序列进行拼接,输出带有纠错保护编码的长dna序列;
51、模块b4:将待纠错解码的长dna序列输入进解码模块,评估解码根据基因内存储的长dna信息以及重新编码生成的前后信息进行比对,利用打分与穷举纠错对长dna序列中的错误碱基进行纠错;
52、解码模块包括:
53、模块j1:哥隆尺滑动窗口提取信息;
54、模块j2:哈希函数映射;
55、模块j3:纠错信息嵌入基因;
56、模块j4:扫描校验;
57、模块j5:对比打分;
58、模块j6:会诊与纠错尝试。
59、第三方面,本发明提供一种电子设备,包括存储器、处理器及存储在所述存储器上并可在所述处理器上运行的计算机程序,所述处理器执行所述程序时实现第一方面所述基于哥隆尺的基因序列纠错算法。
60、本发明的有益效果如下:
61、本发明作为一种针对基因序列的纠错算法,与传统纠错码相比,覆盖的纠错类型更全面,包括但不限于替换错、插入错、删除错以及基因片段丢失(大量连续增删错),而传统纠错码仅有纠正替换错的能力。在编码阶段,将基因序列中碱基符号抽象为一个数据位,整个基因抽象为一个数组,对基因序列数组构造纠错码并存储在另一段dna中,在基因序列需要解码提取信息前,从dna中提取纠错码信息针对基因序列进行校验纠错:若无错则校验成功;若有错则尝试进行纠错,当错误程度小于纠错码纠错能力时,纠错码能成功纠错还原基因序列;当错误程度远大于纠错码纠错能力时,无法成功纠错但能够指示并报告错误。
62、本发明在针对基因序列的编码阶段,构造纠错码时对基因序列长度不做限制,基因序列可以任意长,覆盖世界上已知的所有基因组测序序列。
63、本发明在针对基因序列的解码阶段,会提取纠错码作为进一步解码的指示,覆盖所有的错误类型,做出错误指示甚至能完全恢复基因序列。
64、本发明所述方法能够处理世界上已测序的任意规模的基因组序列,且能应对混合错误。该方法面对长度为450万个碱基的大肠杆菌基因组序列,也具有较优的校验纠错能力。该方法利用氨基酸与密码子映射关系中的信息冗余进行纠错信息嵌入,即不影响生物活性,也不会产生新的碱基序列,为未来dna存储纠错的生物普适性提供了一种新的研究思路。
本文地址:https://www.jishuxx.com/zhuanli/20240731/185394.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。