一种抗菌肽判别器的获取方法
- 国知局
- 2024-08-05 11:55:19
本发明涉及抗菌肽识别领域,尤其涉及一种抗菌肽判别器的获取方法。
背景技术:
1、抗菌肽是多细胞生物为抵抗外界入侵的第一道防线而产生的分子,存在于从原核生物到人类生物体的各种生物中,包括哺乳动物、两栖动物、昆虫、微生物和植物等。抗菌肽通常带正电荷,由10到50个氨基酸残基组成,具有广泛的活性,可以直接杀死细菌、真菌、病毒甚至癌细胞。基于机器学习的抗菌肽识别综合运用数学理论、计算机技术和生物信息学,通过某种方法提取出抗菌肽的序列模式、结构特征以及生化特性等信息,再使用机器学习算法从已有的数据和经验中进行学习,通过推理、归纳或者模型拟合的方式将学习到的规律应用于判断未知序列是否是抗菌肽。机器学习的发展为抗菌肽识别提供了新的工具,研究人员开始结合深度学习的方法来识别抗菌肽,受自然语言处理启发,hamid等人使用跳字模型在蛋白质数据库上训练了静态的氨基酸向量,并使用循环神经网络来获取序列级别的信息,进而将建模结果用于识别抗菌肽。另外,研究者们期望利用大规模肽数据上训练的模型能够学习到训练数据集中隐含的生化性质、二三级结构和功能活性,并将训练得到的模型像语言模型那样用于各类肽相关的下游任务,如肽功能预测、肽结构预测和特定功能肽的生成,这类预训练模型被称为蛋白质语言模型。在大规模无标注文本语料库上训练的语言模型是目前自然语言处理广泛使用的基础工作,能够学习通用的语法和语义等信息,这些信息在下游任务中效果显著。蛋白质与文本具有相似的特性,基于预训练语言模型的思想在无标注蛋白质数据上训练的蛋白质语言模型在蛋白质研究中具有广阔前景。本发明从相关的蛋白质数据库中收集抗菌肽和非抗菌肽构建抗菌肽数据集,为抗菌肽的智能识别提供了一种新思路。
技术实现思路
1、为了实现对抗菌肽的准确与智能识别,本发明提出了一种抗菌肽判别器的获取方法,所述抗菌肽判别器包括输入词嵌入层、目标模型主体与输出层;所述输入词嵌入层用于利用最优分词策略获取肽序列对应的固定维度向量;所述模型主体用于对固定维度向量进行特征提取并输出潜在空间特征向量;所述输出层用于对模型主体输出的潜在空间特征向量进行线性变换、归一化与激活函数的处理,并利用处理后的潜在空间特征向量预测输入的肽序列为抗菌肽的概率,所述潜在空间特征向量表示对输入的肽序列建模的潜在特征空间;所述获取方法包括:
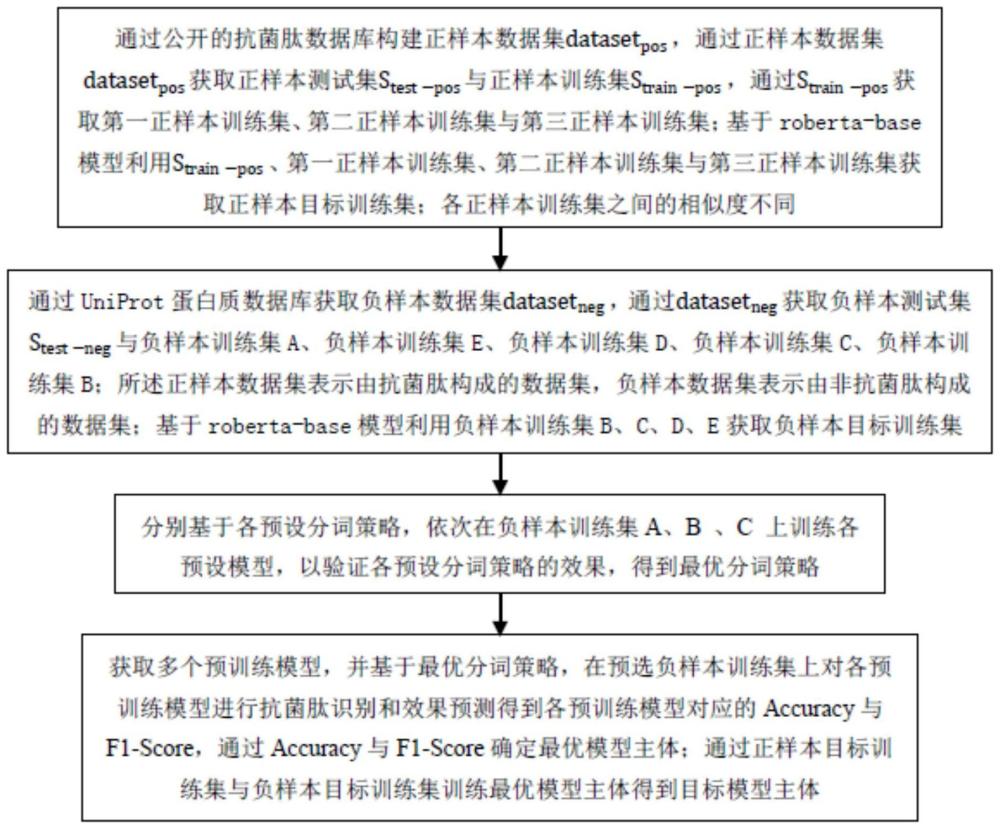
2、通过公开的抗菌肽数据库构建正样本数据集datasetpos,通过正样本数据集datasetpos获取正样本测试集stest-pos与正样本训练集strain-pos,通过strain-pos获取第一正样本训练集、第二正样本训练集与第三正样本训练集;基于roberta-base模型利用strain-pos、第一正样本训练集、第二正样本训练集与第三正样本训练集获取正样本目标训练集;各正样本训练集之间的相似度不同;
3、通过uniprot蛋白质数据库获取负样本数据集datasetneg,通过datasetneg获取负样本测试集stest-neg与负样本训练集a、负样本训练集e、负样本训练集d、负样本训练集c、负样本训练集b;所述正样本数据集表示由抗菌肽构成的数据集,负样本数据集表示由非抗菌肽构成的数据集;基于roberta-base模型利用负样本训练集b、c、d、e获取负样本目标训练集;
4、分别基于各预设分词策略,依次在负样本训练集a、b、c上训练各预设模型,以验证各预设分词策略的效果,得到最优分词策略;
5、获取多个预训练模型,并基于最优分词策略,在预选负样本训练集上对各预训练模型进行抗菌肽识别和效果预测得到各预训练模型对应的accuracy与f1-score,通过accuracy与f1-score确定最优模型主体;通过正样本目标训练集与负样本目标训练集训练最优模型主体得到目标模型主体。
6、进一步地,所述通过strain-pos获取第一正样本训练集、第二正样本训练集与第三正样本训练集,具体为:
7、通过cd-hit将冗余度阈值分别设置为0.5、0.7和0.9对strain-pos进行冗余筛除,得到相似度小于50%的第一正样本训练集strain-pos-id50、相似度小于70%的第二正样本训练集strain-pos-id70、相似度小于90%的第三正样本训练集strain-pos-id90。
8、进一步地,所述通过datasetneg获取负样本测试集stest-neg与负样本训练集e、负样本训练集d、负样本训练集c、负样本训练集b,具体为:
9、从datasetneg中按照datasetpos的长度分布抽取了多条非抗菌肽序列作为非抗菌肽数据集a;
10、将非抗菌肽数据集a按照8:2的比例随机划分成负样本训练集a和负样本测试集stest-neg;
11、将stest-neg从datasetneg中剔除后按照随机方式抽取多条非抗菌肽序列作为负样本训练集e;
12、从负样本训练集e中按照0.5的概率抽取序列构建负样本训练集d;
13、从负样本训练集d中按照0.5的概率抽取序列构造负样本训练集c;
14、从负样本训练集c中按照0.34的概率抽取序列构造负样本训练集b。
15、进一步地,所述在负样本训练集a、b和c上验证预设分词策略的效果,具体为:
16、通过正样本训练集strain-pos作为抗菌肽训练集,负样本训练集a、b、c作为非抗菌肽训练集分别对bert-base-cased、bert-base-uncased以及roberta-base进行训练,通过stest-pos与stest-neg进行测评,得到各模型分别在负样本训练集a、b、c上对应的accuracy和f1-score指标,通过accuracy和f1-score指标确定最优分词策略。
17、进一步地,所述输入词嵌入层具体用于根据最优分词策略将肽序列的每一个氨基酸视为一个独立的词,然后对每一个氨基酸进行嵌入得到一个固定维度的向量输入到模型主体中,所述向量中包括每个氨基酸的原始信息与位置关系。
18、进一步地,所述正样本目标训练集的具体获取方法为:
19、分别通过strain-pos、第一正样本训练集、第二正样本训练集与第三正样本训练集训练roberta-base模型,得到各训练集对应的accuracy与f1-score,通过accuracy与f1-score获取正样本目标训练集。
20、进一步地,所述负样本目标训练集的具体获取方法为:
21、分别通过负样本训练集b、c、d、e训练roberta-base模型,得到各训练集对应的accuracy与f1-score,通过accuracy与f1-score获取负样本目标训练集。
22、进一步地,所述预训练模型包括bert-base-cased、bert-large-cased、roberta-base、roberta-large、biobert-base-cased、protbert、esm2-t12-35m-ur50d与esm2-t30-150m-ur50d。
23、进一步地,所述预设模型包括:bert-base-cased、bert-base-uncased以及roberta-base模型;所述公开的抗菌肽数据库包括:apd3、camp和lamp2。
24、与现有技术相比,本发明至少含有以下有益效果:
25、(1)本发明通过公开的抗菌肽数据库构建多个相似度不同的正样本训练集,并基于roberta-base模型利用通过各正样本训练集获取正样本目标训练集,通过uniprot蛋白质数据库获取多个负样本训练集,并基于roberta-base模型利用负样本训练集b、c、d、e获取负样本目标训练集;通过多个负样本训练集获取最优分词策略;并基于最优分词策略,在预选负样本训练集上对各预训练模型进行抗菌肽识别和效果预测得到各预训练模型对应的accuracy与f1-score,通过accuracy与f1-score确定最优模型主体;通过正样本目标训练集与负样本目标训练集训练最优模型主体得到目标模型主体,通过最优分词策略、目标模型主体与输出层构建出抗菌肽判别器,通过抗菌肽判别器实现了对抗菌肽的准确与智能识别;
26、(2)为了验证筛除相似度数据对抗菌肽判别器的负面影响或者对抗菌肽判别器准确性的影响,本发明构建了三个数据集strain-pos-id50、strain-pos-id70和strain-pos-id90,分别表示仅保留相似度小于50%、相似度小于70%和相似度小于90%的抗菌肽用于构建正样本训练集,通过此三个数据集训练roberta-base模型,得到各训练集对应的accuracy与f1-score,通过accuracy与f1-score获取正样本目标训练集,通过正样本目标训练集训练抗菌肽判别器的模型主体,进而提高了抗菌肽判别器的识别准确性;
27、(3)为了验证正负样本比例对抗菌肽判别器的影响,本发明构建了相对strain-pos大小不同倍数的负样本训练集b、c和d,通过负样本训练集b、c、d、e训练roberta-base模型,得到各训练集对应的accuracy与f1-score,通过accuracy与f1-score获取负样本目标训练集,通过负样本目标训练集训练抗菌肽判别器的模型主体,进一步提高了抗菌肽判别器的识别准确性。
本文地址:https://www.jishuxx.com/zhuanli/20240802/260114.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。