基于自适应算术编码的DNA数据存储方法
- 国知局
- 2024-07-12 10:31:19
本发明涉及数据存储方法,尤其涉及一种基于自适应算术编码的dna数据存储方法。
背景技术:
1、随着科学技术和信息技术的飞速发展,全球的数据信息总量正在以惊人的速度增长。据估计,到2040年全球信息总量将达到3×1024bits。而目前的存储介质在面对日益增长的数据,有许多无法解决的问题,如存储介质的密度较低加剧了土地资源的过度使用和能耗过大,存储介质较短的使用年限增加了数据迁移和维护成本等等。因此寻找新的存储介质,发展新的存储技术刻不容缓。
2、dna是生物体携带遗传信息的物质,由腺嘌呤(a)、鸟嘌呤(g)、胸腺嘧啶(t)和胞嘧啶(c)构成,具有存储密度高、存储时间长和存储文件稳定、易维护等特点。在传统存储介质无法存储海量数据时,dna存储带来了希望。dna存储技术是一种将数字信息以基因序列的形式存储在dna分子中的新型信息存储方法。dna存储技术全流程一般可分为6个主要步骤,包括编码、合成、保存、获取、测序、解码,其中编码、解码不仅可以保证dna存储密度高、生化操作顺利和数据准确恢复,还从计算机程序角度较低了dna存储的成本,推动了dna存储技术的发展,是dna存储技术重要的一环。
3、在dna存储的早期,由于读写长dna序列的困难,这时期的dna存储编解码都是对小文件进行实验。随着dna合成和测序技术发展,从2012年church团队首次成功实现较大数据的dna存储后,越来越多的dna存储编解码方法被提出。目前发表的编码算法大致可以分成两类,第一类为在基本映射关系基础上没有增加筛选过滤步骤的编码算法,第二类为在基本映射关系基础上增加筛选过滤步骤的编码算法。第一类有church编码算法、goldman编码算法、grass编码算法、blawat编码算法等。这类编码算法是在考虑单碱基重复或者其他要求下确定了映射规则,将数据流直接编码成dna序列。第二类包含dna-fountain编码算法、yin-yang双编码算法、raptor编码算法等,该类算法均采用四进制模型(00,01,10,11编码a,t,c,g)进行编码,在编码过程中增加了筛选步骤,控制碱基序列gc含量、均聚物长度。
4、现在dna数据存储中编解码的研究出发点主要是集中在提升编码密度、生化约束和数据的稳健性。第一类编码算法利用固定的映射关系进行编码,编码信息密度较低,生化约束条件没有很好的控制。第二类编码算法的编码密度、生化约束、数据稳健性都比较好,但该类编码算法编码过程引入了筛选过程,增加了编码代价,同时dna存储潜力没有完全利用。
5、在dna数据存储过程中,主要包含压缩-引入纠错码-映射编码碱基三个过程。压缩是为了减少存储数据需要的空间,提高编码密度。在压缩中主要用霍夫曼编码和喷泉码,其中霍夫曼编码是dna压缩过程中的主要方法。霍夫曼编码是无损压缩,可以有效的对编码文件进行压缩,但是霍夫曼编码采用整数进行编码,在精度上存在缺陷,无法逼近香农极限。而自适应算术编码相较于霍夫曼编码没有精度缺陷,压缩编码后的码长更加接近信息熵的极限,在处理动态数据时具有适应性和实时性。
技术实现思路
1、本发明所要解决的技术问题是如何提供一种压缩效率高、可保证dna序列生化约束和数据准确性的基于自适应算术编码的dna数据存储方法,同时该方法不仅可以应用于静态数据存储,还可以存储实时变化的动态数据。
2、为解决上述技术问题,本发明所采取的技术方案是:一种基于自适应算术编码的dna数据存储方法,包括:dna编码步骤和dna解码步骤,所述dna编码步骤包括:
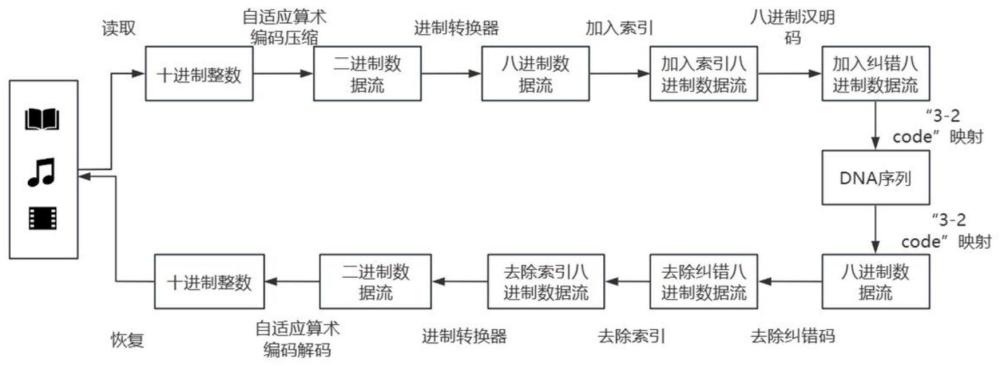
3、首先将需要存储的文件按照每个字节(byte)读取为无符号整数,每个字节将被解释为范围在0到255之间的整数,对0到255整数预处理后采用自适应算术编码进行压缩得到二进数据流,二进制数据流经过进制转换器转变为八进制数据流;
4、然后给每段八进制数据流添加索引,对加入索引的八进制数据流引入八进制汉明纠错码;
5、最后对加入纠错的八进制数据流按照“3-2code”进行映射。
6、dna解码是dna编码的逆过程,是将dna序列恢复成数字信息的过程;解码过程中,对dna序列以“3-2code”映射关系解码获得八进制数据流,然后将八进制数据流先去除八进制汉明码,再去除索引,把所得的八进制数据流经过进制转换器转为二进制数据流,最后通过自适应算术编码将二进制数据流解码成文件字节的十进制整数,进一步恢复成文件内容。
7、进一步的技术方案在于,所述采用自适应算术编码进行压缩的方法包括如下步骤:
8、s1:对不同文件进行预处理操作,获得待编码的数据流;
9、s2:从待编码的数据流中读取数字;
10、s3:获取该数字对应阶数的上下文,计算此阶数下的概率值范围(left_pt,right_pt);
11、s4:判断该概率值范围的right_pt是否为0
12、s5:如果right_pt=0,ppm算法输出逃逸符号,并从当前的上下文阶开始,逐步降低阶数重新计算概率范围,当right_pt不等于0或者阶数等于-1停止;
13、s6:如果right_pt不等于0,根据概率值范围和总区间范围span,用如下公式计算出区间范围,然后进行算术编码压缩:
14、
15、s7:更新上下文树的节点和频率,如果该数字第一次出现,则创建新的节点,将其加入当前上下文树各个节点中,否则在上下文树中更新其频率;
16、s8:更新上下文表,上下文表的长度不能大于阶数5;
17、s9:重复执行s2-s8,直到处理完数据流中的每个符号;
18、s10:当所有符号都被处理完毕时,完成编码过程,并生成压缩后的数据。
19、进一步的技术方案在于,所述八进制汉明码纠错的方法包括如下步骤:
20、dna编码阶段
21、(1)添加校验位
22、将添加索引后的八进制数据流按照信息码元的长度计算校验位的数量和位置并添加校验位,设计(n,k)八进制汉明码(n为传输码元的总长度,k为信息码元的长度);
23、(2)添加纠错码
24、通过矩阵运算计算校验位的值,将生成矩阵和信息码元相乘计算出校验位原始值,然后经过“取模8”和“8相减”,得到八进制的校验值,每个校验位对应一定数量的数据位,具有检测和纠正错误的能力;
25、(3)编码碱基
26、将添加纠错码后的八进制数据流根据“3-2code”映射,得到碱基序列;
27、dna解码阶段
28、(1)碱基解码
29、碱基序列通过“3-2code”映射,得到含有八进制纠错码的八进制数据流;
30、(2)检验
31、根据编码设计的(n,k)八进制汉明码,选取长度为n的传输码元与监督矩阵相乘,计算出校正子,通过校正子判断数据是否出现错误;
32、(3)纠错
33、如果检测到错误,通过校正子计算出错误发生的位置(该位置的数字称为误码),用误码和校正子非零数相减得到正确的码元。相减时,当误码小于校正子非零数,误码加8再减。
34、进一步的技术方案在于,所述“3-2code”映射方案包括:
35、将添加八进制纠错码的数据流映射成碱基组合,其本质为:每三位二进制数映射编码为两个碱基,两个碱基设计必须是在a、t和g、c中任取其一组合,得到ac、ag、ca、ga、tc、tg、ct、gt八种组合;每个碱基组合的c或g含量为50%,随着编码的进行,组合起来的碱基序列中cg含量可以稳定保持在50%,均聚物的长度不会大于2。
36、采用上述技术方案所产生的有益效果在于:所述方法主要包括压缩、纠错和映射三部分,在压缩中采用了精度更高的自适应算术编码,提高了压缩效率,可以压缩静态和动态的数据;在纠错中,提出了八进制汉明纠错码;在映射中,设计了可以让gc含量稳定保持在50%、均聚物长度不超过2的“3-2code”映射方案。最后将文本、图片、音频不同格式的文件分别进行了编码实验。实验结果表明:基于自适应算术编码的dna存储编码方案每个碱基的平均编码密度为2.99bits,gc含量和均聚物长度符合预期;在纠错时,根据信道质量适当调整汉明码中信息码元的长度,可以保证数据的准确性。
本文地址:https://www.jishuxx.com/zhuanli/20240614/87459.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。