鉴定菊花光反应周期长短的KASP标记、其开发方法及应用
- 国知局
- 2024-06-20 10:48:35
本发明涉及一种鉴定菊花光反应周期长短的kasp标记、其开发方法及应用,属于植物分子标记辅助育种领域。
背景技术:
1、观赏植物的开花期是构成其观赏价值的重要性状之一,开花时间会直接影响其在市场上的价格。菊花(chrysanthemum morifolium ramat)是典型的短日照植物,花期较单一,多集中于秋季,限制了菊花的商业发展。在生产中,需要通过人工补光或者遮光来调控菊花开花,极大增加了生产成本。光反应周期是指植株感受到短日照刺激至达到初花期所需要的天数,不同品种对光周期的敏感度存在着明显差异。光反应周期过长的品种会增加生产和管理成本,而光反应周期敏感品种的生育期较短,有助于控制生产成本,还可以提高种植茬数,从而达到节本增效的目的。因此,培育短光反应周期品种是菊花育种工作的重要目标之一。
2、菊花是一种高度杂合的多倍体植物,且自交不亲和。因此,传统杂交育种和诱变育种仍是培育现代菊花品种的有力手段。但是传统育种依赖育种家的经验将亲本的有利基因进行随机组合,效率较低。随着分子生物学技术的发展,分子标记辅助选择(marker-assisted selection,mas)育种已成为育种的重要手段,可以针对个别性状进行精确改良,效率较高。目前研究大多是针对菊花自然花期(即定植到各个开花时期的天数),而光反应周期研究较少。袁储聪等(2023)发现菊花光反应周期呈现连续性变化的正态分布,说明光反应周期可能属于多基因控制的数量性状。植株从感受到短日照刺激后,直到初开才能判断品种的光反应周期特性,耗时长。因此亟需建立菊花光反应周期的mas育种体系,实现短生育期优良品种的早期选择。
3、菊花mas研究目前处于初步阶段。su等(2019)总结了近年来基于分子标记的菊花花序、株型、花期及抗逆等性状的遗传研究进展。但前期研究主要基于srap、aflp、rapd和ssr等传统分子标记,数量有限且难以高通量分型,严重影响了定位精度与效率。与传统分子标记相比,单核苷酸多态性(single nucleotide polymorphism,snp)和小片段插入缺失(insertion and deletion,indel)标记具有分布广、遗传稳定性好、二等位基因型等特点,更容易实现高通量和自动化检测。但是对于菊花(~8.5gb)等基因组庞大的物种来说,snp和indel测序成本高,难以实现大规模群体基因分型。kasp分型技术,即竞争性等位基因特异性pcr(kompetitive allele specific pcr),是一种新的snp和indel自动化检测方法。kasp标记能够在更大规模个体的基因组dna样本中准确地判断特定位点的snp和indel,且耗时短、成本低,应用前景广阔。bsa-seq是将二代测序技术(next-generationsequencing,ngs)与集群分离分析法(bulked segregant analysis,bsa)结合的一种快速定位与目标性状相关联的基因或位点方法(汪泽民等,2023)。近年来,bsa-seq和kasp分型联合分析策略已经广泛应用于大豆、甜瓜、甘蔗和小麦等经济作物的产量、抗逆等农艺性状的基因精细定位和分子标记辅助育种(wu et al.,2023)。然而,目前在菊花光反应周期的研究中尚未见报道。
技术实现思路
1、发明目的:本发明的第一目的是提供了一种鉴定菊花光反应周期长短的kasp标记。本发明的第二目的是提供上述kasp标记的开发方法及应用。
2、技术方案:本发明提供了一种鉴定菊花光反应周期长短的kasp标记,所述kasp标记包括如下的一种或两种:
3、位于菊花9号染色体上第205233550位的snp变异位点,该位点由t突变为a命名为kasp-chr9_205233550-t/a;位于菊花10号染色体上第20239079位的snp变异位点,该位点由a突变为g,命名为kasp-chr10_20239079-a/g。
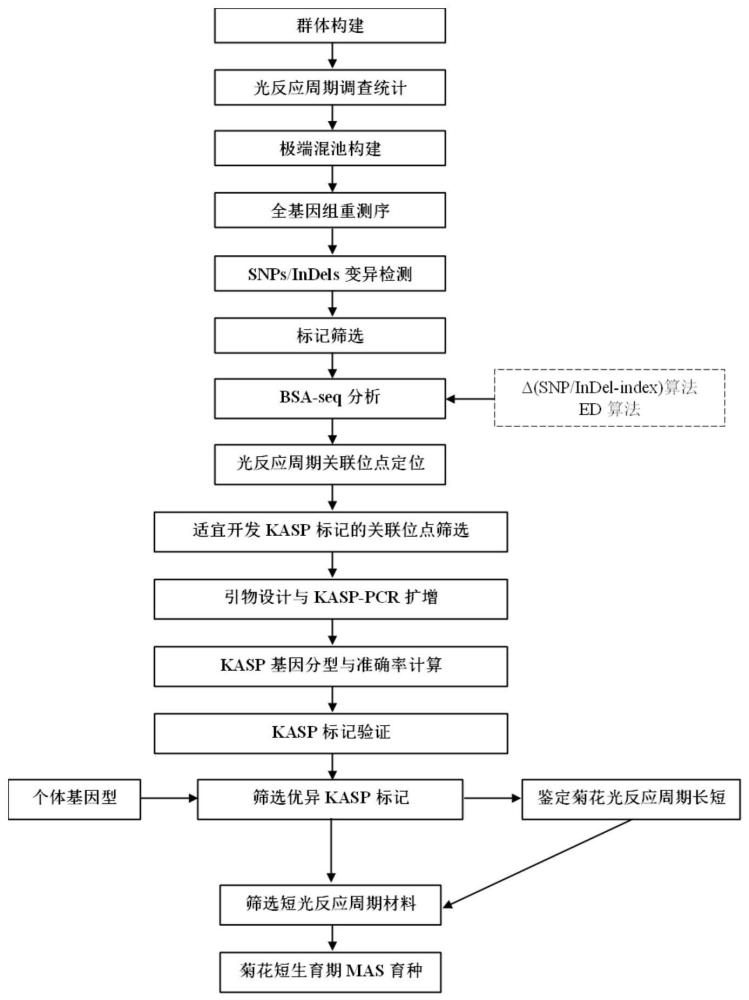
4、本发明还提供了一种基于bsa-seq技术开发上述的鉴定菊花光反应周期长短的kasp标记的方法,(1)以光反应周期长短相差至少14天的两个切花菊品种为亲本,进行杂交获得f1分离群体,对其进行光反应周期表型鉴定;按照生产标准,植株达到20~25cm高时开始进行补光;所述光反应周期为从停止补光到花朵初开期的天数,初开期为40%~50%的花序呈半开放状态的时期;
5、(2)从f1子代中筛选多个短光反应周期极端株系及长光反应周期极端株系,提取亲本和所有极端株系的dna,构建子代极端混池早花池和晚花池,对亲本及两个混池进行全基因组重测序;
6、(3)将测序数据质控后得到的clean reads比对到菊花参考基因组上,进行变异检测,并根据变异位点质量和样本基因型进行过滤;;
7、(4)利用|δ(snp/indel-index)|和欧式距离ed两种算法对步骤(3)过滤后得到的变异位点进行bsa-seq分析,获得菊花光反应周期显著关联位点;
8、(5)采用滑窗的方法对|δ(snp/indel-index)|及ed2进行拟合,取窗口内的平均值作为拟合后的值,按照窗口拟合值从大到小对窗口排序,以top 0.5%处的拟合值作为关联阈值筛选显著窗口,将两种算法获得的显著窗口取交集。
9、(6)选取步骤(5)中获得的交集窗口中|δ(snp/indel-index)|>0.8的变异位点,利用“bedtools getfasta”程序提取每个位点上下200bp序列,筛选符合标准的序列作为适宜开发kasp标记的菊花光反应周期关联位点;
10、(7)根据步骤(6)中筛选到的变异位点设计相应的kasp扩增引物,在该开发群体中进行pcr扩增;
11、(8)针对步骤(7)中开发的kasp标记中分型清晰、阴性对照样本无特异性扩增的kasp标记进行准确率计算,在不考虑杂合位点情况下,将准确率>80%的kasp标记在自然群体中进行验证;当kasp标记在自然群体中的准确率达到80%,则可确定该kasp标记可用于早期鉴定菊花光反应周期长短。
12、其中,步骤(1)中所述的母本为长光反应周期菊花品种‘南农庐火’,父本为短光反应周期菊花品种‘迷你黄’。
13、其中,步骤(1)中所述补光天数为40~50d。
14、其中,步骤(2)中所述用于构建每个极端混池的株系数目为f1子代中该群体大小的10%~20%。
15、其中,步骤(3)中所述的菊花参考基因组为‘钟山紫桂’。
16、其中,步骤(3)的具体步骤包括:对亲本和两个极端混池的原始测序数据去掉接头和低质量序列,获得有效测序数据,使用bwa软件比对到菊花参考基因组上,对结果进行质控,再使用gatk软件检测变异位点,过滤掉低质量变异结果用于下一步分析。
17、其中,snps和indels变异位点的过滤条件分别为“qd<2.0||fs>60.0||mq<40.0||mqranksum<-12.5||readposranksum<-8.0”和“qd<2.0||fs>200.0||sor>10.0||mqranksum<-12.5||readposranksum<-8.0”。
18、其中,步骤(4)中筛选用于bsa-seq分析的标记,过滤标准是:1)亲本分离形式符合f1群体,即nn×np、lm×ll、hk×hk;2)亲本和子代混池的基因型无缺失;3)去除亲本与子代相反表型的纯合相同位点;4)四个样本的测序深度要大于10×且小于500×;5)至少有一个子代池的snp/indel-index大于0.3;6)至少有一个子代池的snp/indel-index小于0.7。
19、其中,步骤(5)中滑窗时使用的窗口大小为500kb,步长大小为50kb,窗口内的snp数目大于等于20时,该窗口为有效窗口,snp数目不足时,该窗口的结果并入下一个窗口。
20、其中,步骤(6)中适合开发kasp标记的变异位点筛选标准是:1)上下游200bp序列中除目标snp或indel位点外,其它变异位点数目不超过5个;2)该片段在菊花基因组中的相似序列个数不超过3个,即blast比对分析时同时满足序列比对一致性百分比identity>83%和覆盖率coverage>80%时得到的序列个数越少越好。
21、其中,步骤(7)中所述引物包含两条竞争性前引物和一条通用后引物,前引物3’末端碱基为snp或indel位点等位变异碱基,前引物5’端分别加上fam和vic荧光序列标签。
22、其中,步骤(7)中kasp-pcr扩增体系为:2×kasp mastermix 0.8μl、primermix0.024μl(前引物f1浓度12μm、前引物f2浓度12μm、通用后引物浓度30μm)、ddh2o0.776μl。其中至少设置一个无dna模板的阴性对照(ntc)。
23、其中,步骤(7)中kasp-pcr扩增程序为:94℃预变性15min;94℃变性20s,55~65℃退火延伸60s,每个循环退火延伸温度降低1.0℃,共计10个循环;95℃变性20s,55℃退火延伸60s,20个循环。
24、本发明还公开了一种上述的kasp标记在鉴定菊花光反应周期长短中的应用,其特征在于,对于标记kasp-chr9_205233550-t/a,若检测基因型为t:t,则为光反应周期短;若检测基因型为a:a,则为光反应周期长;若检测基因型为杂合a:t,则光反应周期大概率长,可结合田间表型进一步确认;对于标记kasp-chr10_20239079-a/g,若检测基因型为a:a,则为光反应周期短;若检测基因型为g:g或g:a,则光反应周期大概率长,可结合田间表型进一步确认;当kasp-chr9_205233550-t/a和kasp-chr10_20239079-a/g标记结合判断菊花品种光反应周期长短时,若检测基因型为t:t-a:a,则光反应周期短;若检测基因型为a:a-g:g或a:a-g:a或a:t-g:g或a:t-g:a,则光反应周期长;若为其它基因型可结合田间观察进一步确认。
25、本发明还公开了一种早期鉴定菊花光反应周期的方法,包括以下步骤:
26、(1)在苗期提取待测样品的dna,作为扩增模板;
27、(2)对上述的kasp标记进行pcr扩增和基因分型,当检测kasp标记为kasp-chr9_205233550-t/a时,若检测基因型为t:t,则光反应周期短,若为a:a,则光反应周期长,若为a:t,则光反应周期大概率长,需结合田间表型进一步确定;当检测kasp标记为kasp-chr10_20239079-a/g时,若检测基因型为a:a,则光反应周期短;若检测基因型为g:g或g:a,光反应周期大概率长,可结合田间表型进一步确认;当检测kasp标记为kasp-chr9_205233550-t/a和kasp-chr10_20239079-a/g时,若检测基因型为t:t-a:a,则光反应周期短;若检测基因型为a:a-g:g或a:a-g:a或a:t-g:g或a:t-g:a,则光反应周期长;若为其它基因型可结合田间观察进一步确认。
28、其中,步骤(2)中用于扩增kasp标记kasp-chr9_205233550-t/a的扩增引物包括核苷酸序列如seq id no:1~2所示的两条竞争性前引物、和核苷酸序列如seq id no:3所示的通用后引物;用于扩增kasp标记kasp-chr10_20239079-a/g的扩增引物包括核苷酸序列如seq id no:4~5所示的两条竞争性前引物和核苷酸序列如seq id no:6所示的通用后引物。
29、有益效果:与现有技术相比,本发明具有如下突出的显著优点:本发明通过基于全基因重测序的bsa-seq技术快速定位到与光反应周期相关的变异位点,将筛选到的snp关联位点转化成kasp标记,相较于传统基于普通pcr扩增的分子标记,kasp标记不需要用到凝胶电泳,可以实现快速、高通量及平台化检测;本发明开发的snp位点chr9_205233550和chr10_20239079,可单独或组合使用,该标记组鉴定菊花品种光反应周期长短的准确率能达到82.67%。不需要通过漫长的田间表型调查,就可以实现菊花光反应周期的早期鉴定,即避免了人工主观判断带来的误差及人力物力的浪费,又大大地提高了选择效率。本发明的鉴定方法,可在菊花生长早期快速、准确地对光反应周期进行判断,有利于亲本的选配及后代优异株系的筛选,为菊花光反应周期mas育种体系的建立奠定了基础,对菊花光反应周期研究及其周年生产具有重要意义。
本文地址:https://www.jishuxx.com/zhuanli/20240619/382.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表