一种快速鉴定无乳链球菌及其高毒力株的方法
- 国知局
- 2024-09-11 14:32:29
本发明属于细菌的鉴定方法、深度学习、拉曼光谱,具体涉及一种快速鉴定无乳链球菌及其高毒力株的方法。
背景技术:
1、细菌性脑膜炎是一种由细菌引起的急性中枢神经系统感染性疾病,发病率和死亡率高,影响所有人群,特别是新生儿。新生儿期是一生中发生严重感染的风险最高,也是生存最脆弱的时期。无乳链球菌(streptococcus agalactiae)是一种革兰阳性细菌,又称 b族链球菌(gbs),是导致新生儿细菌性脑膜炎的主要病原体。在分离的临床菌株中,通过对荚膜多糖的血清分型发现,多数病例为血清型iii、 ia、v、ib和ii,其中最主要的是血清型iii。gbs也可以进行多位点序列分型(multilocus sequencetyping,mlst),一个单一的囊状血清型iii gbs克隆,属于高毒力克隆复合体17,几乎只与脑膜炎和迟发型新生儿gbs感染病例相关,通常被称为高毒力克隆,也被称为iii/st17型gbs高毒力株。
2、iii/st17型gbs高毒力株导致的脑膜炎是世界范围内神经残疾的重要原因,对个人、家庭及社会都有相当大的情绪和经济影响。延迟诊断和治疗对个体有严重的、长期的后果,并且新生儿gbs致病与神经发育的关键时期重叠,及时的诊断和治疗尤其重要。
3、传统的病原菌鉴定方法依赖于表型鉴定,主要采用染色法、培养法和简单的生化检验。宏基因组下一代测序技术可检出iii/st17型gbs高毒力株,与传统的基于pcr的方法相比,具有完全无偏性的关键优势,该方法不受事先选择可疑病原体的限制,可以在一次测试中识别潜在的感染因子,对从新生儿身上获得的有限样本尤其重要,但存在价格昂贵、耗时长的问题。
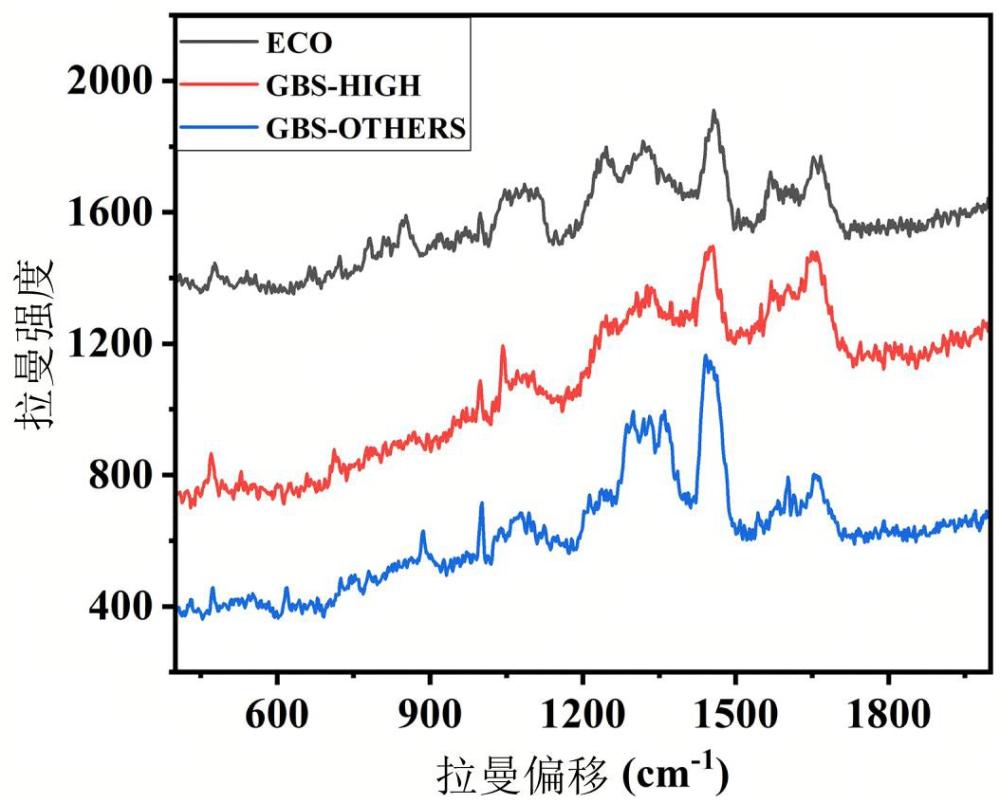
4、拉曼光谱技术是有力、可靠的细菌检测及表征技术。起源于分子基团振动的拉曼图谱被视为生物分子结构及组成的“指纹谱”,已被广泛应用于生物医学、化学检测、矿物质分析等多个领域。深度学习和机器学习的方法能够以数据驱动的方法挖掘数据内隐含的知识,由于其强大的表征能力被广泛应用于图像处理、语音识别、不同物质分类识别等众多领域。本发明提出将拉曼光谱技术与深度学习结合,采集临床菌株的拉曼光谱数据,快速检测gbs及iii/st17高毒力株。
技术实现思路
1、为了克服现有技术中的不足,本发明提供一种基于深度学习结合拉曼光谱的无乳链球菌及高毒力菌株的检测方法。
2、拉曼光谱可以反映化学键的能量,对化合物的结构进行判断。细菌的单细胞拉曼光谱可以反应细菌胞内、细胞膜、细胞壁上的生物大分子及代谢物的成份和浓度,高毒力与低毒力的无乳链球菌的细胞壁结构有显著性的差异,因此其拉曼图谱也分别具有相应的特点,然而这种拉曼图谱的差异依靠肉眼很难观察到,因此本发明利用深度学习的方法对细菌的拉曼光谱进行训练,从而建立可以正确鉴定分类的算法模型。细菌分为三类:gbs-high(无乳链球菌高毒力株)、gbs-others(无乳链球菌非高毒力株)和eco(大肠埃希菌),对训练组数据进行深度学习分析,然后基于分析结果建立深度学习分析模型。最后用验证组数据进行验证,调整模型参数,最后用待测菌的拉曼光谱进行测试。
3、为了达到上述发明目的,解决其技术问题所采用的技术方案如下:
4、一种基于深度学习结合拉曼光谱的无乳链球菌及高毒力菌株的检测方法,包括以下步骤:
5、步骤1:建立拉曼光谱数据库;
6、步骤2:建立gbs及iii/st17高毒力株分型模型;
7、步骤3:评估gbs及iii/st17高毒力株分型模型的性能。
8、步骤1包括以下内容:
9、步骤1.1菌株鉴定:复苏保存的临床分离株至哥伦比亚血琼脂培养基,培养过夜后挑取适量单个菌落,均匀涂抹至maldi靶板的靶点上,形成一薄层。在样品点上覆盖1μlhcca标准溶剂并在室温下自然晾干,将靶板放入基质辅助激光解吸电离飞行时间质谱(maldi-tof-ms),按照自动鉴定流程进行细菌鉴定,当鉴定结果分值≥2.0,且一致性分类为a,确认菌株为gbs;
10、步骤1.2 gbs iii/st17高毒力株的确定:提取经步骤1.1复核后菌株的dna,用biodrop紫外分光光度计检测提取dna的纯度和浓度,吸光度a260 /a280为1.8~2.0,dna浓度>50 ng/μl。采用150 bp双端测序策略进行全基因组测序,利用unicycler进行拼接测序原始短读长序列数据,与相应数据库比对检索,根据分型信息后将gbs分为iii/st17高毒力株和非iii/st17株两组;
11、步骤1.3菌株的处理流程:处理gbs iii/st17高毒力株组、gbs非iii/st17株及对照组菌株,挑取适量菌落洗涤后,用500μl无菌去离子水悬浮于1.5ml离心管中,12000 rpm/min,离心3min,重复洗涤3次,使浊度达到0.5麦氏,最后留200μl备用。
12、步骤1.4拉曼光谱数据库建立:使用激光共聚焦拉曼光谱仪对gbs iii/st17高毒力株组、gbs非iii/st17株组及大肠埃希菌组进行检测,以激发形成不同的拉曼光谱,收集并保存。
13、采集拉曼光谱前样品前处理:吸取样品液各2μl,滴于铝片上,形成直径约2mm的液滴,自然干燥。
14、进行拉曼光谱采集的参数为:激光波长:532 nm;显微镜物镜倍数:100 倍镜(数值孔径 n.a. = 0.9);激光功率:1%;光栅刻线密度:1200 gr/mm;信号采集时间: 50 秒,单次;针孔孔径大小:300 μm;狭缝宽度:200 μm;信号收集频段范围:500 cm-1 -2000 cm-1。
15、步骤2包括以下内容:
16、步骤2.1对步骤1中采集的拉曼光谱数据集进行以下分组:gbs iii/st17高毒力株,gbs 非iii/st17株,大肠埃希菌组。每组以7:1:2的比例随机地划分为训练集、验证集和测试集;
17、步骤2.2数据集处理:对步骤1中采集的拉曼光谱都采用自归一化作为预处理;
18、步骤2.3模型结构设计:基于resnet18残差神经网络架构,将网络中的2d卷积更改为适用于1维拉曼光谱的1d卷积,改进神经网络结构模型,通过仿真实验确定最佳参数;
19、步骤2.4模型训练:以交叉熵作为损失函数,以adam作为训练优化器,在训练集中训练模型的权重,然后通过模型在验证集上的结果选择最优的超参数,确定训练好的模型;
20、步骤2.5模型测试:对于已训练好的卷积神经网络模型,利用测试集测试模型的分类效果。
21、步骤3包括以下内容:
22、用后续收集的gbs iii/st17高毒力株,gbs 非iii/st17株,大肠埃希菌及空白对照组对构建的分型模型进行实验室特异度、准确度评估。用8株的临床菌株对构建的分型模型进行应用评估,评估分型模型的灵敏度、特异度、准确度。以脑脊液培养结果为标准进行评价,分歧结果用宏基因组进行验证。
23、本发明由于采用以上技术方案,使之与现有技术相比,具有以下的优点和积极效果:
24、1.本发明克服了传统的病原菌表型鉴定的方法,创新性地利用激光共聚焦拉曼光谱仪构建gbs及iii/st17高毒力株的分型模型,实现了gbs及iii/st17高毒力株的快速鉴定,具有简单、快速、灵敏度高的优点;
25、2.本发明克服了传统分析方法需要对拉曼光谱进行复杂预处理的缺点,利用卷积神经网络的深度学习方法,创新性地建立gbs及iii/st17高毒力株的拉曼指纹数据库,为iii/st17高毒力株gbs提供高效便捷的快速诊断工具。
本文地址:https://www.jishuxx.com/zhuanli/20240911/291323.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。