基于核酸纳米框架空间三维芯片的自定义长单链DNA的制备方法与信息存储的应用
- 国知局
- 2024-07-31 20:15:23
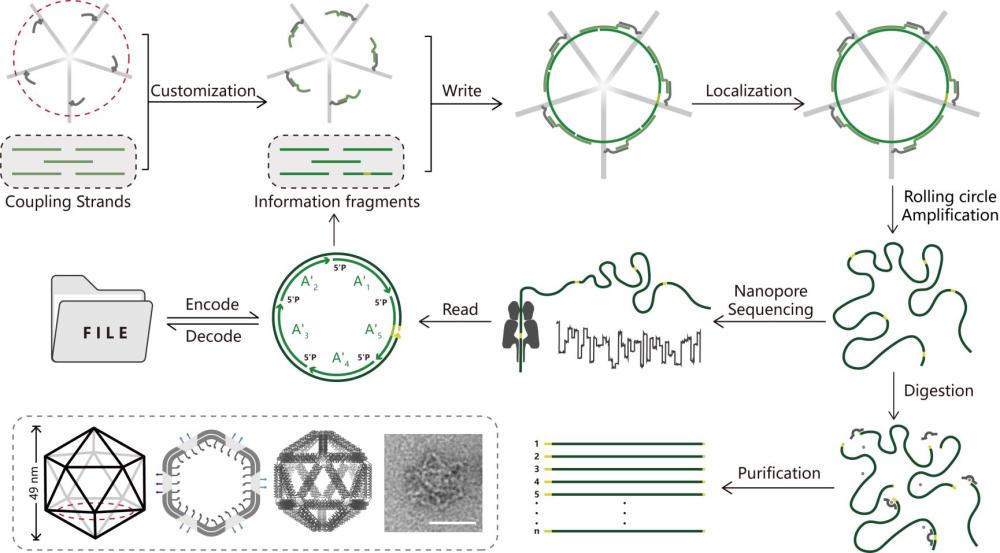
本发明涉及一种基于核酸纳米框架空间三维芯片的自定义长单链dna的制备方法与信息存储的应用,尤其涉及利用dna纳米框架(dna nanoframework,dnf)可寻址性对其进行改造,用于对多条dna链进行有序拼接,制备自定义长线性及环状dna,并应用于信息存储等领域,属于dna纳米。
背景技术:
1、人类文明的发展离不开信息传递与交互,然而,在可预见的未来,基于传统硅基储存介质的储存方式将不可避免地陷入资源枯竭的困境。于是,开发新的储存技术就显得尤为重要。dna作为天然的生命信息储存材料在此方面彰显了其特有的优势,如存储时间长,存储容量大,技术可行性强等。但是,目前以dna为基础的存储技术距离应用仍然面临着两个非常重要的问题:第一,如何获得携载外源信息的任意序列长片段核酸;第二,长片段核酸以何种方式保存从而有利于信息稳定和方便读取。
2、目前,自定义单链dna制备常用的方法主要有寡核苷酸化学合成法、寡核苷酸酶促从头合成法、体外生物酶制备法以及基于噬菌体的生物体内制备法等。虽然这些方法中有些已经非常成熟,但随着对不同长度单链dna的需求日益增加,自定义长单链的合成仍然面临着巨大的挑战。首先,无论是基于dna合成仪的固相合成还是基于末端延长酶促反应的从头合成,合成长度不可避免的受到单步合成效率的影响,在长度达到60-80碱基之后,进一步延长的dna链纯度将逐碱基显著下降,达到120-150碱基的极限后其纯度往往不再满足后续保真读取的需求。其次,基于酶法扩增或噬菌体扩增制备的单链序列需要经历预先拼接和多步骤的分子生物学操作以及多维生物操作,涉及特定酶剪切位置的序列也无法做到完全自定义。在dna存储的具体应用中,越长的核酸片段越可以连续保存大容量的信息,其涉及的索引序列和纠错序列等冗余的占比也越低,从而一定程度上提高存储密度,而上述两个方面的局限性使得dna存储领域对获得任意序列长单链dna(大于150 nt,尽可能长)的技术突破产生巨大需求。
3、另一方面,现有的dna存储方法涉及的核酸链均为30-120nt的线性单链dna,以游离形式保存于水溶液中。暴露的5’和3’末端提高了外切酶降解的概率,由随机碱基互补造成的序列间的相互作用和多组分聚集产物的产生也无法避免。因此,获得环状的,束缚于限域空间中的长单链dna将进一步优化dna存储状态,结合滚环复制反应提供便捷高效的读取方案,并提供更多的应用场景。
4、结构dna纳米技术由纽约大学nadrian seeman教授提出,通过设计在目标结构特定位置上彼此互补配对的若干核酸链,将其自组装成为精准的纳米结构。该技术经历了40年的蓬勃发展,在生物、医学、材料、化学等领域具有极其广泛的应用。2006年dna折纸术的出现降低了dna纳米结构的设计难度,提升了其可编程性,从而大大拓展了dna纳米技术的应用场景。dna折纸术的基本原理是基于碱基互补配对原则,使用上百条称作订书钉链(staple strand)的短链dna将一条称作脚手架链(scaffold strand)的长链dna绑定为预先设计的形状,如正方形、矩形、三角形、星星和笑脸等形状。在随后十余年的发展中,dna折纸术在结构维度、结构曲率、拓扑编织、动态控制以及模块化超组装方面不断取得突破,实现了更精细、更巨大、更复杂结构的组装。利用dna折纸结构的全面可寻址性指导其他分子或材料在纳米尺度下的精准定位与组装从而获得具有全新理化性质的纳米复合结构是该技术的重要应用方向。
5、近年来,dna纳米技术的迅速发展使得在纳米级别实现精确结构设计和合成成为可能,应用dna纳米技术进行信息存储受到越来越广泛的关注。目前,虽然已有利用dna纳米技术进行信息存储的尝试,但是通过将三维dna核酸框架构建为空间芯片从而辅助任意序列长片段环状单链dna的拼接合成,进而实现信息存储与读取的方法未见报道。
技术实现思路
1、本发明的目的是:为了解决自定义长单链制备困难及dna信息存储后读取过程复杂的技术问题,本发明提供了一种dna纳米框架结构作为三维空间芯片,利用芯片内精确的空间位点定义彼此连接的序列顺序,通过设计桥接链将2-30条任意序列的寡核苷酸依序连接并通过酶促连接获得完整的环状单链dna,实现自定义单链的制备,以及信息在三维空间内的存储;基于核酸纳米框架结构自定义拼接单链dna存储信息在模式细菌内进行自主复制,通过对细菌进行简单测序,即可获得目的信息的方法。
2、为了实现上述目的,本发明采取了以下技术方案:
3、本发明的第一方面,提供一种基于核酸纳米框架空间三维芯片的信息存储方法,包括以下步骤:
4、步骤1:利用dna折纸技术构建dna纳米框架结构,并选定用于存储信息的位置序列;
5、步骤2:将待存储信息所对应序列(将待存储的数字信息转码得到的dna序列信息)或待制备自定义长单链dna序列转化为相应互补序列,并分成若干条信息片段,所述信息片段均带有5’端的磷酸化修饰,根据信息片段以及在正二十面体结构内部所选定的位置序列设计耦合序列;若干条所述信息片段的其中一条信息片段上还包含一段dnazyme的互补序列,扩增后形成dnazyme;由于待存储的目的序列和自定义长单链dna序列较长,通过传统的dna合成技术难以实现,而短片段的dna合成技术非常成熟,故将其进行分段化处理,再通过拼接和扩增以此实现目的序列的制备,及信息读取的目的;
6、步骤3:将耦合序列与dna纳米框架结构孵育,使得位置序列与耦合序列的编码部分杂交,以便信息片段后续的锚定;
7、步骤4:将5’磷酸化修饰的信息片段加入体系,再次进行孵育,使得信息片段与耦合序列进行充分的杂交,并保证信息序列之间的上游3’-oh 与下游5’-p彼此邻接,在正二十面体结构内部形成一个带有缺口的圆环;即所述缺口形成于相互连接的信息片段之间;
8、步骤5:加入t4连接酶孵育过夜,消除信息片段之间的缺口,使之成为一个完整的,封闭的信息环状模板,此时,即完成了信息在空间三维芯片内的存储(可在-20℃条件下长期储存)。同时也完成了一种dna信息存储体系的构建。
9、优选地,所述步骤1中的dna纳米框架结构为具有内外拓扑特性的自封闭空间三维立体结构(包括但不限于正二十面体)。
10、优选地,所述步骤2中分成的信息片段长度为60个碱基以下。
11、优选地,所述步骤2中若要进行细菌内部信息自主复制与存储读取,需将细菌质粒进行切口酶切割,外切酶消化,获得质粒的单链化状态,然后对其进行限制性酶切,获得的线性单链作为在细菌内部自主复制的启动片段,将此单链片段与信息片段一起在二十面体内部完成悬挂与拼接。
12、优选地,所述步骤5中若需进行细菌内部存储信息自主复制,需将完成所述存储的dna纳米框架结构转化入细菌内部,在细菌内完成自主复制与信息扩增。
13、优选地,所述细菌为大肠杆菌。
14、本发明的第二方面,提供一种dna信息储存体系,所述dna信息储存体系通过本发明第一方面所述的方法获得。
15、本发明的第三方面,提供一种对本发明第一方面所述的信息存储方法存储的dna信息进行信息读取的方法,或对本发明第二方面所述的dna信息储存体系中存储的dna信息进行信息读取的方法,包括以下方法中的任意一种:
16、方法一:利用细菌复制内部存储信息,然后对细菌进行培养扩增,再提取扩增后的质粒进行测序,即完成存储信息的读取;
17、方法二:所述步骤5之后加入dntp及phi29dna聚合酶进行滚环扩增反应,然后进行纳米孔测序,即完成存储信息的读取。
18、优选地,所述方法一中利用细菌复制内部存储信息,所述细菌在步骤2或步骤5之后导入。
19、本发明的第四方面,提供一种基于核酸纳米框架空间三维芯片的自定义长单链dna的制备方法,包括以下步骤:
20、步骤1:利用dna折纸技术构建dna纳米框架结构,并选定用于存储信息的位置序列;
21、步骤2:将待制备自定义长单链dna序列转化为相应互补序列,并分成若干条信息片段,根据信息片段以及在正二十面体结构内部所选定的位置序列设计耦合序列;所述信息片段均带有5’端的磷酸化修饰,其中一条信息片段上还包含一段dnazyme的互补序列;
22、步骤3:将耦合序列与正二十面体dna框架孵育,使得位置序列与耦合序列的编码部分杂交,以便信息片段后续的锚定;
23、步骤4:将信息片段加入体系,再次进行孵育,使得信息片段与耦合序列进行充分的杂交,在正二十面体结构内部形成一个带有缺口的圆环;所述缺口形成于相互连接的信息片段之间;
24、步骤5:加入t4连接酶孵育过夜,消除信息片段之间的缺口,使之成为一个完整的,封闭的环状模板;
25、步骤6:加入dntp和phi29dna聚合酶进行滚环扩增反应,再将反应体系进行孵育(例如可以置于37℃条件下孵育)使dnazyme形成发卡结构,再加入锌离子(例如具体可以是加入zncl2),孵育(例如可以在37℃下孵育2h)后进行剪切,最后利用page纯化,即制备得到纯的自定义长单链dna序列。
26、本发明的第五方面,提供一种dna结构,将本发明第四方面所述的方法获得的不同的长单链dna进行孵育(例如可以在57℃条件下进行孵育),组装得到新的dna结构。
27、本发明的第六方面,提供一种基于核酸纳米框架空间三维芯片信息库的构建方法,将本发明第二方面所述的dna信息储存体系(即存储有不同信息的正二十面体外部)分别杂交荧光标记序列以及指针序列,杂交结束,相互混合即构建得到信息库。
28、本发明的第七方面,提供一种基于核酸纳米框架空间三维芯片信息库的随机访问方法,包括以下步骤:
29、步骤1:将本发明第二方面所述的dna信息储存体系(存储不同信息的正二十面体外部)分别杂交荧光标记序列以及指针序列,杂交结束,相互混合构建信息库;
30、步骤2:加入修饰有生物素(biotin)的捕获序列(按照现有技术方法,根据指针序列设计的捕获序列,其含有生物素修饰)与信息库杂交;
31、步骤3:加入修饰有链霉亲和素的磁珠(捕获磁珠),将信息库与磁珠混合,震荡孵育(孵育时间可以是1.5h),使含有目标信息的二十面体结构充分与磁珠相结合,随后回收信息库(即溶液部分),清洗三遍;
32、步骤4:通过链置换的原理,设计能够与指针序列进行链置换的序列,将其加入到磁珠体系中,震荡孵育(孵育时间可以是2h),实现正二十面体结构的释放,即回收访问信息。
33、本发明与现有技术相比,具有如下有益效果:
34、(1)本发明提供了一种dna框架结构制备的方法,与现有的信息存储的方法相比,具有设计可控性,精确可寻址性等优势;
35、(2)上述dna框架结构为具有内外拓扑特性的自封闭空间三维立体结构,具有强大的表面可修饰性和内部搭载能力,因此,可以实现内部多条信息序列的顺序定向锚定和高效自定义单链dna合成,同时还可以利用外表面进行一定的修饰来进行存储信息的筛选及分类;
36、(3)经过实验,经过内部编码后的dna框架具有高度可设计性,在对若干条信息链的有序拼接方面具有良好的应用能力,具有较高的拼接效率,与现有的方法相比,拼接的准确性得到了很大的提升;
37、(4)本发明所开发的dna框架结构可以通过外部修饰进行功能化,提供了一种信息库随机访问的方法;
38、(5)本发明中利用细菌进行dna框架结构存储信息的自主复制扩增与读取,大大简化了利用dna进行信息存储后读取的复杂步骤。
本文地址:https://www.jishuxx.com/zhuanli/20240731/185576.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表