一种利用cre-lox系统构建毕赤酵母菌株的方法与流程
- 国知局
- 2024-08-22 14:21:23
本发明涉及微生物基因工程,具体涉及一种利用cre-lox系统构建毕赤酵母菌株的方法。
背景技术:
1、多年前,人类就开始利用酵母来制作发酵产品。如今酵母的应用不仅局限于传统的发酵领域,还涉及到生物医药、生物能源、生物材料等新兴的生物制造领域。利用基因工程和合成生物学等技术,人们可以改造或优化酵母的代谢途径,使其能够合成多种有价值的代谢产物,从而将酵母转化为高效、安全、可控的生物合成平台,即生物工厂。毕赤酵母(pichiapastoris)具有真核生物的亚细胞结构,具有糖基化、脂肪酰化、蛋白磷酸化等翻译后修饰加工功能,且具有高效的蛋白外泌表达能力,是优秀的生物工厂之一。
2、酵母的遗传操作方便。同源重组是一种精确的修复机制,在酿酒酵母(saccharomycescerevisiae)中非常有效。通过供体片段的不同设计可以分别实现基因的插入、删除及替换,是外源基因整合入基因组及基因组定向改造的主要手段。但大多数其他酵母物种如毕赤酵母(p.pastoris)倾向于非同源末端连接修复途径而非同源重组,非同源末端连接过程会随机引入碱基的插入、突变和缺失,其修复效果难以预测和控制。增强同源重组或消除非同源末端连接可能是改善同源整合的前进方向,可以提高编辑效率。研究发现,敲除参与非同源末端连接的关键基因ku70可以有效阻断非同源末端连接的发生,从而显著提高基因组的精准编辑效率。
3、此外,现有的基因敲除技术大多是利用筛选标记基因替换掉待敲除基因这种方法进行目的基因的敲除及敲除菌株的筛选,遗留的筛选标记可能会对酵母的生长造成不利影响。且由于酵母中筛选标记是有限的,不能回收的筛选标记也将会限制酵母接受基因改造的次数。因此,我们迫切需要一种方法来改造毕赤酵母,以提高基因组的精准编辑效率及编辑次数。
技术实现思路
1、本发明的目的在于提供一种利用cre-lox系统构建毕赤酵母菌株的方法,解决以下技术问题:现有的基因敲除技术大多是利用筛选标记基因替换掉待敲除基因这种方法进行目的基因的敲除及敲除菌株的筛选,遗留的筛选标记可能会对酵母的生长造成不利影响。且由于酵母中筛选标记是有限的,不能回收的筛选标记也将会限制酵母接受基因改造的次数。
2、本发明的目的可以通过以下技术方案实现:
3、一种利用cre-lox系统构建毕赤酵母菌株的方法,包括以下步骤:
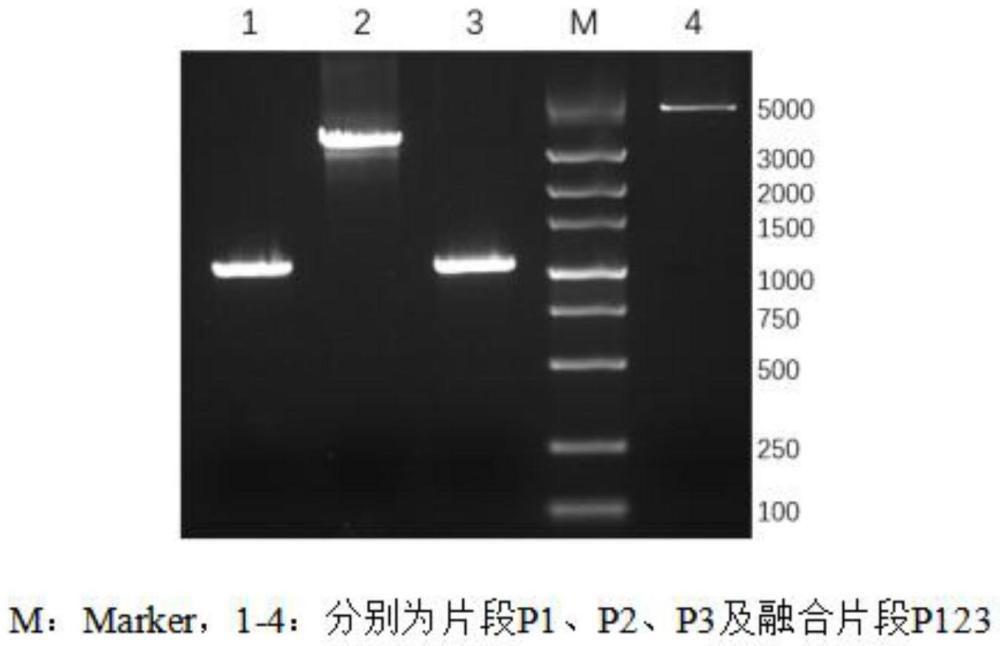
4、s1、从毕赤酵母gs115基因组中分别扩增目的基因ku70的上游1000bp序列p1、下游1000bp序列p3;
5、s2、从质粒ppicza-zeo-cre中扩增zeo抗性及cre重组酶序列p2;
6、s3、通过融合pcr将3片段p1、p2及p3整合到一起,p1及p2的重叠融合部分构成了lox71位点,p2及p3的重叠融合部分构成了lox66位点;
7、s4、将融合片段通过电转化的方法导入毕赤酵母gs115中;
8、s5、通过zeo抗性筛选转化子,pcr验证转化子是否整合到正确的位置;
9、s6、甲醇诱导正确转化子中cre重组酶的瞬时表达,启动lox位点间的重组;
10、s7、表型筛选后通过pcr验证zeo抗性筛选标记及cre重组酶序列是否正确切除。
11、作为本发明进一步的方案:步骤s3中,pcr结束后进行琼脂糖电泳检测,条带大小验证正确后使用gel dna extraction minikit进行产物的纯化回收,回收产物记为p123。
12、作为本发明进一步的方案:步骤s4中具体步骤为:
13、1)取80μl毕赤酵母gs115感受态细胞,加入6μg融合片段p123,混匀后转移到预冷的0.2cm电击杯中;
14、2)冰上放置5min;
15、3)根据对酵母电击参数设置,电击;其中参数设为1.5kv,25uf,200ω;
16、4)立即加入2ml预冷的1msorbitol+hepes;具体为10ml1msorbitol+100ul2mhepes,ph=8.0,转移至2ml无菌离心管中;
17、5)30℃静置孵育1-2h;
18、6)取300μl孵育后菌液涂布于含有zeo抗性的ypd平板上,30℃培养至长出克隆。
19、作为本发明进一步的方案:步骤s5中,采用3对引物验证转化子是否正确整合;其中ku70-f及ku70-r用于验证ku70基因;yz-f1及yz-r2根据融合片段p123之外的染色体dna设计;yz-r1及yz-f2根据融合片段p123内部序列设计;
20、只有当转化子同时具备:ku70-f及ku70-r扩增ku70基因无条带、yz-f1及yz-r1可扩增到正确大小的片段2677bp、yz-f2及yz-r2可扩增到正确大小的片段3230bp这3个条件时,才可说明转化子中融合片段成功替换掉了ku70基因,并且整合到了正确的位置。
21、作为本发明进一步的方案:步骤s6融合片段中,cre重组酶之前的序列中包含aox1启动子,可在甲醇存在的条件下启动下游基因即cre重组酶基因的表达;当cre重组酶存在时,lox71位点和lox66位点之间可以发生重组,从而切除两个位点间的dna序列,达到回收zeo抗性筛选标记及cre重组酶序列的目的;具体操作如下:
22、1)挑取正确的转化子,接种于5m l的ypm液体培养基中,ypm液体培养基为1%酵母提取物、2%蛋白胨和1%甲醇,在30℃,200rpm的条件下过夜培养;
23、2)4000rpm离心5min,弃上清,重悬沉淀涂布于ypd平板上,30℃培养至长出克隆;
24、3)将长出的克隆重悬于无菌水中,调od600=0.02,点板于含有zeo抗性的ypd平板上,30℃培养3天;
25、4)记录无法在含有zeo抗性的ypd平板上生长的克隆编号。
26、作为本发明进一步的方案:步骤s7中,表型筛选是将筛选出的克隆点板至新的含有zeo抗性的ypd平板上,gs115原始菌株以及诱导前的转化子也点板至此板上作为对照。
27、作为本发明进一步的方案:步骤s7中,将表型筛选平板上的所有克隆进行pcr验证,引物、体系以及扩增程序同步骤s5。
28、本发明的有益效果:
29、本发明敲除毕赤酵母中参与非同源末端连接的关键基因ku70,通过阻断非同源末端连接的发生来提高基因组的精准编辑效率。通过诱导cre酶的瞬时表达来回收zeo抗性筛选标记及cre重组酶序列,方便后续继续进行其他基因的编辑。
技术特征:1.一种利用cre-lox系统构建毕赤酵母菌株的方法,其特征在于,包括以下步骤:
2.根据权利要求1所述的一种利用cre-lox系统构建毕赤酵母菌株的方法,其特征在于,步骤s3中,pcr结束后进行琼脂糖电泳检测,条带大小验证正确后使用geldnaextractionminikit进行产物的纯化回收,回收产物记为p123。
3.根据权利要求2所述的一种利用cre-lox系统构建毕赤酵母菌株的方法,其特征在于,步骤s4中具体步骤为:
4.根据权利要求1所述的一种利用cre-lox系统构建毕赤酵母菌株的方法,其特征在于,步骤s5中,采用3对引物验证转化子是否正确整合;其中ku70-f及ku70-r用于验证ku70基因;yz-f1及yz-r2根据融合片段p123之外的染色体dna设计;yz-r1及yz-f2根据融合片段p123内部序列设计;
5.根据权利要求1所述的一种利用cre-lox系统构建毕赤酵母菌株的方法,其特征在于,步骤s6融合片段中,cre重组酶之前的序列中包含aox1启动子,可在甲醇存在的条件下启动下游基因即cre重组酶基因的表达;当cre重组酶存在时,lox71位点和lox66位点之间可以发生重组,从而切除两个位点间的dna序列,达到回收zeo抗性筛选标记及cre重组酶序列的目的;具体操作如下:
6.根据权利要求5所述的一种利用cre-lox系统构建毕赤酵母菌株的方法,其特征在于,步骤s7中,表型筛选是将筛选出的克隆点板至新的含有zeo抗性的ypd平板上,gs115原始菌株以及诱导前的转化子也点板至此板上作为对照。
7.根据权利要求1所述的一种利用cre-lox系统构建毕赤酵母菌株的方法,其特征在于,步骤s7中,将表型筛选平板上的所有克隆进行pcr验证,引物、体系以及扩增程序同步骤s5。
技术总结本发明公开了一种利用cre‑lox系统构建毕赤酵母菌株的方法,涉及微生物基因工程技术领域,具体包括以下步骤:S1、从毕赤酵母GS115基因组中分别扩增目的基因Ku70的上游1000bp序列P1、下游1000bp序列P3;S2、从质粒pPICZA‑Zeo‑Cre中扩增zeo抗性及Cre重组酶序列P2;S3、通过融合PCR将3片段P1、P2及P3整合到一起,本发明敲除毕赤酵母中参与非同源末端连接的关键基因ku70,通过阻断非同源末端连接的发生来提高基因组的精准编辑效率。通过诱导Cre酶的瞬时表达来回收zeo抗性筛选标记及cre重组酶序列,方便后续继续进行其他基因的编辑。技术研发人员:柳曹枝,陈彦羽受保护的技术使用者:南京瑞源生物技术有限公司技术研发日:技术公布日:2024/8/20本文地址:https://www.jishuxx.com/zhuanli/20240822/278299.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。