一种基因组DNA中8-氧代鸟嘌呤的精准定位方法
- 国知局
- 2024-09-05 14:35:28
本发明属于生物,涉及dna氧化损伤在基因组中的定位分析,具体涉及一种基因组dna中8-氧代鸟嘌呤的精准定位分析方法。
背景技术:
1、细胞在新陈代谢和炎症反应的过程中会产生许多活性氧物种(ros),鸟嘌呤相较于其他的碱基氧化还原电位最低,因此最容易被氧化。7,8-二氢-8-氧鸟嘌呤(简称8-氧代鸟嘌呤或og)是一种常见的dna氧化损伤,在dna聚合酶经过时og可能与a配对,进而导致g到t的突变。近年来,许多研究表明鸟嘌呤的氧化在基因表达的调节中起着表观遗传标记的作用,例如在缺氧调节基因中的启动子中og水平升高会导致转录因子的募集,在雌激素调节转录和炎症反应等过程中也观察到了类似的模型。
2、虽然通常在疾病状态下可以检测到dna损伤水平的升高,但是我们对于dna损伤导致疾病发生的具体机制还不清楚。对og在基因组范围内进行精准的定位分析可以帮助我们了解og在不同染色质结构的分布特点以及细胞在受到氧化刺激后og分布状态的变化,从而更好地理解细胞对氧化损伤的应对机制。此外,对og的精准定位分析有助于将og导致的基因突变与癌症相关的突变特征联系起来,还能够帮助阐明og修复过程中产生的ber修复机器与特定基因的转录激活之间的联系。对og生物学意义的深入理解能够进一步帮助我们了解细胞对ros的应答过程,并为疾病的靶向治疗提供新思路。
3、近年来,随着二代测序技术的发展,越来越多的研究工作关注于dna氧化损伤在基因组中的定位情况。由于og在基因组中的丰度很低(1~2og位点/106碱基),因此对含有og位点的dna的富集是og全基因组测序的关键,这也就带来了非特异性富集的问题。对于依赖于抗体识别的oxidip-seq(amente,s.,et al.(2018)genome-wide mapping of 8-oxo-7,8-dihydro-2’-deoxyguanosine reveals accumulation of oxidatively-generateddamage at dna replication origins within transcribed long genes of mammaliancells.nucleic acids research,47,221-236),非特异性抗体结合是固有问题。entrap-seq(fang,y.and zou,p.(2020)genome-wide mapping of oxidative dna damage viaengineering of 8-oxoguanine dna glycosylase.biochemistry,59,85-89)通过突变ogg1将其改造并发挥类似抗体的功能,也面临着糖基化酶与dna非特异性结合的问题。og-seq(ding,y.,et al.(2017)sequencing the mouse genome for the oxidativelymodified base 8-oxo-7,8-dihydroguanine by og-seq.journal of the americanchemical society,139,2569-2572)和claps-seq(an,j.,et al.(2021)genome-wideanalysis of8-oxo-7,8-dihydro-2'-deoxyguanosine at single-nucleotideresolution unveils reduced occurrence of oxidative damage at g-quadruplexsites.nucleic acids research,49,12252-12267)都依赖于对og位点的选择性氧化以及化学探针(胺共轭的生物素)与氧化中间体的共价加合,而og的氧化反应是复杂的,氧化产物可能受到多种因素的影响,而且在基因组dna背景中化学探针很可能发生副反应。cd-seq(jin,s.-g.,et al.(2022)concordance of hydrogen peroxide-induced 8-oxo-guaninepatterns with two cancer mutation signatures of upper gi tract tumors.scienceadvances,8,eabn3815)使用了糖基化酶fpg识别和酶切og,但是fpg除了og还能识别多种dna损伤比如8-氧腺嘌呤、5-羟基胞嘧啶等,对og识别的特异性较差。
4、现有的多数og位点全基因组测序方法的分辨率在200bp左右,只有claps-seq和cd-seq能够做到单碱基的分辨率。但是这两种方法都需要经过多步骤的样品处理过程,对og位点的捕获效率非常有限。对于claps-seq,在用磁珠特异性吸附带有og位点的dna链后,利用高保真聚合酶进行引物延伸,对og位点的精准定位依赖于聚合酶的延伸恰好停止在og位点,但是在基因组dna的背景下影响dna聚合酶的引物延伸过程的因素是多样的,高gc含量的dna序列或者特殊的dna二级结构也会影响dna聚合酶的工作。对于cd-seq,首先需要将基因组dna全部片段化并环化,但是dna片段环化的效率是有限的,没有被环化的dna序列信息会被丢失,用fpg和ape1在氧化损伤位点断开双链环状dna后,还需要从断开位点处进行两个方向的测序,通过对比两套测序数据来确定og位点的位置信息,数据处理的过程也可能导致部分og位点位置信息的丢失。
5、针对目前og位点基因组定位检测方法中存在的缺陷,迫切地需要开发出一种方法,能够对基因组中的og位点分布实现高特异性、高分辨率、高效的定位检测。
技术实现思路
1、本发明的目的是提供一种能够实现对基因组中og位点分布的高特异性、高分辨率、高效的检测方法,以弥补现有分析方法的诸多不足。
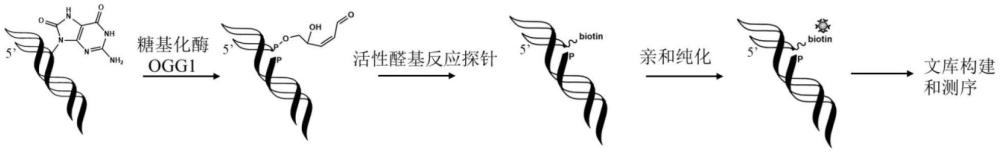
2、本发明的技术方案是,利用8-氧代鸟嘌呤糖基化酶ogg1对og位点的高特异性识别以及特殊的两步酶切性质,直接将og位点转化为dna单链的断裂并且暴露3’磷酸不饱和醛(3’-pua)和5’磷酸(5’-p)末端,随后利用醛基反应探针对3’磷酸不饱和醛中的活性醛基进行高效的化学标记,从基因组dna中特异性富集含有og位点的dna片段,最后通过dna文库的构建和二代测序确定og位点在基因组中的位置,其检测思路如图1所示。
3、具体的,本发明提供的一种基因组dna中8-氧代鸟嘌呤的精准定位方法,包括以下步骤:
4、(1)提取基因组dna并片段化;
5、(2)用醛基反应试剂封闭基因组dna片段中的活性醛基;
6、(3)用ogg1酶切基因组dna片段中的og位点,使含有og位点的dna单链断裂,同时暴露3’-pua和5’-p两种末端;
7、(4)用醛基反应探针标记3’-pua末端中的活性醛基并通过亲和纯化捕获标记后的dna单链片段,没有被标记的5’-p末端的短链和互补链被洗脱下来;
8、(5)针对洗脱下来的5’-p末端的短链构建dna文库和进行二代测序,确定og位点在基因组dna中的位置。
9、在本发明的一些实施方式中,检测流程如图2所示,首先提取基因组dna并片段化,用苄基羟胺封闭基因组dna片段中的活性醛基,然后用ogg1酶切基因组中的og位点,将og位点直接转变为dna单链的断裂,同时暴露3’-pua和5’-p两种末端,之后用含有生物素(biotin)的醛基反应探针标记3’-pua末端中的活性醛基并用链霉亲和素磁珠捕获标记后的dna片段,没有被标记的5’-p末端的短链和互补链会被naoh洗脱下来,我们围绕5’-p末端的短链来构建dna文库,从而将og位点的位置信息标记在dna文库的5’端。
10、上述步骤(1)中,通常采用超声破碎法对基因组dna进行片段化处理,将基因组dna片段化为200-500bp。
11、上述步骤(2)中,所述醛基反应试剂常用的例如o-苄基羟胺(简称苄基羟胺)、o-乙基羟胺等。苄基羟胺封闭基因组dna片段中活性醛基的反应优选以(1h-苯并咪唑-2-亚甲基)胺为催化剂。进一步的,该封闭反应在pbs缓冲液(ph 7.4)中进行,在200μl的反应体系中,基因组dna片段的浓度在10~20ng/μl,苄基羟胺的终浓度为10~20mm,催化剂(1h-苯并咪唑-2-亚甲基)胺的终浓度为10~20mm;室温下反应1~2h,然后分离纯化得到活性醛基被封闭的dna片段。
12、在本发明的一些实施方式中,上述步骤(3)中ogg1酶切反应的缓冲液由20mmtris-hcl ph 7.4、100mm nacl、0.15mg/ml bsa、1mm edta、1mm dtt组成,酶切体系中ogg1的终浓度是0.2~0.4μm,dna片段的浓度在50~100ng/μl,反应体系的体积为40μl,在37℃酶切反应1h后分离纯化得到酶切后的dna片段。
13、上述步骤(4)中,优选的,用含有生物素的醛基反应探针标记3’-pua末端中的活性醛基,然后用链霉亲和素磁珠捕获标记后的dna单链,没有被标记的5’-p末端的短链和互补链被氢氧化钠洗脱下来。所述含有生物素的醛基反应探针例如arp(aldehyde reactiveprobe)、n-pyrrolyl-alanine-edea-biotin等。在本发明的一些实施方式中,标记反应的缓冲液是pbs溶液,反应探针的终浓度是5mm,反应条件是室温过夜。
14、在本发明的一些具体实施方式中,在步骤4)中,用醛基反应探针标记3’-pua末端中的活性醛基后,使dna片段两端连接第一测序接头,再进行亲和纯化;在步骤5)中,洗脱下来的dna连接第二测序接头,然后进行pcr扩增建库和二代测序。其中,所述第一测序接头例如p7接头,所述第二测序接头例如p5接头。
15、上述步骤5)中,对洗脱下来的dna进行纯化、变性处理后,连接第二测序接头,由此在所述5’-p末端的短链的5’端连接第二测序接头,而其3’端连接第一测序接头,通过pcr扩增建库和二代测序后,将测序结果与参考基因序列进行比对,该短链的5’端碱基恰好为og位点后的碱基,从而确定og位点在基因组dna中的位置。
16、本发明提出了基于ogg1酶转化和醛基反应探针化学标记的全基因组og位点单碱基分辨率定位分析方法。本发明具有明显优于现有技术的优点,其主要优点包括:
17、(1)对og位点的定位达到了单碱基分辨率。本发明巧妙利用了ogg1的两步酶切性质,将og位点直接转化为dna单链的断裂,并且利用酶切后暴露的化学基团以具有5’-p末端的短链为基础构建dna文库,进而将og位点的位置信息标记在dna文库的5’端。
18、(2)对og位点富集的特异性高。本发明使用了能够高特异性识别og位点的糖基化酶ogg1,并且通过提前封闭基因组dna中的活性醛基来保证化学探针标记反应的特异性,从而使含有og位点的dna被特异性地从基因组dna中富集出来,确保对og位点定位检测的准确性。
19、(3)定位检测流程简洁高效。本发明利用了糖基化酶ogg1的酶切转化和化学探针来标记og位点,酶切反应和化学标记的反应条件温和,对og位点的富集效率高,此外,在特异性标记og位点的同时通过dna文库构建策略的巧妙设计实现了对og位点位置信息的单碱基分辨率标记,不需要额外的步骤来确定og位点的位置,使得整个定位检测流程简洁高效。
本文地址:https://www.jishuxx.com/zhuanli/20240905/287160.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。