抗生素耐药性在无抗生素环境下可逆性的预测方法及其应用
- 国知局
- 2024-09-19 14:31:26
本发明涉及抗生素耐药性,尤其涉及一种抗生素耐药性在无抗生素环境下可逆性的预测方法及其应用。
背景技术:
1、抗生素显著降低了人类的死亡率和发病率,是人类历史上最重要的生物医学成就之一。然而,抗生素的大量使用使得抗生素耐药性快速传播与进化,每年造成数百万人死亡。为了抵抗抗生素耐药性带来的众多不良影响,目前常用的策略主要有开发新型抗生素和预防耐药性的产生和传播。抗生素的开发往往需要耗费大量的资金和人力,且需要的时间长。耐药性的产生和传播是由抗生素产生的选择压力驱动的进化过程。与预防耐药性出现相反,是否可以通过停用抗生素使得已经存在的耐药性迅速减少,目前仍未有明确的答案。我们猜想,如果抗生素的耐药基因符合(1)具有高适应度代价,即在无抗生素环境中快速淘汰;(2)野生型耐药基因的近端突变体中,无适应度代价的抗性突变体非常罕见,这两个条件,则停用该种抗生素可以使得该耐药基因耐药性迅速减少。
2、粘菌素及其耐药基因mcr-1(mobilized colistin resistance-1)是用于评估停用抗生素对其耐药性影响的潜在模型。粘菌素是一种广谱抗革兰氏阴性菌的抗生素,是临床的最后一线用药,对粘菌素的耐药性常常意味着没有其他更有效的抗生素可以用于治疗。mcr-1基因是粘菌素耐药性的最主要来源,也是第一个被发现存在于质粒上(即可以通过水平转移传播)的粘菌素耐药基因,它对粘菌素的耐药水平居于中等水平。在革兰氏阴性菌中,mcr-1编码的酶可以将磷酸乙胺(petn)转移到脂质a上,脂质a是脂多糖(lps)的脂质成分,转移到脂质a上的petn中和了lps携带的负电荷,阻止了lps与粘菌素阳离子多肽的静电相互作用,从而降低了粘菌素与细菌结合的亲和力,赋予细菌耐药性。目前只有34种mcr-1突变体被分离和记录,意味着mcr-1的进化还处在较早的时期,它具有很大的潜力进化出针对粘菌素的更强的耐药突变体。同时,其他证据表明,mcr-1基因除了具有粘菌素抗性以外,还带有相当大的适应度代价。更重要的是,有报道指出,自我国禁止在动物饲料中添加粘菌素以来,mcr-1的流行率迅速下降。因此,可以通过研究mcr-1突变体的特点,验证我们的猜想,即抗生素耐药性在无抗生素环境下的可逆性的预测方法与判断流程。
技术实现思路
1、本发明所要解决的技术问题在于,提供一种抗生素耐药性在无抗生素环境下可逆性的预测方法,通过耐药基因等位基因文库准确预测其在无抗生素环境下的耐药性可逆性。
2、本发明还要解决的技术问题在于,提供一种上述预测方法在筛选耐药性可以被逆转或耐药性可以通过限用加以控制的抗生素耐药基因中的应用。
3、本发明还要解决的技术问题在于,提供一种上述预测方法在推算限制抗生素使用后可以长期存在的低适应度代价的抗性突变中的应用。
4、本发明还要解决的技术问题在于,提供一种上述预测方法在预测菌株流行率下降所需时间与抗性菌株的适应度的关系中的应用。
5、为了解决上述问题,本发明公开了一种抗生素耐药性在无抗生素环境下可逆性的预测方法,包括以下步骤:
6、(1)构建耐药基因的等位基因文库,所述等位基因文库包含野生型耐药基因和耐药基因突变体;
7、(2)将所述等位基因文库与无功能对照菌混合后在有和/无抗生素环境下进行竞争性培养,计算野生型耐药基因和耐药基因突变体相对于无功能对照菌的相对生长速率;
8、根据相对生长速率将耐药基因突变体进行分型,所述分型包括在无抗生素环境下分为无适应度代价、适应度代价下降或适应度代价增加,在有抗生素环境下分为没有抗性、抗性降低或抗性增强;
9、(4)若耐药基因具有高适应度代价,且无适应度代价的抗性耐药基因突变体在其近端突变体中罕见,则判定该耐药基因在无抗生素环境下具有可逆性。
10、作为上述技术方案的改进,步骤(1)中所述等位基因文库的建立方法如下:
11、(1.1)引入单点突变,合成带有突变的耐药基因片段;
12、(1.2)以带有突变的耐药基因片段作为上游引物,带有随机碱基序列条形码的基因片段作为下游引物,进行pcr扩增,并连接到耐药质粒上;
13、(1.3)将连接好的耐药质粒转入大肠杆菌并筛选,收集阳性克隆作为耐药基因的等位基因文库,对等位基因文库中的质粒进行提取并冷冻保存。
14、作为上述技术方案的改进,步骤(2)中野生型耐药基因和耐药基因突变体相对于无功能对照菌的相对生长速率的计算方法如下:
15、(2.1)对冷冻保存的质粒进行取样,记录t0时间点不同基因型的频数,然后将取样的质粒分别转入不同浓度的抗生素环境下进行竞争性培养,抗生素的浓度为0μg/ml~4μg/ml,分别在不同时间点进行样本采集;
16、(2.2)将不同时间点采集的样本进行质粒dna的提取,进行第一轮pcr,扩增出质粒dna上的条形码片段;纯化后进行第二轮pcr,以平均测序深度125读长/条形码~135读长/条形码进行测序,记录t时间点不同基因型的频数;
17、(2.3)将同一基因型的所有条形码的频数合计为该基因型的频数,根据下式计算相对生长速率:
18、
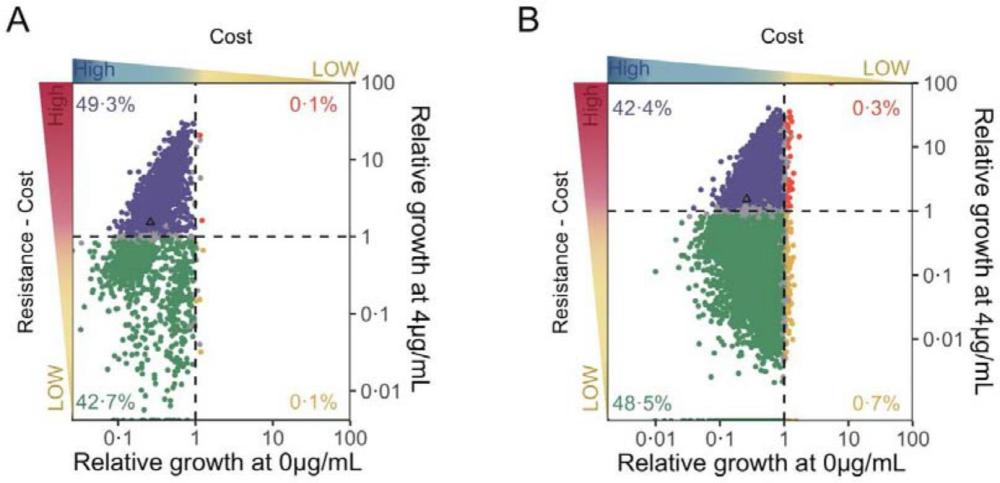
19、其中,ft为t时间点基因型的频数,f0为t0时间点基因型的频数。
20、作为上述技术方案的改进,步骤(2)中还包括对基因型进行过滤,过滤标准如下:
21、在t0时间点,该基因型的所有条形码的频数>100。
22、作为上述技术方案的改进,步骤(2)中,所述等位基因文库与无功能对照菌的混合比例为(40~60):1。
23、作为上述技术方案的改进,步骤(3)的分组方法如下:
24、在无抗生素环境下,若耐药基因突变体的相对生长速率≥1,则该耐药基因突变体无适应度代价;若耐药基因突变体的相对生长速率>野生型耐药基因的相对生长速率,则该耐药基因突变体适应度代价下降;若耐药基因突变体的相对生长速率<野生型耐药基因的相对生长速率,则该耐药基因突变体适应度代价增加;
25、在抗生素环境下,若耐药基因突变体的相对生长速率<1,则该耐药基因突变体没有抗性;若耐药基因突变体的相对生长速率>野生型耐药基因的相对生长速率,则该耐药基因突变体抗性增强;若耐药基因突变体的相对生长速率≥1,且耐药基因突变体的相对生长速率<野生型耐药基因的相对生长速率,则该耐药基因突变体抗性降低。
26、作为上述技术方案的改进,步骤(4)包括以下步骤:以无抗生素环境下的相对生长速率为横坐标,以抗生素环境下的相对生长速率为纵坐标,以无功能对照菌的相对生长速率为分界线,将不同耐药基因突变体进行分类,分类包括无适应度代价的抗性突变体、有适应度代价的抗性突变体、有适应度代价的非抗性突变体、无适应度代价的非抗性突变体,并计算每个类别的突变体占比;其中有适应度代价包括适应度代价下降和适应度代价增加,抗性包括抗性降低和抗性增强;
27、若有适应度代价的突变体的占比≥90%且无适应度代价的抗性突变体的占比≤1%,则认为有较强证据支持该耐药基因在无抗生素环境下具有可逆性。
28、相应的,本发明还公开了上述的预测方法在粘菌素耐药性预测中的应用,所述耐药基因为mcr-1;
29、根据适应度代价构建群体遗传学模型,预测菌株流行率下降所需时间与抗性菌株的适应度的关系,关系如下:
30、
31、其中,m为抗性菌株相对于敏感菌株的适应度,f0为耐药基因的初始频率,p为t时刻的频率与初始频率的比值。
32、相应的,本发明还公开了一种上述的预测方法在筛选耐药性可以被逆转或耐药性可以通过限用加以控制的抗生素耐药基因中的应用。
33、相应的,本发明还公开了一种上述的预测方法在推算限制抗生素使用后可以长期存在的低适应度代价的抗性突变中的应用。
34、实施本发明,具有如下有益效果:
35、1、本发明通过研究mcr-1突变体在有/无粘菌素环境中的相对生长速率得到mcr-1的适应度代价变化和抗性变化,mcr-1具有高适应度代价,且无适应度代价的抗性mcr-1突变体在其近端突变体中罕见,因此可以利用耐药基因的适应度代价变化和抗性变化预测抗生素耐药性在无抗生素环境下具有可逆性。
36、2、本发明的等位基因文库涵盖98.9%所有可能的单点mcr-1突变体,相同基因型的不同条形码之间的一致性高,不同生物学重复样本间的相关性高,预测方法的可重复性好。
37、3、本发明构建了群体遗传学模型,推导出抗性菌株的适应度与菌株流行率下降到原先一半时所需要的时间之间的关系,并应用这个模型验证现实世界中耐药基因适应度代价与耐药基因流行率下降之间的关系,本发明构建的群体遗传学模型推导的mcr-1耐药基因的适应度与真实数据的mcr-1耐药基因的适应度吻合度高。
38、4、本发明的预测方法可以应用到筛选耐药性可以被逆转或耐药性可以通过限用加以控制的抗生素耐药基因中,若耐药基因具有高适应度代价,且无适应度代价的抗性耐药基因突变体在其近端突变体中罕见,则该耐药基因在无抗生素环境下具有可逆性,通过停用该抗生素,可以控制或逆转耐药性,从而提高该抗生素的药物作用。
39、5、本发明的预测方法可以应用到推算限制抗生素使用后可以长期存在的低适应度代价的抗性突变中,通过对此类低适应度代价的抗性突变进行研究,分析进化路径,更好地了解此类突变在细菌群体中的传播特点。
本文地址:https://www.jishuxx.com/zhuanli/20240919/298617.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。