一种基于多层级要素响应的氮磷营养物基准制定方法与流程
- 国知局
- 2024-09-19 14:38:29
本发明属于营养物基准制定领域,尤其涉及一种基于多层级要素响应的氮磷营养物基准制定方法。
背景技术:
1、营养物基准(nutrient criteria)是指对水体功能或用途不产生不良或有害影响的营养物最大浓度或水平。氮磷是表征和评价水体富营养化的重要指标。
2、营养物基准制定的主要技术方法包括统计分析法和压力—响应模型法,前者一般适用于人类干扰较少的区域,是对连续观测的数据进行统计分析后,按照设定的频数区间计算基准值;后者适用于人类活动干扰严重的区域,通过研究环境压力因子与响应因子之间的响应关系确定基准值,又分为非参数拐点法、分类回归树法等,压力因子一般为氮磷等,响应因子一般为叶绿素a、溶解氧、透明度等单因子。现行营养基准制定方法学中,重要问题之一是响应因子均为初层级单一因子指标,对生态系统的代表性有限,无法真正揭示生态系统对氮磷等环境压力的响应关系,据此制定的营养物基准值是否真正能够控制富营养化值得商榷。
技术实现思路
1、为解决上述技术问题,本发明提供了一种基于多层级要素响应的氮磷营养物基准制定方法,包括:
2、基于高通量测序技术获取生物群落大数据,并对所述生物群落大数据进行生物类群跨域的大数据分析;
3、以氮磷输入下的多层级要素响应因子为响应变量,表征氮磷输入后生态系统的响应关系,推导获得氮磷营养物的基准值。
4、优选地,基于高通量测序技术获取生物群落大数据的过程包括:
5、设置采样点进行水体样品采集并获得环境dna滤膜样品,对所述水体样品进行水体理化指标测定,并提取所述滤膜样品中的环境dna;
6、针对不同类群水生生物的宏条形码基因设计通用扩增引物,对水生生物宏条形码基因进行扩增;
7、电泳检测所述滤膜样品的目标条形码基因扩增情况,切出目标条带使用核酸纯化柱对样品进行纯化,将纯化后的样品进行高通量测序获得生物群落大数据;所述生物群落大数据包括但不限于生物的种类、操作分类单元、相对丰度。
8、优选地,设置采样点进行水体样品采集并获得环境dna滤膜样品的过程包括:
9、设置有利于观察生物群落、生态系统对氮磷营养物浓度有效响应的采样点位;其中,所述采样点位采集的水样包括用于水体理化检测的第一水样和用于环境dna提取的第二水样;
10、通过硝酸纤维滤膜对所述第二水样进行过滤,将过滤后的硝酸纤维膜保存在-80℃,获得所述环境dna滤膜样品。
11、优选地,对所述水体样品进行水体理化指标测定的过程包括:
12、测定所述第一水样中硝酸盐、亚硝酸盐、叶绿素a的含量,并在采样现场对所述第一水样中的do、水温、氧化还原电位、浊度、电导率进行检测。
13、优选地,提取所述滤膜样品中的环境dna的过程包括:
14、基于dna提取试剂盒进行基因组dna提取,获得硝酸纤维膜上水生生物的环境dna,并用1%琼脂糖凝胶电泳检测dna的完整性,同时利用nanodrop和qubit检测dna的浓度和纯度。
15、优选地,对水生生物宏条形码基因进行扩增的过程包括:
16、针对浮游植物,目标条形码基因为线粒体基因18s的v9区,引物如下:
17、正向引物:tccctgcchtttgtacacac,seq id no:1;
18、反向引物:ccttcygcaggttcacctac,seq id no:2;
19、针对浮游动物,目标条形码基因为线粒体基因18s的v4区,引物如下:
20、正向引物:agggcaakyctggtgccagc,seq id no:3;
21、反向引物:grcggtatctratcgyctt,seq id no:4;
22、针对鱼类,目标条形码基因为线粒体基因12s,引物如下:
23、正向引物:gtcggtaaaactcgtgccagc,seq id no:5;
24、反向引物:catagtggggtatctaatcccagtttg,seq id no:6;
25、针对底栖无脊椎动物,目标条形码基因为coi,引物如下:
26、正向引物:ggdacwggwtgaacwgtwtaycchcc,seq id no:7;
27、反向引物:caaacaaatardggtattcgdty,seq id no:8;
28、反应体系组成包括:15μl的high-fidelity pcr mastermix(newengland biolabs)+0.2μm的引物+10ng模板环境dna;
29、扩增流程包括:98℃变性1分钟+30个扩增循环+72℃延伸5分钟;
30、其中,每个循环反应参数为:98℃变性10s,50℃复性30s,72℃延伸30s。
31、优选地,电泳检测所述滤膜样品的目标条形码基因扩增情况,切出目标条带使用核酸纯化柱对样品进行纯化,将纯化后的样品进行高通量测序的过程包括:
32、切胶纯化目的条带至dna占比≥50%,将扩增子上机测序,进行混库、加测序接头、纯化测序文库,文库经检验合格后通过双端或单端测序方法进行序列测定;
33、将测序得到的原始图像数据文件经碱基识别分析转化为原始测序序列,并将所述原始测序序列以fasto文件格式存储;所述原始测序序列包括测序序列的序列信息以及对应的测序质量信息。
34、优选地,对所述生物群落大数据进行生物类群跨域的大数据分析的过程包括:
35、对高通量测序的下机数据进行清洗质控后,通过序列聚类获得otu序列集;
36、通过数据库比对对所述otu序列集进行物种注释,获得浮游生物类群信息;所述浮游生物类群信息包括但不限于每个采样点位的浮游生物物种的种类和otu相对丰度;
37、计算每个点位硅甲藻比、浮游植物香浓-维纳多样性指数与浮游生物香浓-维纳多样性指数。
38、优选地,对高通量测序的下机数据进行清洗质控后,通过序列聚类获得otu序列集,通过数据库比对对所述otu序列集进行物种注释的过程包括:
39、对高通量测序的下机数据进行清洗,去除接头,过滤低质量测序序列,获得清洗后的数据;
40、对过滤后的双端测序序列进行拼接,并根据引物在两端的比对结果去除引物序列,以及测序错误超过1%的序列;
41、过滤后将所有样本的拼接序列进行合并,通过otu聚类或扩增序列变异降噪的方法计算代表序列,得到otu序列集;
42、将所述otu序列集与目标基因宏条码数据库的参考序列进行biast比对,过滤掉比对长度低于90%且相似度低于80%的序列,根据相似度98%,95%,90%,85%的阈值依次获得种,属,科,目,纲五个分类水平的注释;
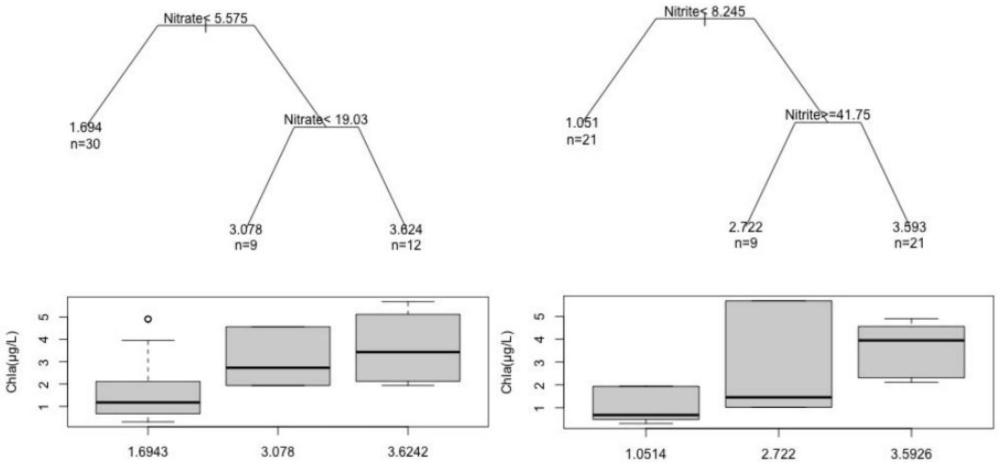
43、基于otu表的物种注释,对不同样本的多样性,群落结构,优势物种,入侵物种进行统计分析;
44、所述香浓-维纳多样性指数的计算表达式为:
45、
46、
47、其中,s是类群数目,pi是频度,ni是第i个类群的个体数,n是所有类群的总个体数。
48、优选地,获得氮磷营养物的基准值的过程包括:
49、构建压力—响应模型,所述压力-响应模型法包括分类回归树模型、非参数拐点分析和贝叶斯拐点分析;
50、分别以采样点位的硝酸盐浓度、亚硝酸盐浓度为压力变量,叶绿素浓度、otu丰度、硅甲藻比、浮游植物香浓-维纳多样性指数与浮游生物香浓-维纳多样性指数为响应变量,输入所述分类回归树模型,通过停止、剪枝过程,选择最优树,将所述最优树的节点所对应的水体营养物浓度作为营养物基准候选值;
51、基于所述营养物基准候选值进行营养物基准推导,首先使用分类回归树模型中的根节点得到首个压力指标基准值,然后通过子节点得到后续压力指标基准值;当分类回归树模型中所有节点均为首个污染压力指标时,采用非参数拐点分析获得后续压力指标基准值;当所有变量指标数据均符合正态分布时,采用贝叶斯拐点分析得到最终的营养物基准值。
52、与现有技术相比,本发明具有如下优点和技术效果:
53、(1)本发明克服了传统压力—响应法基于单一因子建立响应关系的局限,建立包括生物群落特征与水体理化等信息的多层级要素响应关系,实现以生态系统健康为指向,便于制定更加科学完善的营养物基准。
54、(2)本发明使用了环境dna技术,具有环境友好性,属于非破坏性采样,并且克服了传统形态学鉴定成本高、耗时耗力、结果不精准的问题,便于获得生物群落大数据,更有利于获得确定性高的营养物基准值;
55、(3)本发明技术流程易于标准化,便于不同区域研究的对比分析和研究。
本文地址:https://www.jishuxx.com/zhuanli/20240919/299321.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表