一种检测水稻单细胞基因序列变异的方法及其应用
- 国知局
- 2024-10-09 15:29:27
本发明属于农业,尤其涉及一种检测水稻单细胞基因序列变异的方法及其应用。
背景技术:
1、诱变育种是作物中一种常见的育种途径,在创制生物安全性高、可直接用于种质选育的优异新基因方面具有较大优势。传统诱变育种对植物诱变材料的研究水平多集中在组织、器官水平,这种传统的测序思路只能反映细胞的平均数据,不能提供单个细胞之间的异质性信息,不利于进行细胞水平精细化、针对性的变异挖掘和利用,进而可能忽略单细胞中特有的位点变异。
2、随着基因组测序技术和生物信息学的飞速发展,对变异机理的研究起到了极大的促进作用。全基因组重测序在植物诱变育种领域应用最为广泛,极大地降低某些未知功能的重要基因序列的鉴定障碍,使诱变基因资源得到充分的挖掘和利用,为相关研究提供技术支撑。单细胞全基因组测序技术(single-cell whole-genome sequencing,scwgs)在单细胞分辨率下揭示了生物样本中的基因组异质性,弥补传统测序方法的不足,帮助我们理解植物发育过程中细胞的独特性功能。
3、相对动物细胞而言,单细胞测序在植物中的研究应用较少。植物细胞壁的存在给单细胞分离与获取增加了难度,由于目前只能通过分离原生质体的方法对单细胞进行测序,这给部分原生质体分离存在困难的物种材料造成了较大影响,并且可能导致测序结果产生偏差,也对植物单细胞测序的普及造成一定困难,因此单细胞测序仅在玉米等少数作物中得到应用。
4、水稻作为最重要的粮食作物之一,通过诱变育种创制新的变异基因和种质资源对保障我国乃至世界粮食安全具有重要意义。通过创新水稻小孢子性细胞等单细胞的分离技术、水稻单细胞全基因组测序技术和单细胞基因序列变异检测,对于深入挖掘和利用诱变处理的水稻单细胞基因序列变异位点,具有重要意义。
技术实现思路
1、为解决上述技术难题,本发明提供了一种检测水稻单细胞基因序列变异的方法及其应用。本发明中的方法主要包括取样、测序和生信分析三方面,其中取样获取的是单个水稻小孢子性细胞。本发明成功分离出单个水稻小孢子性细胞,并对已有水稻单细胞变异检测技术流程所存在的完整性不强、检测连贯性有待提升等缺陷进行了完善,此外还通过重离子诱变技术,鉴定确认了变异位点的真实性。本发明中的方法拓宽了水稻单细胞诱变材料的来源范围,为创制更多样的育种材料、进行更高效的诱变育种提供了新的思路和方法。同时小孢子性细胞作为雄配子,基因组携带的突变能够通过杂交进入子代,可实现变异的遗传和固定,也为优异变异基因的快速利用提供了途径。
2、为达上述目的,本发明采用如下的技术方案:
3、本发明提供了一种检测水稻单细胞基因序列变异的方法,包括以下步骤:
4、(1)单细胞分离和dna扩增:
5、先通过拉尖的玻璃毛细管连接微量注射器制作的简易单细胞分离装置,吸取单个存活的单核早期小孢子,然后基于mda phi29 dna聚合酶等温扩增体系,使用单细胞全基因组扩增试剂盒进行单细胞dna扩增;
6、(2)全基因组测序和数据过滤:
7、先把基因组dna打成dna片段进行平末端修复,然后在dna片段两端连接da尾,并连接测序接头,随后通过磁珠进行纯化,再选择300-400bp范围的片段进行pcr扩增、纯化、质检以及上机测序,最后使用软件fastp过滤;
8、(3)单细胞基因组的真实变异鉴定:
9、使用软件gatk进行变异检测,按照单倍型的方法获取snp位点,让软件根据测序reads的情况直接判断该位点的碱基类型,输出该位点碱基类型;取测序深度100×时,杂合率为0.4-0.6的位点作为杂合突变位点;在此范围外的位点,校正为对应的高频碱基的纯合位点;在此过程中还要去除因为基因组背景差异和dna扩增过程错配两方面原因造成的假阳性数据,以及使用重离子或γ射线诱变技术对小孢子基因序列变异进行验证;
10、(4)单细胞基因组变异的偏好性分析:
11、从单细胞测序结果中筛选高频突变基因(hmg)、低频突变基因(lmg)和无突变基因(nmg),在hmg中,snp的数量和基因的长度具有极显著的正相关关系,为了排除基因的长度对snp数量的影响,以snp比例作为筛选指标,snp比例为单位长度(bp)的snp数量;
12、(5)单细胞基因组变异的生物信息学分析:
13、包括a.go富集分析和b.数据分析和绘图。
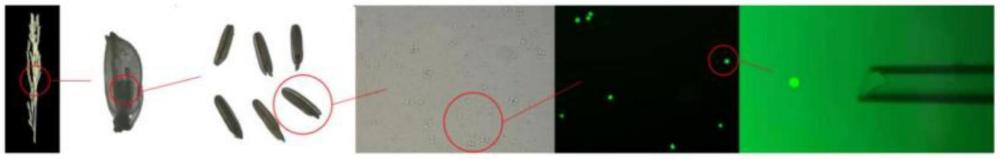
14、优选的,所述单细胞的分离方法具体为:先通过fda-pi对小孢子进行染色,然后在20倍物镜的荧光显微镜下观察细胞活性,最后通过拉尖的玻璃毛细管连接微量注射器制作的简易单细胞分离装置,吸取单个存活的单核早期小孢子。
15、优选的,所述单细胞的dna扩增方法具体为:在单细胞样品中加入3μl buffer d2,轻弹管壁混匀并短暂离心收集;65℃孵育10分钟;加入3μl buffer n,轻弹管壁混匀并短暂离心;将40μl的反应混合液加入到准备好的10μl dna样品中,轻弹管壁混匀并短暂离心收集,30℃孵育3-4小时;65℃孵育5分钟失活discover-sc dna polymerase;将扩增好的dna样品进行1%的琼脂糖凝胶电泳检测,主条带清晰明亮的dna样品放入-80℃保存备用。
16、优选的,所述通过磁珠进行纯化时具体使用的磁珠为ampure xp磁珠。
17、优选的,所述因为基因组背景差异造成的假阳性数据去除方法为:若测序品种和gatk比对使用的参考基因组品种不一致,则材料本身的基因组背景有差异,设置10个空白对照样品,统计空白对照样品的测序的变异位点,对比到辐照样品中,相同的位点即认为是来源于基因组背景的差异,进行筛除。
18、优选的,所述因为dna扩增过程错配造成的假阳性数据去除方法为:高保真性phi29 dna聚合酶在进行整个水稻基因组的多次的循环复制,每个样品仍会产生成千上万的错配位点,但在循环扩增的过程中错配发生得越晚,被检测到的杂合率就越低,取第一次就发生错配的极限值,将杂合率低于25%的位点进行校正,改为对应的高频碱基的基因型,筛除扩增过程中产生的假阳性。
19、优选的,所述使用重离子或γ射线诱变技术对小孢子基因序列变异进行验证时,所使用的重离子为3-5gy的重离子,γ射线为9-11gy的γ射线。
20、进一步优选的,所使用的重离子为4gy的重离子,γ射线为10gy的γ射线。
21、优选的,所述go富集分析的方法为:将目标基因向go数据库的各term映射,并计算每个term的基因数,从而得到具有某个go功能的基因列表及基因数目统计;使用超几何检验,找出与整个基因组背景相比,在目标基因中显著富集的go term;将目标基因集与go数据库关联,对目标基因进行go富集分析,获取基因显著富集的go term;所述数据分析和绘图的方法为:生物信息相关数据的整理和分析使用的是microsoft excel 2016和spss19.0,使用origin 2022进行图形的绘制。
22、本发明还提供了所述方法在水稻育种中的应用。
23、本发明的有益效果是:
24、针对现有技术中的缺陷和不足:(1)细胞是生命体最基本的结构单位,传统的测序方法只能反映细胞的平均数据,不能提供单个细胞之间的异质性信息;单细胞测序技术可以弥补传统测序方法的不足,但目前在作物中的研究应用较少,在水稻中,单细胞测序的取样、测序和数据分析的方法有待完善。植物细胞壁的存在给单细胞分离与获取增加了难度,不同物种、组织及发育阶段的细胞壁厚度不同,且原生质体的分离过程会导致细胞状态的变化,但目前只能通过分离原生质体的方法对单细胞进行测序。这给部分原生质体分离存在困难的物种材料造成了较大影响,并且可能导致测序结果产生偏差,也对植物单细胞测序的普及造成一定困难。(2)水稻性细胞在单细胞水平的取样、测序和数据分析全流程操作步骤不够完善。
25、本发明的创新之处主要体现在:本发明基于新兴发展的单细胞测序技术,以单核早期小孢子为材料,完善了水稻性细胞的取样、测序和数据分析流程,首次建立了较为完善的水稻小孢子性细胞在单细胞水平的变异检测体系,并进行了实例验证。本发明技术指标清晰、可操作性强、可进行批量化变异检测,为高效批量进行水稻性细胞突变体的变异检测提供技术体系支撑。
26、具体体现在以下几个方面:
27、(1)材料来源方面,目前水稻上进行研究的诱变材料多集中在组织、器官层级,难以进行单细胞水平上精细变异的研究。
28、本发明成功分离出单个水稻小孢子性细胞,进行重离子诱变,鉴定确认了变异位点的真实性。该发明拓宽了水稻单细胞诱变材料的来源范围,为创制更多样的育种材料、进行更高效的诱变育种提供了新的思路和方法。同时小孢子性细胞作为雄配子,基因组携带的突变能够通过杂交进入子代,实现变异的遗传和固定,为优异变异基因的快速利用提供途径。
29、(2)检测流程方面,已有的水稻的单细胞变异检测技术流程的完整性不强,检测的连贯性有待提升。
30、本发明完善了相关检测流程,主要包括三方面的流程:取样、测序和生信分析:
31、a.取样方面的创新之处主要在于单细胞分离和dna扩增技术的创新。本发明利用简易单细胞分离装置,吸取单个存活的单核早期小孢子,并确认了利用基于mda的phi29dna聚合酶等温扩增体系对单细胞dna进行扩增,符合后续单细胞测序的质量要求。
32、b.测序方面利用磁珠法进行dna纯化,针对性选择300-400bp范围的片段进行pcr扩增,提高了测序质量,并能够顺利获取小孢子单细胞水平的基因组,为后续变异做好基础数据准备。
33、c.分析方面步骤的创新主要是真实变异位点的鉴定。确认小孢子单细胞在重离子诱变后发生了基因组水平上可检测的真实性突变,意义重大,只有鉴定确认能够诱发真实变异,本发明的诱发小孢子基因组变异的方法,才可确认为有效。
34、此外,变异位点检测和真实变异鉴定,在技术分析上明确了假阳性数据来自“基因组背景差异”和“dna扩增错配引入的假阳性变异位点”这两个方面,并进行针对性的滤除步骤,有效保证了数据的真实性,为后续进行基因功能分析奠定基础。
本文地址:https://www.jishuxx.com/zhuanli/20241009/308916.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。