一种基于高通量测序的土壤微生物种群绝对定量方法
- 国知局
- 2024-08-22 14:45:08
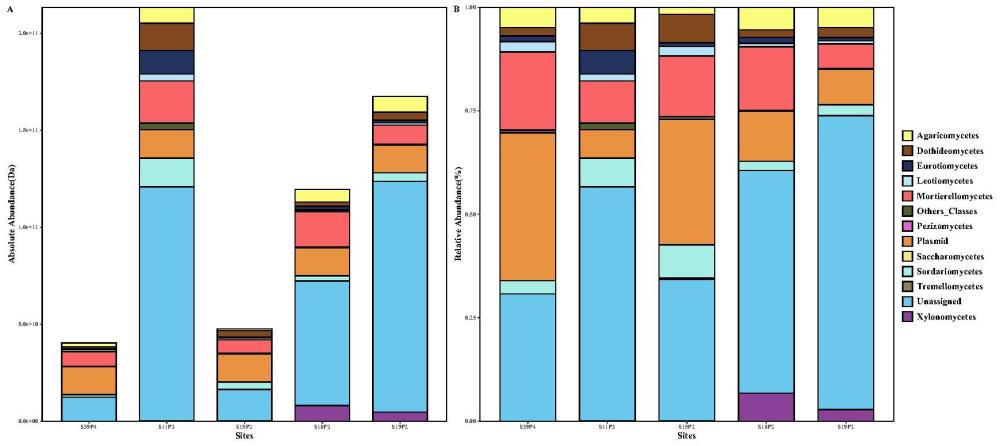
本发明涉及微生物信息学和生物,尤其涉及一种基于高通量测序的微生物种群绝对定量分析方法。
背景技术:
1、微生物群落影响人类和动物的疾病和免疫、地球化学养分循环和生态系统功能与过程。不同领域的研究揭示了不同微生物群之间的微妙关系,现在常用的研究方法是利用pcr(聚合酶链反应)扩增微生物的保守区域后,进行高通量测序以揭示不同的扩增子家族(即衍生自特定pcr引物对的otu,例如细菌16s、真核18s或真菌its(internal transcribedspacer))进行比较,从而得到各类微生物的相对丰度,但该方法忽略了微生物绝对丰度的变化。为了揭示肠道、土壤和其他环境的真正复杂性,微生物群落的决定定量分析能够为相关研究提供更为全面充分的信息。
2、扩增子测序是对特定长度的pcr产物或者捕获的片段进行测序,目前主要是应用高通量测序技术对特定环境中特定基因片段进行测序。大多数天然微生物都不能通过传统的分离培养方法进行分离和克隆,这对于天然微生物的定性和定量分析以及微生物多样性探索构成严峻的挑战。扩增子测序可以克服传统分离培养的弱点,对样品中的微生物进行定性和定量分析,目前在微生物多样性检测中得到了广泛应用。
3、16s和18s rdna是细菌和真菌中16s或18s rdna基因的高变区,而its则是细菌、真菌和古细菌中小亚基和大亚基rrna基因之间的间隔区dna。这些特定的遗传物质都包括进化保守性及进化可变区。由于pcr扩增容易,可以将目标片段从少量dna中扩增出来,而基因序列即使在近缘种间也有高度的变异,因而扩增子测序在分类学和分子系统学中得到了广泛的应用。
4、本发明使用合成的嵌合dna作为内标用以对不同扩增子家族进行绝对定量。具体方法如下:合成的嵌合dna直接添加到环境样品中;提取环境样本的基因组dna;pcr扩增真菌its序列、细菌16s序列和原核生物18s序列,进行高通量测序;高通量测序结果计算扩增子家族的相对丰度和绝对丰度(例如每单位质量样品的原核16s、真核18s和真菌its)。
5、本发明以高度复杂的环境样品作为研究对象证明了该方法能够灵敏准确地鉴定群体的绝对丰度。本方法适应同时检测多种微生物群落(包括细菌、真菌和原核生物),包括来自土壤的环境样品,来自人类肠道的样品以及来自食物的样品。本发明方法操作简单、检测准确、应用广泛,在微生物群落的鉴定分析中具有广阔的市场前景。
技术实现思路
1、本发明基于高通量测序技术分析微生物群落绝对丰度的方法及应用,属于微生物扩增子检测技术领域。
2、本发明的目的是针对现有技术中的不足,提供一种基于扩增子测序的微生物绝对定量的分析方法。
3、为实现上述目的,本发明采取的技术方案是:
4、s1、设计合成嵌合dna。设计包含的要素有:(1)分别来自用于鉴定某些类或某些种微生物的常见基因的引物结合位点(pbss);(2)优化的合成填充物序列,其长度和gc含量与体内目标相同;(3)易于获得、易于处理的合成dna来源。在每种情况下,gc含量均设计为与其环境基因对应物相似,其序列使用随机dna生成器设计(https://www.faculty.ucr.edu)。通过基因合成公司合成了嵌合dna,并将其克隆到质粒中作为嵌合内标质粒。
5、s2、称取固定量土壤样品中加入定量已知浓度的嵌合内标质粒。
6、s3、提取将s2中混合好的样品的土壤微生物基因组dna。使用土壤dna提取试剂盒对样品进行处理。
7、s4、pcr扩增s3中提取到的土壤微生物基因组dna,用特定的引物进行pcr扩增,得到相应的扩增子。汇集最终的pcr产物,并用pcr纯化试剂盒进行纯化。
8、s5、将s4中纯化得到的pcr产物进行高通量测序。将样品送至公司,通过illuminamiseq 300pe进行测序。测序数据使用定制的linux和python脚本进行处理,并辅以fastx-toolkit的脚本(http://www.hannonlab.cshl.edu)。
9、s6、分析高通量测序得到的otus结果,分析土壤微生物群落的相对定量和绝对定量。
10、本发明使用合成的嵌合dna作为内标用来分析不同扩增子家族的绝对定量。包括以下步骤:合成的嵌合dna直接添加到环境样品中;提取环境样本的基因组dna;pcr扩增真菌its序列、细菌16s序列和原核生物18s序列,进行高通量测序;高通量测序结果通过在数据库中比对,通过得到的分类单元获得微生物群落的相对丰度;根据内标嵌合质粒计算微生物群落的绝对丰度。本项目以高度复杂的环境样品作为研究对象证明了该方法能够灵敏准确地鉴定特定群体的绝对丰度。本方法适应任何扩增子特异性组,包括来自土壤的环境样品,来自人类肠道的样品以及来自食物样品的样品。
技术特征:1.一种基于高通量测序的微生物种群绝对定量的分析方法,其主要步骤包括:
2.如权利要求1所述的基于高通量测序的微生物种群绝对定量的方法,其特征在于,所述设计的人工合成序列中有三个关键因素:(1)引物结合位点(pbss),分别来自用于鉴定原核生物(p)、细菌(b)、真菌(f)等微生物的常见基因;(2)优化的合成填充物序列,其长度和gc含量与体内目标相同;(3)容易获得、易于处理的合成dna来源。
3.如权利要求1所述的基于高通量测序的微生物种群绝对定量的方法,其特征在于,所述的嵌合内参质粒与土壤样品的混合比例不能相差太大,容易引起实验误差。
4.如权利要求1所述的基于高通量测序的微生物种群绝对定量的方法,其特征在于,所述的pcr扩增中的引物为鉴定物种的特异性引物,是嵌合内标质粒中的序列。
技术总结本发明开发了一种基于高通量测序技术分析微生物群落绝对丰度的方法并予以应用,属于微生物扩增子检测技术领域。本发明使用合成的嵌合DNA作为内标以同时对样品中的微生物群落(包括细菌、真菌和原生生物)进行绝对定量。具体方法如下:合成的嵌合DNA直接添加到环境样品中;提取环境样本的基因组DNA;PCR扩增真菌ITS序列、细菌16S序列和原核生物18S序列,进行高通量测序;高通量测序结果计算扩增子家族的相对丰度和绝对丰度(例如每单位质量样品的原核16S、真核18S和真菌ITS)。本发明以高度复杂的环境样品作为研究对象证明了该方法能够灵敏准确地量化特定群体的绝对丰度。本方法广泛适用于不同来源样品的微生物群落分析,包括来自土壤的环境样品、来自植物组织的样品、来自人类肠道的样品以及来自食物样品的样品。本发明方法操作简单、检测准确、应用广泛,在微生物群落的鉴定分析中具有广阔的市场前景。技术研发人员:张凤,刘向,张馨尹受保护的技术使用者:兰州大学技术研发日:技术公布日:2024/8/20本文地址:https://www.jishuxx.com/zhuanli/20240822/279703.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表