一组可应用于中华绒螯蟹地理种群快速鉴定的SNP标记的制作方法
- 国知局
- 2024-09-11 14:58:01
本发明属于群体遗传学领域,具体是涉及一组可应用于中华绒螯蟹地理种群快速鉴定的snp标记。
背景技术:
1、中华绒螯蟹(eriocheir sinensis)是重要的甲壳类淡水养殖经济物种。
2、近年来,由于养殖区域过于集中、种质混杂以及盲目引种等问题的存在,许多养殖群体内部的遗传多样性较低,经过多代的人工繁育和养殖,导致种质退化和养殖性能下降,引种的问题也随之愈发重要,这也给精准的种质鉴定和遗传结构解析工作提出了新的要求。由于种群间基因交流现象的存在,其不同地理种群之间的遗传分化较小,亲缘关系较近,这也进一步给主要流域的种群划分带来了困难。此外,底栖甲壳类种群划分对于资源量的判定、补充群体数量的估计以及渔业资源研究和管理政策的制定都有着重要的意义。
技术实现思路
1、本发明的目的在于针对现有技术存在的养殖群体内部的遗传多样性较低,因种群间基因交流,不同地理种群间的遗传分化较小,使得种群划分困难等问题,提供一组可应用于中华绒螯蟹地理种群快速鉴定的snp标记,应用于中华绒螯蟹的野生资源研究及育种产业。
2、为了实现上述目的,本发明提供一组可应用于中华绒螯蟹地理种群快速鉴定的snp标记,组成可应用于中华绒螯蟹地理种群快速鉴定的低密度snp分子标记数据集,包含如表1所示的3825个snp标记;所述snp标记与中华绒螯蟹不同地理群体的遗传分化具有较强关联性。
3、表1本发明提供的3825个snp标记信息
4、
5、
6、
7、
8、
9、
10、
11、
12、
13、
14、
15、
16、
17、
18、
19、
20、注:chr_info一栏的格式为“染色体_物理位置_参考位点基因型_变异位点基因型”。
21、本发明提供所述3825个snp标记可在中华绒螯蟹地理种群鉴定工作中的应用。
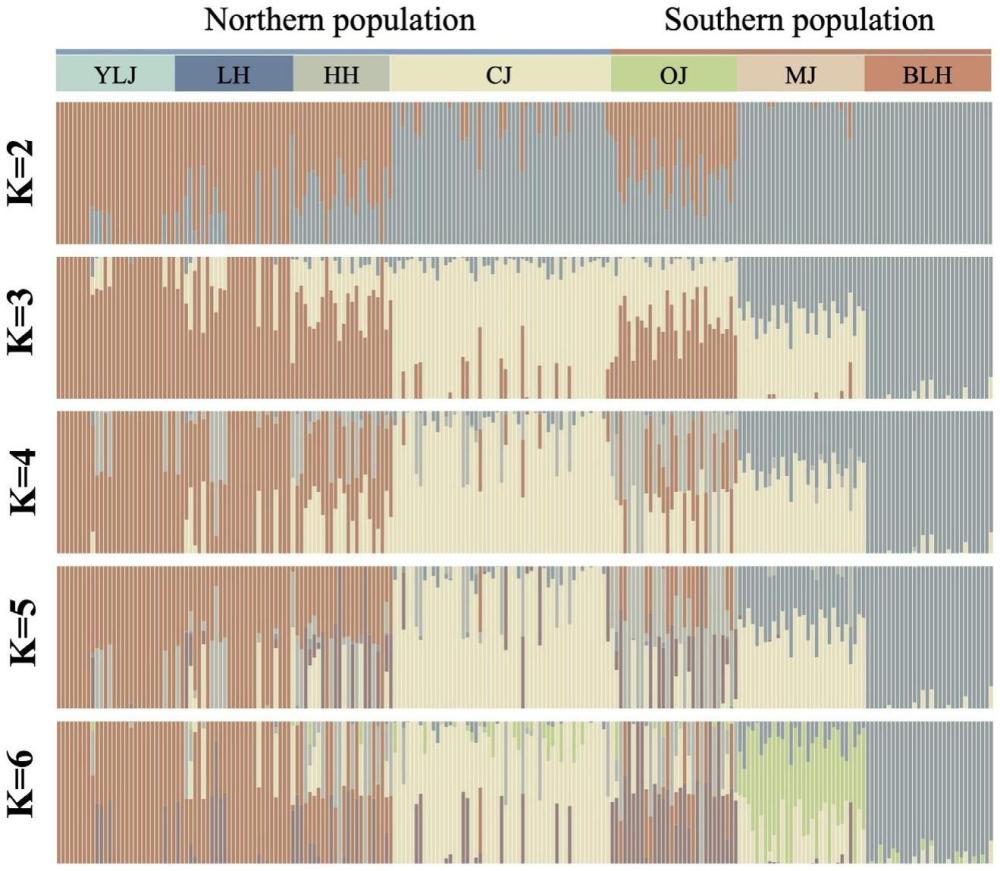
22、所述应用的具体方法可为:从待鉴定的中华绒螯蟹个体中提取高质量的基因组dna;根据snp标记信息,对3825个snp标记进行基因分型;将待鉴定中华绒螯蟹个体的基因型信息和多流域野生中华绒螯蟹个体的群体基因型文件(base_genotyping.vcf)进行整合,根据进化树和遗传成分结构的可视化结果对中华绒螯蟹个体的地理群体来源进行鉴定;
23、具体的,一种中华绒螯蟹地理种群鉴定方法,包括以下步骤:
24、1)提取待鉴定的中华绒螯蟹个体的基因组dna,使用琼脂糖凝胶电泳检测dna的质量;质量合格的个体基因组dna用于步骤2)中基因分型测序分析;
25、2)采取测序技术对如权利要求1所述3825个snp标记进行基因分型,获取待鉴定中华绒螯蟹个体的snp基因型,所述测序技术包括简化基因组测序、全基因组重测序、基因分型阵列、多重pcr;
26、3)根据步骤2)的snp标记分型结果,至少保证所述3825个snp标记具有90%以上分型成功率,结合3825个snp标记信息文件及多流域野生中华绒螯蟹个体的群体基因型文件(base_genotyping.vcf)进行整合,使用开源软件fasttree和admixture对整合后的文件进行遗传结构评估,并用r-studio和itol工具对结果进行可视化,实现对待鉴定中华绒螯蟹个体的地理群体来源鉴定。
27、fasttree:http://www.microbesonline.org/fasttree/;
28、admixtur:http://dalexander.github.io/admixture/index.html;
29、r-studio:www.rstudio.com/products/rstudio/;
30、itol:https://itol.embl.de/。
31、所述多流域野生中华绒螯蟹个体的群体基因型文件(base_genotyping.vcf)可通过以下方式获得:采集一定数量的多流域野生中华绒螯蟹样本经基因组dna提取后,进行全基因组重测序,与最新版本的中华绒螯蟹基因组(bioproject:prjna737102)对比,用gatk进行全基因组尺度的变异位点挖掘,用vcftools和plink对原始数据进行质量控制和过滤,得到全基因组数据vcf文件;进一步对全基因组数据vcf文件使用开源软件bcftools以3825个snp位点的坐标信息为参考进行位点过滤,得到多流域个体的3825个位点的变异信息,命名为多流域野生中华绒螯蟹个体的群体基因型文件(base_genotyping.vcf)。
32、bcftools:https://samtools.github.io/bcftools/。
33、所述多流域野生中华绒螯蟹样本可采集自南北方代表性流域,每个流域至少采集30个样本。
34、利用本发明提供的3825个分子标记数据集,可将其应用于中华绒螯蟹的野生资源研究及育种产业中,以实现对地理种群的快速且精准鉴定。借由上述提供的标记信息和应用流程,本发明至少具有下列优点及有益效果:
35、1)本发明利用群体间分歧度检验(fixation index,fst)筛选到一组(3825个)可使地理群体模型预测准确性达到最佳的snp分子标记集合;并公开其在中华绒螯蟹最新版本基因组(bioproject:prjna737102)上的物理位置(图1和表1)。
36、2)利用本发明提供的3825个标记信息,可将其应用于中华绒螯蟹的野生资源研究及育种产业中,以实现对地理种群的快速且精准的划分,为种质鉴定工作提供高效的策略。
37、3)本发明提供的3825个标记信息,与国内代表性流域中华绒螯蟹地理群体分化具有较强的相关性和代表性,在种质鉴定工作中,鉴于其数量较少,能够极大的减少基因分型成本和时间成本(约5~10min),具有广阔的应用前景和商业价值。
技术特征:1.一组可应用于中华绒螯蟹地理种群快速鉴定的snp标记,其特征在于包含如下所示的3825个snp标记:
2.如权利要求1所述一组可应用于中华绒螯蟹地理种群快速鉴定的snp标记在中华绒螯蟹地理种群鉴定中的应用。
3.一种中华绒螯蟹地理种群鉴定方法,其特征在于采用如权利要求1所述一组可应用于中华绒螯蟹地理种群快速鉴定的snp标记,具体步骤为:从待鉴定的中华绒螯蟹个体中提取高质量的基因组dna;根据snp标记信息,对3825个snp标记进行基因分型;将待鉴定中华绒螯蟹个体的基因型信息和多流域野生中华绒螯蟹个体的群体基因型文件进行整合,根据进化树和遗传成分结构的可视化结果对中华绒螯蟹个体的地理群体来源进行鉴定。
4.如权利要求3所述一种中华绒螯蟹地理种群鉴定方法,其特征在于包括以下步骤:
5.如权利要求3或4所述一种中华绒螯蟹地理种群鉴定方法,其特征在于所述多流域野生中华绒螯蟹个体的群体基因型文件通过以下方式获得:采集一定数量的多流域野生中华绒螯蟹样本经基因组dna提取后,进行全基因组重测序,与中华绒螯蟹基因组bioproject:prjna737102对比,用gatk进行全基因组尺度的变异位点挖掘,用vcftools和plink对原始数据进行质量控制和过滤,得到全基因组数据vcf文件;对全基因组数据vcf文件使用开源软件bcftools以3825个snp位点的坐标信息为参考进行位点过滤,得到多流域个体的3825个位点的变异信息vcf文件,命名为多流域野生中华绒螯蟹个体的群体基因型文件。
技术总结一组可应用于中华绒螯蟹地理种群快速鉴定的SNP标记,属于群体遗传学领域。基于国内从南到北代表性流域240个野生个体的全基因组遗传变异信息,利用群体间分歧度检验筛选到一组可使地理群体模型预测准确性达到最佳的SNP分子标记集合,公开其在中华绒螯蟹最新版本基因组上的物理位置BioProject:PRJNA737102。利用这组SNP标记在对采集到的野生个体进行群体分型,与利用全基因组遗传信息的策略相比,表现出较高预测准确性,且极大的减少基因分型成本和计算时间。可快速将其应用到野生资源研究及育种产业中,短时间之内就可准确的鉴定其地理种群,为种质鉴定工作提供高效的策略,具有广阔的应用前景和商业价值。技术研发人员:白玉麟,徐东坡,梁翼东,马俊蕾,张婷,王安琪受保护的技术使用者:中国水产科学研究院淡水渔业研究中心技术研发日:技术公布日:2024/9/9本文地址:https://www.jishuxx.com/zhuanli/20240911/292701.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。