一种用于狼尾草属牧草遗传多样性分析的SRAP分子标记引物及其遗传多样性分析方法
- 国知局
- 2024-10-15 10:05:02
本发明属于分子生物学dna标记技术与应用领域,具体涉及一种用于狼尾草属牧草遗传多样性分析的srap分子标记引物及其在狼尾草属牧草遗传多样性分析中的应用以及狼尾草属牧草遗传多样性分析的方法。
背景技术:
1、相关序列扩增多态性(sequence-related amplified polymorphism,srap)是一种新型的基于pcr的标记系统,为显性标记,是由美国加州大学蔬菜系li与quiros博士于2001年在芸薹属作物开发出来。该标记具有简便、高效、产率高、高共显性、重复性好、易测序、便于克隆目标片段的特点。目前已成功地应用于作物遗传多样性分析、遗传图谱的构建、重要性状的标记以及相关基因的克隆等方面。
2、狼尾草属牧草作为世界重要牧草资源之一,主要包括象草、狼尾草、以及各种种间杂交品种(杂交狼尾草和杂交象草)等,狼尾草属牧草在世界各地广泛种植,并且育成的新品种也日渐增多,从而使得品种间发生了多样的遗传分化。这种情况下,传统的分类法已经很难识别这些品种,近年来研究者们也开始利用分子标记技术对狼尾草属牧草进行遗传多样性和亲缘关系分析。但目前为止未见到srap方法对狼尾草属牧草品种进行遗传多样性分析的研究报道。
3、本研究以20个狼尾草属牧草品种为对象,利用合成的srap分子标记引物,进行srap研究其遗传多样性。以供狼尾草属牧草种质资源保护利用和新品种(系)的选育提供分子水平的参考。
技术实现思路
1、本发明的目的在于提供一种能够用于狼尾草属牧草遗传多样性分析的srap分子标记引物体系及其狼尾草属牧草遗传多样性分析的方法。
2、本发明的目的通过如下技术方案实现:
3、一种用于狼尾草属牧草遗传多样性分析的srap分子标记引物,它包括8对引物,8对引物分别为:
4、me1-em5,其核苷酸序列如seq id no.1和seq id no.25所示;
5、me2-em3,其核苷酸序列如seq id no.2和seq id no.23所示;
6、me3-em5,其核苷酸序列如seq id no.3和seq id no.25所示;
7、me4-em3,其核苷酸序列如seq id no.4和seq id no.23所示;
8、me5-em2,其核苷酸序列如seq id no.5和seq id no.22所示;
9、me6-em7,其核苷酸序列如seq id no.6和seq id no.27所示;
10、me7-em2,其核苷酸序列如seq id no.2和seq id no.22所示;
11、me8-em2,其核苷酸序列如seq id no.8和seq id no.22所示。
12、本发明合成了多条srap引物并随机分别选出20条正向引物和20条反向引物(引物序列见表2),并进行优化,两两组合成400对引物组合,随机抽取4个提取的dna模板样品进行引物组合筛选,最后筛选出上述8对引物。
13、本发明提供一种利用所述的srap分子标记引物进行狼尾草属牧草遗传多样性分析的方法,采用所述的srap分子标记引物对待检测的狼尾草属牧草样品dna进行扩增、检测和srap。具体包括以下步骤:
14、(1)采用ctab法提取待检测样品的dna备用;
15、(2)利用权利要求1所述的srap分子标记引物对步骤(1)中提取的dna样品进行扩增,分别得到pcr扩增产物;
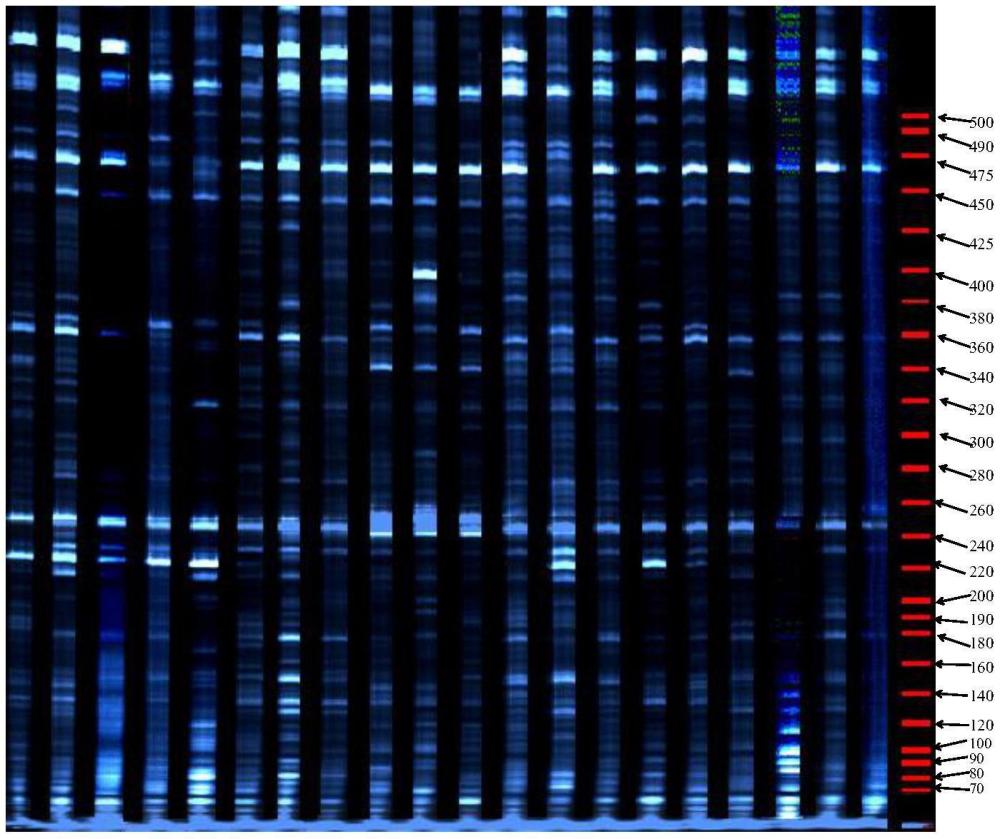
16、(3)将所述步骤(2)得到的pcr扩增产物分别进行凝胶电泳、成像后,得到对应的谱带信息;
17、(4)根据所述步骤(3)得到的谱带信息对样品进行聚类分析,生成聚类图。
18、步骤(2)中所述的pcr扩增的反应体系具体为:2.5μl 10xpcr buffer、0.5μldntps、0.5μl taq酶、引物各1μl和2μl模板dna,ddh2o补充到总体积25μl;
19、所述的引物为权利要求1中的所述me1-em5、me2-em3、me3-em5、me4-em3、me5-em2、me6-em7、me7-em2、me8-em2中的任意一对。
20、所述pcr扩增的程序为:pcr程序:94℃预变性5min;94℃变性1min、35℃退火1min、72℃延伸1min,5个循环;94℃变性1min、50℃退火1min、72℃延伸1min,35个循环;循环结束后72℃延伸10min,4℃保存。扩增反应在美国伯乐pcr仪上进行。
21、所述引物可合成采用荧光引物,所有扩增产物用荧光检测。荧光引物的合成以及荧光检测属于本领域常规的检测方法,在此不作赘述。
22、步骤(4)的具体方法为:
23、由自动测序仪377扫描得到的样品凝胶图,用genescan 3.1软件分析样品凝胶图,通过binthere软件提取样品的各片段大小的结果;excel转换将表内的数值不为0转换为1(即电泳图谱上同一位置有条带出现的记为1,同一位置没有条带出现的记为0),数值为0的不转换,从而生成由“1”和“0”组成的原始矩阵;用ntsyspc-2.11f软件进行数据分析;对原始矩阵用simqual程序求dice相似系数矩阵,并获得相似系数矩阵;用shan程序中的upgma方法进行聚类分析,并通过tree plot模块生成聚类图。
24、所述的srap分子标记引物在狼尾草属牧草遗传多样性分析中的应用。
25、较之现有技术而言,本发明的优点在于:本发明合成了多条srap引物并随机分别选出20条正向引物和20条反向引物,并进行优化,两两组合成400对引物组合,随机抽取4个提取的dna模板样品进行引物组合筛选,最后筛选出8对srap分子标记引物。这8对srap分子标记引物扩增条带多、多态性丰富、亮度大、重复性好,可用于狼尾草属牧草品种的遗传多样性分析。这8对srap分子标记引物对20个狼尾草属品种的pcr扩增,共得到1278个位点,其中多态性位点有1273个,占总数的99.6%。如引物组合me1-em5扩增得到的多态性位点最多达216个,引物组合me3-em5扩增到的多态性位点最少有131个。平均位点数为159.8个,平均多态性位点数为159.1个。可以看出选取的这8对srap引物组合在狼尾草属牧草上多态性极其丰富,可以进行遗传多样性分析。
技术特征:1.一种用于狼尾草属牧草遗传多样性分析的srap分子标记引物,其特征在于:包括8对引物,8对引物分别为:
2.一种利用权利要求1所述的srap分子标记引物进行狼尾草属牧草遗传多样性分析的方法,其特征在于:采用权利要求1所述的srap分子标记引物对待检测的狼尾草属牧草样品dna进行扩增、检测和分析。
3.根据权利要求2所述的方法,其特征在于:它包括以下步骤:
4.根据权利要求3所述的方法,其特征在于:步骤(2)中所述的pcr扩增的反应体系具体为:2.5μl 10xpcr buffer、0.5μl dntps、0.5μl taq酶、引物各1μl和1μl模板dna,ddh2o补充到总体积25μl;
5.根据权利要求4所述的方法,其特征在于:所述pcr扩增的程序为:pcr程序:94℃预变性5min;94℃变性1min、35℃退火1min、72℃延伸1min,5个循环;94℃变性1min、50℃退火1min、72℃延伸1min,35个循环;循环结束后72℃延伸10min,4℃保存。
6.根据权利要求3所述的方法,其特征在于:步骤(4)的具体方法为:
7.如权利要求1所述的srap分子标记引物在狼尾草属牧草遗传多样性分析中的应用。
8.一种分析狼尾草属牧草遗传多样性的试剂盒,其特征在于:它包括权利要求1所述的用于狼尾草属牧草遗传多样性分析的srap分子标记引物。
技术总结本发明涉及一种用于狼尾草属牧草遗传多样性分析的SRAP分子标记引物及其在狼尾草属牧草遗传多样性分析中的应用以及狼尾草属牧草遗传多样性分析的方法;所述SRAP分子标记引物,它包括8对引物,8对引物分别为:ME1‑EM5,ME2‑EM3,ME3‑EM5,ME4‑EM3,ME5‑EM2,ME6‑EM7,ME7‑EM2,ME8‑EM2,本发明筛选出的这8对SRAP分子标记引物扩增条带多、多态性丰富、亮度大、重复性好,可用于狼尾草属牧草品种的遗传多样性分析。这8对SRAP分子标记引物对20个狼尾草属品种的PCR扩增,共得到1278个位点,其中多态性位点有1273个,占总数的99.6%。如引物组合ME1‑EM5扩增得到的多态性位点最多达216个,引物组合ME3‑EM5扩增到的多态性位点最少有131个。平均位点数为159.8个,平均多态性位点数为159.1个。可以看出选取的这8对SRAP引物组合在狼尾草属牧草上多态性极其丰富,可以进行遗传多样性分析。技术研发人员:张晓佩,李文杨,刘远,高承芳受保护的技术使用者:福建省农业科学院畜牧兽医研究所技术研发日:技术公布日:2024/10/10本文地址:https://www.jishuxx.com/zhuanli/20241015/316376.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
下一篇
返回列表