用于改变植物确定性的组合物和方法与流程
- 国知局
- 2024-06-20 11:00:51
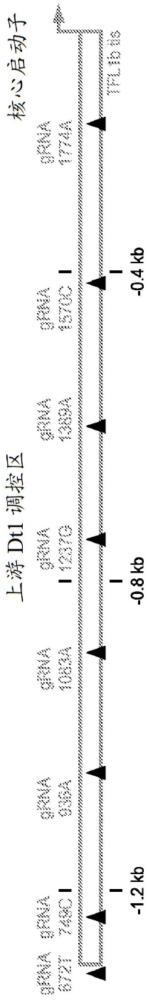
本公开涉及农业生物,并且更具体地涉及用于植物中的基因组编辑的方法和组合物。
背景技术:
1、精确的基因组编辑技术是工程改造基因表达和功能的强大工具,并且有可能改善重要的农业性状。本领域一直需要开发新的组合物和方法来有效并高效地编辑各种作物的植物基因组,以改变确定性(determinacy)、增加产量、减少倒伏并获得其他有益结果。
技术实现思路
1、本文提供了经修饰的植物、植物种子、植物部分或植物细胞,其包含修饰,与在其他方面相同但缺乏所述修饰的植物、植物种子、植物部分或植物细胞中的tfl1或其同源物的表达或活性相比,所述修饰降低tfl1或其同源物的表达或活性。在一些实施方案中,所述修饰存在于内源性tfl1基因或其同源物的至少一个等位基因中。所述tfl1基因可以是tfl1b基因。在特定实施方案中,所述tfl1基因或其同源物编码与seq id no:2具有至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%、至少99.5%或100%序列同一性的蛋白质。在其他实施方案中,所述修饰位于tfl1基因或其同源物的非编码区中,其非限制性实例包括启动子、内含子、5’-非翻译区、3’-非翻译区以及它们的任何组合。本文提供的植物可以是或植物种子、植物部分或植物细胞可来自例如豆科植物、棉花植物、油菜植物、玉米植物、高粱植物、水稻植物、小麦植物、大麦植物、番茄植物或胡椒植物,或其他作物、观赏植物或其他类型的植物物种。在进一步的实施方案中,豆科植物是大豆植物、豆类植物、豌豆植物、鹰嘴豆植物、苜蓿植物、花生植物、角豆植物、扁豆植物或甘草植物。在具体实施方案中,豆科植物是大豆植物。在一些实施方案中,植物、植物种子、植物部分或植物细胞对于修饰是杂合的,而在其他实施方案中,植物、植物种子、植物部分或植物细胞对于修饰是纯合的。在某些实施方案中,植物、植物种子、植物部分或植物细胞被定义为包含tfl1基因的第一等位基因中的第一修饰和tfl1基因的第二等位基因中的第二修饰,第一修饰和第二修饰彼此不同。
2、在某些实施方案中,本文提供的经修饰的植物、植物种子、植物部分或植物细胞可包含降低tfl1或其同源物的表达或活性的修饰,其中所述修饰包含缺失、插入、取代、倒位或它们的任何组合。在一些实施方案中,例如,修饰位于距选自由seq id no:4、67-77、79和81组成的组的序列的3’末端约100个、125个、150个、175个、200个、225个、250个、275个或300个或更多个核苷酸处。在其他实施方案中,tfl1b基因的至少一个等位基因的启动子中的修饰包含在参考序列seq id no:4的核苷酸位置1237与1570之间的基因组区域内。在一些实施方案中,所述修饰包含至少约10个、至少约15个、至少约20个、至少约25个、至少约30个、至少约35个、至少约40个、至少约45个、至少约50个、至少约55个、至少约60个、至少约65个、至少约70个、至少约75个、至少约80个、至少约85个、至少约90个、至少约95个、至少约100个、至少约125个或至少约150个连续核苷酸的缺失。植物、植物种子、植物部分或植物细胞还可包含例如tfl1b基因的启动子的至少一个等位基因中的修饰,其中所述修饰选自由以下组成的组:与参考序列seq id no:4相比,从核苷酸1539至核苷酸1568的30个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1217至核苷酸1604的388个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1518至核苷酸1629的112个碱基对缺失;与参考序列seqid no:4相比,从核苷酸951至核苷酸1222的272个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1364至核苷酸1407的44个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1367至核苷酸1516的150个碱基对缺失;与参考序列seq id no:4相比,从核苷酸754至核苷酸1806的1053个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1216至核苷酸1319的104个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1159至核苷酸1965的807个碱基对缺失;与参考序列seq id no:4相比,从核苷酸760至核苷酸1214的455个碱基对缺失;与参考序列seq id no:4相比,从核苷酸939至核苷酸1028的90个碱基对缺失;37个碱基对倒位,其中seq id no:4的核苷酸1029至核苷酸1065的序列已经缺失、倒位并在同一位置处被重新插入;与参考序列seq id no:4相比,从核苷酸1066至核苷酸1664的599个碱基对缺失;与参考序列seq id no:4相比,从核苷酸952至核苷酸1552的601个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1677至核苷酸1808的132个碱基对缺失;与参考序列seqid no:4相比,从核苷酸1524至核苷酸1558的35个碱基对缺失;与参考序列seq id no:4相比,从核苷酸476至核苷酸1405的930个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1365至核苷酸1559的195个碱基对缺失;与参考序列seq id no:4相比,从核苷酸928至核苷酸1560的633个碱基对缺失;与参考序列seq id no:4相比,从核苷酸593至核苷酸1813的1221个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1552至核苷酸1556的5个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1537至核苷酸1565的29个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1209至核苷酸1246的38个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1552至核苷酸1600的49个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1553至核苷酸1601的49个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1368至核苷酸1378的11个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1368至核苷酸1395的28个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1344至核苷酸1384的41个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1219至核苷酸1453的235个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1215至核苷酸1227的13个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1370至核苷酸1376的7个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1220至核苷酸1556的337个碱基对缺失;与参考序列seq idno:4相比,从核苷酸1216至核苷酸1376的161个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1368至核苷酸1384的17个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1366至核苷酸1648的283个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1370至核苷酸1489的120个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1541至核苷酸1615的75个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1367至核苷酸1375的9个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1551至核苷酸1601的51个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1371至核苷酸1377的7个碱基对缺失;与参考序列seqid no:4相比,从核苷酸1368至核苷酸1553的186个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1365至核苷酸1378的14个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1365至核苷酸1380的16个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1552至核苷酸1594的43个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1367至核苷酸1376的10个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1552至核苷酸1682的131个碱基对缺失;与参考序列seq id no:4相比,从核苷酸954至核苷酸1391的438个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1535至核苷酸1560的26个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1534至核苷酸1644的111个碱基对缺失;与参考序列seq idno:4相比,从核苷酸937至核苷酸1599的663个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1551至核苷酸1648的98个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1526至核苷酸1559的34个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1528至核苷酸1606的79个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1542至核苷酸1602的61个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1214至核苷酸1594的381个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1368至核苷酸1554的187个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1369至核苷酸1477的109个碱基对缺失;与参考序列seqid no:4相比,从核苷酸1550至核苷酸1554的5个碱基对缺失;与参考序列seq id no:4相比,从核苷酸734至核苷酸2000的1267个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1371至核苷酸1560的190个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1537至核苷酸1572的36个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1541至核苷酸1586的46个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1552至核苷酸1556的5个碱基对缺失;与参考序列seq id no:4相比,从核苷酸669至核苷酸1623的955个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1521至核苷酸1558的38个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1369至核苷酸1477的109个碱基对缺失;与参考序列seq idno:4相比,从核苷酸1540至核苷酸1554的15个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1217至核苷酸1674的458个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1546至核苷酸1626的81个碱基对缺失;与参考序列seq id no:4相比,从核苷酸1547至核苷酸1635的89个碱基对缺失;以及它们的任何组合。在一些实施方案中,植物、植物种子、植物部分或植物细胞包含选自由seq id no:30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123、124、125、126、127和128组成的组的多核苷酸序列本文提供的植物、植物种子、植物部分或植物细胞还可包含例如tfl1b基因中的染色体序列,所述染色体序列在缺失、插入、取代或倒位之外的区域中与seq id no:4具有至少70%、至少75%、至少80%、至少85%、至少90%、至少95%、至少96%、至少97%、至少98%、至少99%、至少99.5%或100%序列同一性。
3、本文还提供了经修饰的植物或其种子、植物部分、细胞,其包含修饰,相对于在其他方面相同但缺乏所述修饰的植物而言,所述修饰改变植物的确定性表型。在某些实施方案中,与在其他方面相同但缺乏修饰修饰的植物的确定性相比,所述修饰增加植物的确定性。在一些实施方案中,与在其他方面相同但缺乏修饰的植物相比,经修饰的植物更早到达其终花期,表现出降低的倒伏率,表现出基本相同或增加的产量,或表现出较低的对真菌疾病的易感性,或它们的任何可能的组合。
4、在某些实施方案中,提供了一种多核苷酸,其包含选自由seq id no:30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123、124、125、126、127和128组成的组的序列。还提供了一种指导rna,其包含选自由seq id no:13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28和29组成的组的多核苷酸序列。
5、本文进一步公开了一种用于生产包含经修饰的tfl1基因的植物的方法,所述方法包括:a)将修饰引入植物细胞的内源性tfl1基因或其同源物的至少一个靶位点中;b)鉴定并选择步骤(a)中包含所述tfl1基因或其同源物中的所述修饰的一个或多个植物细胞;和c)从步骤(b)中选择的至少一个或多个细胞再生至少一种植物。在一些实施方案中,所述靶位点位于内源性tfl1基因或其同源物的非编码区中。在其他实施方案中,所述非编码区选自由以下组成的组:启动子、内含子、5’-非翻译区、3’-非翻译区以及它们的任何组合。在进一步的实施方案中,所述非编码区是启动子。在更进一步的实施方案中,通过在所述植物细胞中存在至少一种位点特异性基因组修饰酶来促进修饰。这种酶的非限制性实例包括rna指导的核酸酶、锌指核酸酶、大范围核酸酶、tale核酸酶、重组酶、转座酶以及它们的任何组合。rna指导的核酸酶的实例包括cas核酸酶、cpf1核酸酶或其变体。根据本公开可以找到用途的一些位点特异性基因组修饰酶在靶位点处产生至少一个链断裂。本文公开的方法可用于例如产生根据本公开的任何修饰,包括取代、插入、倒位、缺失、重复以及它们的组合。在一些实施方案中,所述修饰是缺失,并且所述缺失包含至少10个、至少15个、至少20个、至少25个、至少30个、至少35个、至少40个、至少45个、至少50个、至少55个、至少60个、至少65个、至少70个、至少75个、至少80个、至少85个、至少90个、至少95个、至少100个、至少125个或至少150个连续核苷酸的区域。
6、本文提供的方法可用于例如生产具有所需表型的植物。这种表型的非限制性实例包括更早的终花期、降低的倒伏率、基本上相同或增加的产量、较低得对真菌疾病的易感性和增加的确定性,或它们的任何可能的组合。在具体实施方案中,当与在其他方面相同但缺乏根据本公开的赋予这种表型的修饰的植物相比,这种表型可被定义为存在于植物中。
本文地址:https://www.jishuxx.com/zhuanli/20240619/896.html
版权声明:本文内容由互联网用户自发贡献,该文观点仅代表作者本人。本站仅提供信息存储空间服务,不拥有所有权,不承担相关法律责任。如发现本站有涉嫌抄袭侵权/违法违规的内容, 请发送邮件至 YYfuon@163.com 举报,一经查实,本站将立刻删除。
上一篇
CD73化合物的制作方法
下一篇
返回列表